
介绍
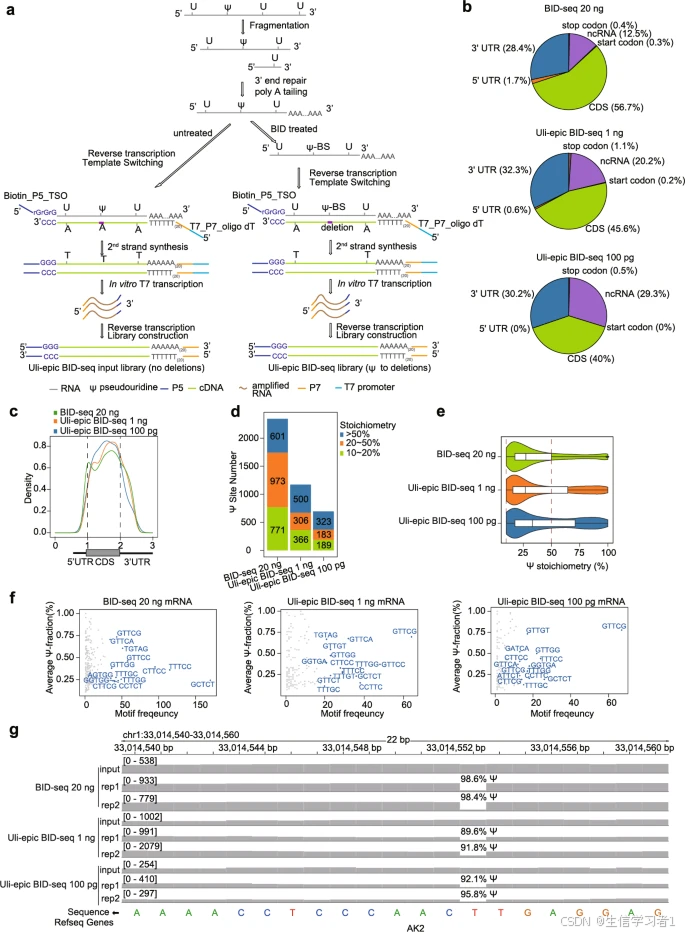
通过化学标记的高通量测序能够以单核苷酸的分辨率对 RNA 修饰进行全面、可靠的检测。然而,这些方法通常需要大量的 RNA,因为需要进行严格的处理。我们引入了 Uli-epic 这种创新的文库构建策略,能够使用 100 皮克至 1 纳克的 RNA 来分析表观转录组修饰。利用 Uli-epic BID-seq 技术,我们仅使用 500 皮克的 rRNA 去除 RNA 就能够研究野生型和胎儿生长受限小鼠神经干细胞和精子 RNA 中的假尿嘧啶(Ψ)位点。Uli-epic GLORI 则使用 10 纳克的 rRNA 去除 RNA 来量化野生型和胎儿生长受限小鼠的精子和神经干细胞中的 m6A。
自 1951 年首次发现化学修饰的 RNA 核苷酸 Ψ 以来,已鉴定出超过 170 种 RNA 的化学修饰类型[1]。这些修饰对于 RNA 的生命周期的各个方面都至关重要,包括二级结构[2]、基因表达[3,4,5,6,7];前体 mRNA 剪接[3]、核输出[8]、RNA 稳定性[9,10]以及翻译效率[11,12]。四种碱基以及核糖骨架都可以发生化学修饰,从而产生多种不同的类型,如 N6-甲基腺苷(m6A)、Ψ、m7G、N6,2′-O-二甲基腺苷(m6Am)、N1-甲基腺苷(m1A)、肌苷(I)、5-甲基胞嘧啶(m5C)、5-羟甲基胞嘧啶(hm5C)、2′-O-甲基化(Nm)和 N4-乙酰胞嘧啶(ac4C)。为了探究这些修饰的生物学功能,研究人员开发了一系列方法来分析转录组中的 RNA 修饰,包括抗体亲和力法、酶辅助法和化学辅助测序技术。
利用抗 RNA 修饰抗体来富集 RNA 片段,生成分辨率为 100 至 200 个核苷酸的图谱(例如在 m6A-seq 或 MeRIP-seq 中)——由于其易于使用的特性,已成为表观转录组学领域广泛采用的方法。这种方法极大地推动了 RNA 修饰的研究。研究人员进一步优化了 m6A-seq 策略,以在单细胞水平上描绘 RNA m6A 修饰情况[15,16,17]。然而,基于抗体的技术存在显著的局限性,包括低分辨率以及缺乏定量信息[18]。为了克服这一瓶颈,已经开发出了各种酶辅助和化学辅助技术,这些技术能够在转录组的单核苷酸分辨率水平上定量 RNA 修饰的摩尔比。例如,m6A-SAC-seq [19]、eTAM-seq [19] 和 GLORI 用于 m6A 检测 [20, 21];m1A-MAP [22]、m1A-quant-seq [23];以及用于 m1A 检测的 red-m1A-seq [24];用于 Ψ 检测的 BID-seq [25]、BACS [26] 和 PRAISE [27];用于 m5C 检测的 UBS-seq [28] 和 m5C-TAC-seq [29];AlkAniline-Seq [30] 用于 m7G 和 N3-甲基胞嘧啶(m3C)的分析;m7G-seq [31] 用于 m7G 的检测;ac4C-seq [32] 和 RedaC:T-seq [33] 用于 ac4C 的检测;以及 DAMM-seq,它同时检测 m1A、m3C、N1-甲基鸟苷(m1G)和 N2,N2-二甲基鸟苷(m2,2G)的甲基化 [34] 。
在这些方法中,酶辅助法以及几种相对温和的化学辅助法能够以纳克级的 RNA 输入量实现 RNA 修饰的定量分析。例如,用于检测 m6A 的 m6A-SAC-seq 需要 2 纳克的 mRNA [25];eTAM-seq [19] 能够在单细胞水平上进行 m6A 分析;用于检测 Ψ 的 BID-seq [25] 需要 10 纳克的 mRNA;用于检测 m5C 的 UBS-seq [28] 需要 10 纳克的 mRNA;而用于检测 m1A 的 m1A-quant-seq 需要 100 纳克的 mRNA。酶辅助法通常对 RNA 样本造成的损伤较小,但它们往往涉及易碎的酶,这会带来稳定性风险。相比之下,基于化学的方法更稳定且更易于操作,但可能会因苛刻的处理而导致 RNA 损失。例如,GLORI [20, 21] 允许对 m6A 进行绝对定量,并且操作简便且稳定,但需要 200 纳克的 mRNA,这限制了其临床适用性。在某些生物学情况下,例如在胚胎发育过程中或在处理临床样本时,可能只有几纳克甚至皮克的 rRNA 脱乏输入可用。因此,迫切需要一种稳定且易于操作的策略,能够利用化学方法在极其有限的 RNA 样本中检测 RNA 的修饰情况。
参考
- Uli-epic: profiling RNA modifications from ultra-low input samples






















 1923
1923

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










