如果你使用本代码,请引用:Changwu Wu. 2022. Pan-cancer analyses reveal molecular and clinical characteristics of cuproptosis regulators. iMeta 1: e68. https://doi.org/10.1002/imt2.68
代码编写及注释:农心生信工作室。
写在前面
气泡图 (bubble map) 具有多种衍生的形式,在气泡图中加入线段可以展现一些变量之间的相关性。本期我们挑选2022年12月5日刊登在iMeta上的Pan-cancer analyses reveal molecular and clinical characteristics of cuproptosis regulators- iMeta|湘雅医院刘庆组-泛癌分析揭示铜死亡调节子的临床和分子特征,选择文章的Figure 9A进行复现,基于ggplot2包,讲解和探讨棒棒糖图的绘制方法,先上原图:

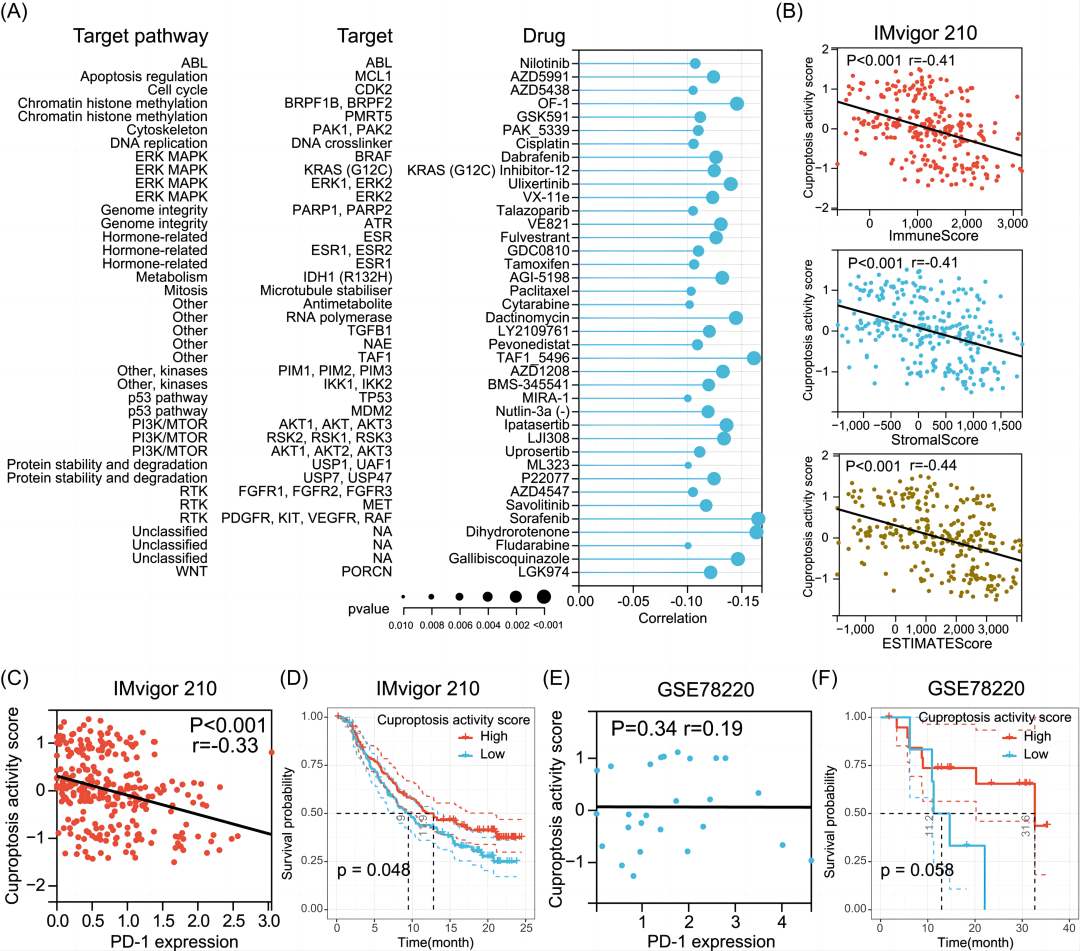
接下来,我们将通过详尽的代码逐步拆解原图,最终实现对原图的复现。
R包检测和安装
01
安装核心R包ggplot2以及一些功能辅助性R包,并载入所有R包。
if (!require("ggplot2"))
install.packages('ggplot2')
if (!require("cowplot"))
install.packages('cowplot')
# 加载包
library(ggplot2)
library(cowplot)生成测试数据
02
原图表示铜死亡活性与药物敏感性(IC50值)之间的相关性。每一行代表一种药物和药物靶标。线的长度代表相关系数。点的大小代表统计学意义,点越大,统计学显著性越高。我们根据原图特征生成测试数据test.CSV。相关示例数据可以在GitHub上下载: https://github.com/iMetaScience/iMetaPlot/tree/main/221220Lollipops
#读取test.CSV对应表
df <- read.csv("test.CSV",header = T)
#固定列的顺序
df$Drug <- factor(df$Drug,levels = df$Drug)
df$Target <- factor(df$Target,levels = df$Target)
df$Target.Pathway <- factor(df$Target.Pathway,levels = df$Target.Pathway)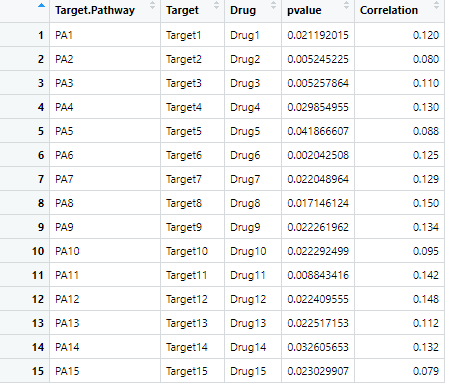
作图预览
03
开始作图,首先根据Grug一列做y轴,画一个最基本的气泡图:
p1 <- ggplot(df,aes(Correlation,Drug))+
geom_point(aes(size = pvalue),color = "skyblue")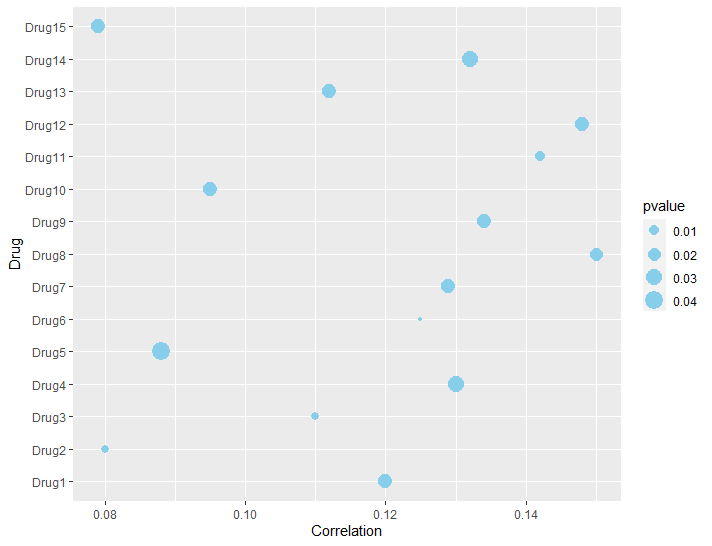
04
添加线段,这里使用的关键函数是geom_segment,它可以通过映射x坐标和y坐标的起点终点,最终得到有方向由长度的向量。本例中所有线段都是垂直与x轴,长度代表相关性,因此需要先根据相关性生成一个包括x和y终点和起点的数据框:
#生成线段数据框
segment_df <- data.frame(x = 0,y = seq(1:15),xend = df$Correlation,yend = seq(1:15))
p1 <- ggplot(df,aes(Correlation,Drug))+
geom_point(aes(size = pvalue),color = "skyblue")+
geom_segment(segment_df,mapping = aes(x = x, y = y, xend = xend, yend = yend),size = 0.8,color = "skyblue")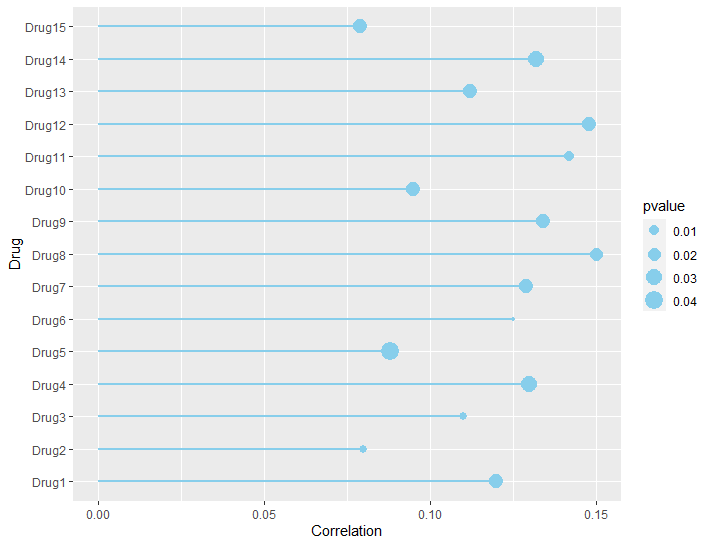
05
调整参数美化图片:
p1 <- ggplot(df,aes(Correlation,Drug))+
geom_point(aes(size = pvalue),color = "skyblue")+
geom_segment(segment_df,mapping = aes(x = x, y = y, xend = xend, yend = yend),size = 0.8,color = "skyblue")+
scale_x_continuous(limits = c(0,0.152), #设置x轴范围
expand = expansion(mult = 0))+ #expansion函数的参数mult: 百分比间距,可以接受一个向量
theme(panel.background = element_blank(), #删除背景
panel.grid = element_line (colour = "lightgrey"), #设置网格颜色
panel.border = element_rect(fill = NA,colour = "black",size = 0.8))+ #设置边框
ylab("") #删除y轴名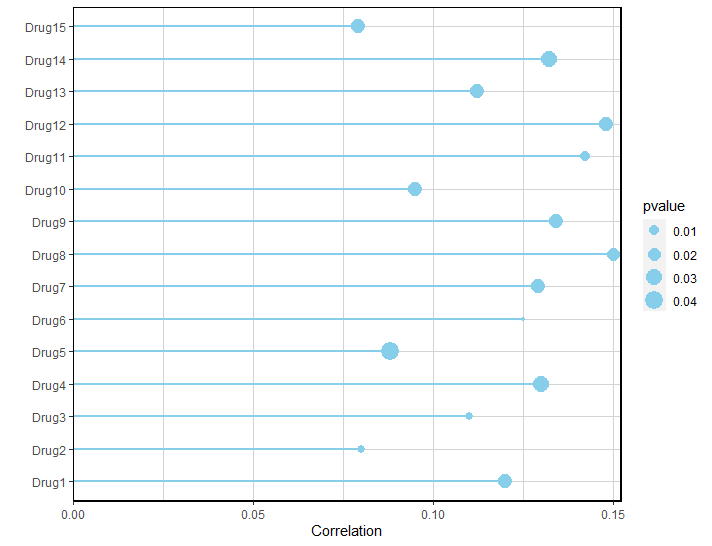
06
通过ggplot2绘制两幅只显示Target.Pathway和Target列名的图片,最后通过cowplot包将所有图片组合。
#Target
p2 <- ggplot(df,aes("",Target))+
theme(panel.background = element_blank(),#去除背景
panel.border = element_blank(), #去除边框
axis.ticks.y = element_blank(), #去除y轴刻度
axis.ticks.x = element_blank())+ #去除x轴刻度
xlab("")+ #去除x轴标题
ylab("")+ #去除y轴标题
coord_fixed(ratio = 10) #将y轴和x轴标度比例设置为10:1
#Target.Pathway,同上
p3 <- ggplot(df,aes("",Target.Pathway))+
theme(panel.background = element_blank(),#去除背景
panel.border = element_blank(),
axis.ticks.y = element_blank(),
axis.ticks.x = element_blank())+
xlab("")+
ylab("")+
coord_fixed(ratio = 10)
p <- plot_grid(p3,p2,p1,
ncol = 3, #三张图片分三列合并
rel_widths = c(1,1,2), #三列所占比例为1:1:2
labels = c("Target.Pathway","Target","Drug"), #每一幅子图片标题名
label_size = 8) #标题大小
完整代码
if (!require("ggplot2"))
install.packages('ggplot2')
if (!require("cowplot"))
install.packages('cowplot')
# 加载包
library(ggplot2)
library(cowplot)
#读取test.CSV对应表
df <- read.csv("test.CSV",header = T)
#固定列的顺序
df$Drug <- factor(df$Drug,levels = df$Drug)
df$Target <- factor(df$Target,levels = df$Target)
df$Target.Pathway <- factor(df$Target.Pathway,levels = df$Target.Pathway)
#生成线段数据框
segment_df <- data.frame(x = 0,y = seq(1:15),xend = df$Correlation,yend = seq(1:15))
p1 <- ggplot(df,aes(Correlation,Drug))+
geom_point(aes(size = pvalue),color = "skyblue")+
geom_segment(segment_df,mapping = aes(x = x, y = y, xend = xend, yend = yend),size = 0.8,color = "skyblue")+
scale_x_continuous(limits = c(0,0.152), #设置x轴范围
expand = expansion(mult = 0))+ #expansion函数的参数mult: 百分比间距,可以接受一个向量
theme(panel.background = element_blank(), #删除背景
panel.grid = element_line (colour = "lightgrey"), #设置网格颜色
panel.border = element_rect(fill = NA,colour = "black",size = 0.8))+ #设置边框
ylab("") #删除y轴名
#Target
p2 <- ggplot(df,aes("",Target))+
theme(panel.background = element_blank(),#去除背景
panel.border = element_blank(), #去除边框
axis.ticks.y = element_blank(), #去除y轴刻度
axis.ticks.x = element_blank())+ #去除x轴刻度
xlab("")+ #去除x轴标题
ylab("")+ #去除y轴标题
coord_fixed(ratio = 10) #将y轴和x轴标度比例设置为10:1
#Target.Pathway,同上
p3 <- ggplot(df,aes("",Target.Pathway))+
theme(panel.background = element_blank(),#去除背景
panel.border = element_blank(),
axis.ticks.y = element_blank(),
axis.ticks.x = element_blank())+
xlab("")+
ylab("")+
coord_fixed(ratio = 10)
pdf("Figure 9A.pdf",width = 11, height = 5.7)
plot_grid(p3,p2,p1,
ncol = 3, #三张图片分三列合并
rel_widths = c(1,1,2), #三列所占比例为1:1:2
labels = c("Target.Pathway","Target","Drug"), #每一幅子图片标题名
label_size = 8) #标题大小
dev.off()
#> quartz_off_screen
#> 2以上数据和代码仅供大家参考,如有不完善之处,欢迎大家指正!
更多推荐
(▼ 点击跳转)
iMeta封面 | 宏蛋白质组学分析一站式工具集iMetaLab Suite(加拿大渥太华大学Figeys组)
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
iMeta | 华南农大陈程杰/夏瑞等发布TBtools构造Circos图的简单方法
往期回顾
🔗:https://onlinelibrary.wiley.com/toc/2770596x/2022/1/1
🔗:https://onlinelibrary.wiley.com/toc/2770596x/2022/1/2
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://wileyonlinelibrary.com/journal/imeta
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
微信公众号
iMeta
责任编辑
微微
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集

























































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








