转自:ChallengeHub
A.监督学习
1.EDA(Exploratory Data Analysis)
2.K-Nearest Neighbors(KNN)
3.线性回归
4.交叉验证(CV)
5.正则化回归
6.ROC曲线与逻辑回归
7.超参数调优
8.SVM
B.无监督学习
1.Kmeans聚类
2.聚类效果评价
3.标准化
4.层次分析法
5.T-分布随机近邻嵌入(T-SNE)
6.主成分分析(PCA)
监督学习
可以查看机器学习全面教程-有监督学习篇
无监督学习
无监督学习: 它使用无标记的数据,并从无标记的数据中发现隐藏的模式信息。例如,有些骨科病人数据没有标签,你不知道哪个骨科病人是正常的,哪个是不正常的。
1.Kmeans聚类
让我们尝试我们的第一个无监督的方法,即KMeans Cluster
KMeans聚类:该算法基于所提供的特征迭代地将每个数据点分配给K组中的一个。基于特征相似性对数据点进行聚类
KMeans(n_clusters = 2): n_clusters = 2表示创建2个簇群
先看数据的分布
# As you can see there is no labels in data
data = pd.read_csv('column_2C_weka.csv')
plt.scatter(data['pelvic_radius'],data['degree_spondylolisthesis'])
plt.xlabel('pelvic_radius')
plt.ylabel('degree_spondylolisthesis')
plt.show()

再看通过Kmeans聚类分类效果
# KMeans Clustering
data2 = data.loc[:,['degree_spondylolisthesis','pelvic_radius']]
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters = 2)
kmeans.fit(data2)
labels = kmeans.predict(data2)
plt.scatter(data['pelvic_radius'],data['degree_spondylolisthesis'],c = labels)
plt.xlabel('pelvic_radius')
plt.xlabel('degree_spondylolisthesis')
plt.show()

2.聚类效果评价
我们把数据分成两组。这是正确的聚类吗? 为了评估聚类效果,我们将使用交叉表。
有两个集群,分别是0和1
标签0包括138例异常患者和100例正常患者
标签1包括72例异常患者和0例正常患者
两个簇群中大多数为异常患者。
# cross tabulation table
df = pd.DataFrame({'labels':labels,"class":data['class']})
ct = pd.crosstab(df['labels'],df['class'])
print(ct)

上面存在一个问题是,我们事先知道了数据中是几分类问题,但是如果不知道呢? 那这有点像KNN或回归中的超参数问题了。
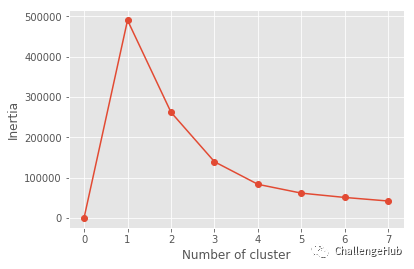
inertia:簇群与每个样本之间的距离
更小的inertia意味着更多的簇
簇群的最佳数量是多少?
更小的inertia,没有太多的簇群平衡,所以我们可以选择elbow
# inertia
inertia_list = np.empty(8)
for i in range(1,8):
kmeans = KMeans(n_clusters=i)
kmeans.fit(data2)
inertia_list[i] = kmeans.inertia_
plt.plot(range(0,8),inertia_list,'-o')
plt.xlabel('Number of cluster')
plt.ylabel('Inertia')
plt.show()
3.标准化
标准化对于监督学习和非监督学习都很重要,可以消除量纲影响
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
scalar = StandardScaler()
kmeans = KMeans(n_clusters = 2)
pipe = make_pipeline(scalar,kmeans)
pipe.fit(data3)
labels = pipe.predict(data3)
df = pd.DataFrame({'labels':labels,"class":data['class']})
ct = pd.crosstab(df['labels'],df['class'])
print(ct)

4.层次分析法
竖线是簇
树状图高度: 归并簇之间的距离
方法= 'single': 表示簇的最近点
from scipy.cluster.hierarchy import linkage,dendrogram
merg = linkage(data3.iloc[200:220,:],method = 'single')
dendrogram(merg, leaf_rotation = 90, leaf_font_size = 6)
plt.show()

5.T-SNE
学习率:正常情况下50-200
T-sne只有fit_transform
from sklearn.manifold import TSNE
model = TSNE(learning_rate=100)
transformed = model.fit_transform(data2)
x = transformed[:,0]
y = transformed[:,1]
plt.scatter(x,y,c = color_list )
plt.xlabel('pelvic_radius')
plt.xlabel('degree_spondylolisthesis')
plt.show()

6.PCA
基本的降维技术
第一步是去相关:
旋转数据样本以使其与轴对齐
移动数据样本,使其均值为零
没有信息丢失
Fit():学习如何移动样本
Transform():应用学到的转换。也可以应用测试集
由此产生的PCA特征不是线性相关的
主成分:是方差方向
# PCA
from sklearn.decomposition import PCA
model = PCA()
model.fit(data3)
transformed = model.transform(data3)
print('Principle components: ',model.components_)

# PCA variance
scaler = StandardScaler()
pca = PCA()
pipeline = make_pipeline(scaler,pca)
pipeline.fit(data3)
plt.bar(range(pca.n_components_), pca.explained_variance_)
plt.xlabel('PCA feature')
plt.ylabel('variance')
plt.show()

第二步:内在维度: 需要特征数量来近似数据背后的基本思想
当样本具有任意数量的特征时,主成分分析识别内在维数
内在维度 = 具有显著方差的PCA特征的数量
为了选择内在维度,尝试所有的方法并找到最佳的精度
# apply PCA
pca = PCA(n_components = 2)
pca.fit(data3)
transformed = pca.transform(data3)
x = transformed[:,0]
y = transformed[:,1]
plt.scatter(x,y,c = color_list)
plt.show()

建议阅读:
【时空序列预测第一篇】什么是时空序列问题?这类问题主要应用了哪些模型?主要应用在哪些领域?
【AI蜗牛车出品】手把手AI项目、时空序列、时间序列、白话机器学习、pytorch修炼
公众号:AI蜗牛车
保持谦逊、保持自律、保持进步
个人微信
备注:昵称+学校/公司+方向
如果没有备注不拉群!
拉你进AI蜗牛车交流群
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








