load("D:/ARDS_scripts_1012/ARDS/Step2_harmony_f200_R3/0805/cluster_merge/sepsis_cluster_merge.rds")## 改路径
as.matrix(table(Idents(All.merge), All.merge$stim))
library(Seurat)
#'Dlat','Lias','Pdha1',
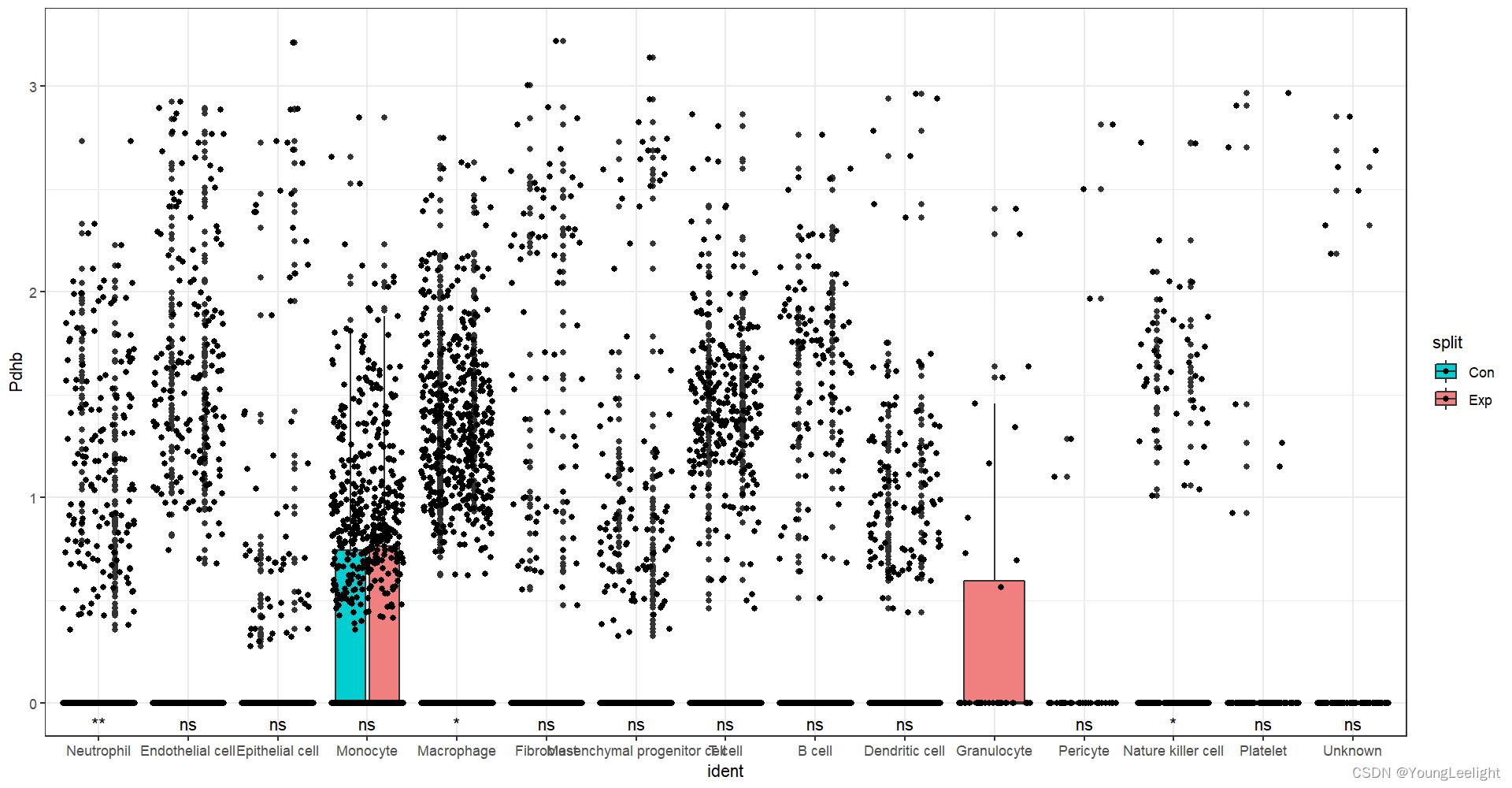
p=VlnPlot(All.merge,features = c('Pdhb'),split.by = 'stim') #+geom_split_violin()
head(p)
mydata=p$data
head(mydata)
head(mydata)
library(ggplot2)
library(ggsignif)
library(ggpubr)
ggplot(p$data,aes(ident,Pdhb, fill = split)) +
geom_boxplot(scale = "width", adjust =.02, trim = TRUE) +
geom_jitter()+
stat_compare_means(aes(group = split),label="p.signif", label.y.npc = "bottom", vjust = 2)+
scale_fill_manual(values = c("#00CED1", "#F08080"))+RotatedAxis()+
theme_bw()
##'Dlat','Lias','Pdha1'
getwd()
Sys.setenv(LANGUAGE = "en")
getwd()
load("D:/Win10 System/Documents/WeChat Files/wxid_f27yna03e0v622/FileStorage/File/2022-11/merge.Rdata")
library(Seurat)
head([email protected])
dim(merge) #[1] 15499 10979
table(merge$stim)
DimPlot(merge,label = T,group.by = "anno")
VlnPlot(merge,features = "FPR1")
merge=FindVariableFeatures(merge,nfeatures = nrow(merge))
Assays(merge)
slotNames(merge)
head(merge@assays$RNA@data)
DotPlot(merge,features = "Fpr1")
which(rownames(merge)=="Fpr1")
rownames(merge)
Idents(merge









 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 444
444











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










