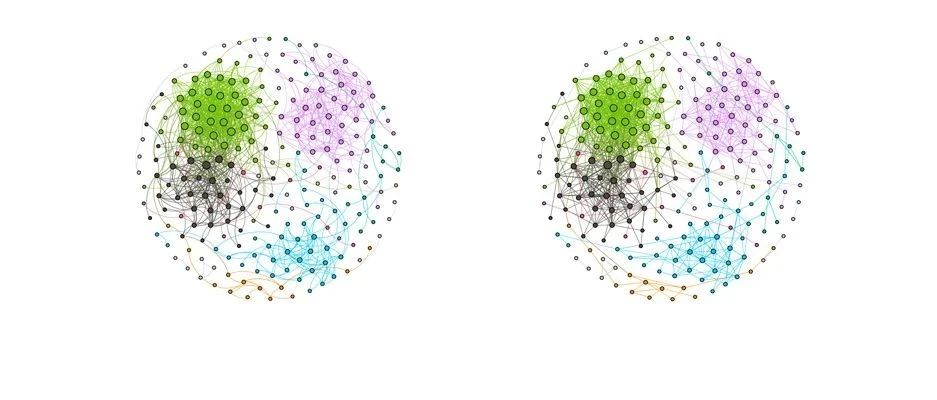
| 撰文:莫北
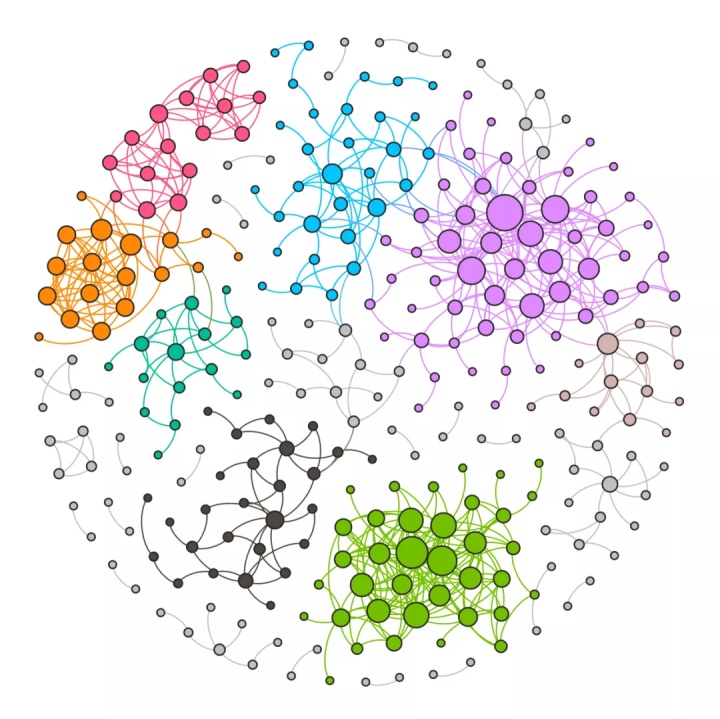
在之前《如何用Gephi绘制漂亮的网络图》一文中,已经为大家介绍过如何使用Gephi绘制下图这样的共发生网络图(Co-occurrence network),该文主要是介绍通过准备“边文件”和“点文件”来创建网络图。
但有时候绘图比较容易,难的是准备数据。
前文也提过,最常见的共发生网络分析过程是先计算相关系数矩阵,接着使用igraph生成gml格式的网络图文件,最后导入Gephi对网络图进行个性化绘制。
当然,如果你不愿使用R语言计算,也可以使用OmicShare Tools中的组内相关性分析工具计算得到相关系数矩阵,然后整理成“边文件”,使用Gephi或Cytoscape做个性化绘图。
工具网址: https://www.omicshare.com/tools/
但如果数据量较大,手工整理显然非常费时费力,有OmicShare用户在OmicShare Forum中留言:“数据的准备太难了!”
因此,下面我将从最初的OTU绝对丰度表开始,介绍如何使用R语言进行网络图数据的准备、网络图文件生成和网络图绘制。
本文的重点内容是数据的过滤、相关系数的计算和“graphml”格式网络图文件的生成。
数据导入与过滤
为了便于大家练习使用,本文所用到的范例数据已上传到OmicShare论坛上,







 本文详细介绍了如何使用R语言处理OTU绝对丰度表,计算相关系数,生成graphml格式网络图文件,并使用Gephi进行网络图的绘制和个性化调整。重点内容包括数据过滤、相关系数计算、网络图生成和使用igraph及Gephi的绘图技巧。
本文详细介绍了如何使用R语言处理OTU绝对丰度表,计算相关系数,生成graphml格式网络图文件,并使用Gephi进行网络图的绘制和个性化调整。重点内容包括数据过滤、相关系数计算、网络图生成和使用igraph及Gephi的绘图技巧。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








