CRBPDL: Identification of circRNA-RBPinteraction sites using an ensemble neural network approach
期刊:PLOS COMPUTATIONAL BIOLOGY
中科院分区:二区
代码和数据集链接:https://github.com/nmt315320/ CRBPDL.git
摘要
circRNAs是通过反向剪接机制产生的具有特殊环状结构的非编码RNA,环状 RNA 可以直接与 RNA 结合蛋白 (RBP) 结合,并在多种生物活动中发挥重要作用。
在我们的工作中,我们建立了一种新的计算预测器 CRBPDL,它基于集成深度网络来识别 circRNA-RBP 相互作用位点。首先,采用5种编码方案为模型训练提供全面的特征信息,包括k-核苷酸频率(KNF)、Doc2vec、电子-离子相互作用赝势(EIIP)、核苷酸化学性质(CCN)和累积核苷酸频率(ANF)。由于特征描述符的分布不同,我们首先将卷积滤波器分别应用于特征,然后将它们连接成特征矩阵。随后,为了从特征描述符中自动提取高阶局部和全局上下文信息,我们构建了一个深度神经网络架构,该架构由一个深度多尺度残差网络(ResNet)和一个具有自注意力机制的双向门控循环单元组成(BiGRUs) 网络组成。我们使用深度多尺度残差网络 (MRSN) 和 BiGRU 来学习局部和全局上下文信息,并且可以有效地表示高级特征。然后,使用自注意力机制来训练模型的鲁棒性。经过模型训练和选择,我们可以得到优化后的深度学习模型(为方便起见,集成前的深度学习模型命名为“sig-CRBPDL”)。最后,使用 AdaBoost 算法集成深度学习模型。
整体模型框架

数据集
我们总共获得了与 37 个环状 RNA 数据集相关的 32,216 个环状 RNA。此外,我们比较了 CRBPDL 识别线性 RNA -RBP 相互作用位点的效率。我们下载了线性 RNA 数据集,其中包括 CLIP-Seq 数据与 31 个RBP 相结合的线性 RNA 数据集。每个数据集有 5000 个训练集和 1000 个测试集。
特征编码
k-核苷酸频率
KNF 描述了序列中所有可能的 k 个核苷酸的多核苷酸的频率。我们取k = 1、2、3,即单核苷酸组成频率、二核苷酸组成频率和三核苷酸组成频率。
Doc2vec

Electron–ion interaction pseudopotential(EIIP)
序列中可能出现的四个字符(即“A”、“T”、“C”、“G”)的EIIP值分别为0.1260、0.1335、0.1340和0.0806。EIIP 编码方法可用于将 DNA 序列编码为数字载体。例如,AATCCGA 编码是一个由 (0.1260, 0.1260,0.1335, 0.1340, 0.1340, 0.0806,0.1260) 组成的数字向量。
Chemical characteristic of nucleotide (CCN)
每个核苷酸具有三种化学特性(CCN):化学功能(包括氨基和酮基)、环状结构(包括双环嘌呤和单环嘧啶)和氢键(包括弱氢键和强氢键)。对于环结构,A和G属于嘌呤,编码为1,C和T属于嘧啶,编码为0。对于化学功能,A和C属于氨基,编码为1; G和T属于酮基,编码为0。对于氢键,A和T属于弱氢键,编码为1,而C和G属于强氢键,编码为0。例如AATCCGA可以编码为 (1,1,1,1,1,1,0,0,1,0,1,0,0,1,0,1,0,0,1,1,1)
Accumulated nucleotide frequency(累积的核苷酸频率)

多尺度残差网络
为了获得丰富的特征信息,构建了一个多尺度的 CNN 层来捕获高级特征。我们对五个特征使用卷积滤波器,卷积核为 128,然后级联。MSRN 框架包含一个浅层 CNN 提取层,卷积核的大小为 3。然后,使用包含 6 个级联多尺度残差块(MSRB)模块的 inception 模块,卷积核为 64。每个MSRB 包括一个 3 个卷积层。基于分层特征融合结构(HFFS),将每个MSRB的输出组合起来进行全局特征融合。随后,输入一层卷积核后,有192个滤波器,一个1×1的卷积可以增减通道数,跨通道组织信息,增加特征变换,计算量小,非线性变换为提高网络表达能力。之后,有一个 dropout 值为 0.4 的合并层。
实验细节
Model performance under different learning rate
分析了三种学习率衰减方案(阶梯式衰减学习率方案、线性学习率衰减方案、多项式学习率方案)。两个固定的学习率(0.002, 0.0005)。

Model Performance under different feature encoding schemes
我们分析了五种特征编码的不同性能。可以发现,在 37 个数据集上,Doc2vec 编码方案相对优于其他四个。说明 RBP 结合位点的全局文本特征比较明显。我们的词向量模型似乎已经从语义中学习了微妙的序列上下文,从而提高了识别性能。对于circRNA数据集,实验结果表明,本文提出的自学习词向量编码方案具有良好的应用前景。

Performance of neural network structures with different depths
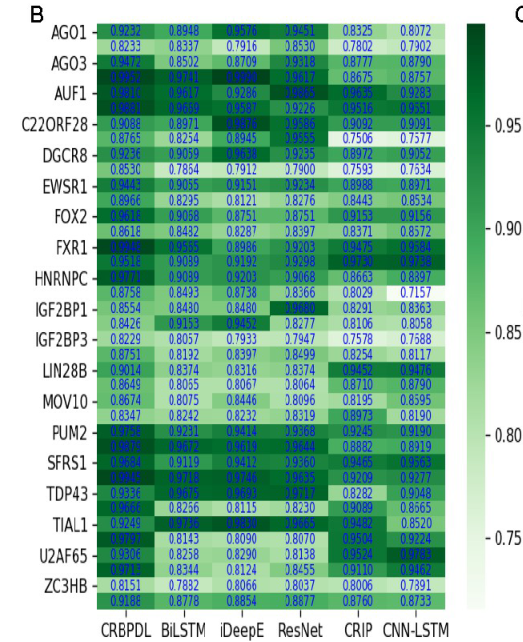
我们将其性能与 5 种结构进行了比较。CNN-LSTM(包括两个双向 LSTM 层和两个全连接层)。iDeepE(结合了全局网络和局部神经网络的输出特征,两层局部多通道神经网络(卷积、ReLU和max pooling)表达高层特征,然后将特征图输入到两个全连接层)。ResNet(使用21层的局部多通道网络,在两个网络之间插入快捷连接,使网络成为对应的残差网络)。CRIP (使用了两层,一个提取高级特征的 CNN 和一个获取序列长期依赖关系的 RNN)。CNN-BiLSTM (包括两个双向 LSTM 和两个全连接层)。
我们进一步分析了 MSRN 和 BiGRU 的不同性能,可以发现,虽然两者的差异比较小,但是MSRN的效果明显优于BiGRU,说明在CRBPDL模型中,MSRN的贡献更大。

Prediction performance of CRBPDL on 37 circRNA data sets

Prediction performance of CRBPDL on 31 linear data sets

Evaluation metrics
我们采用了五个评估指标:敏感性(SN)、特异性(SP)、准确性(ACC)、马修相关系数(MCC)和AUC ,定义如下:
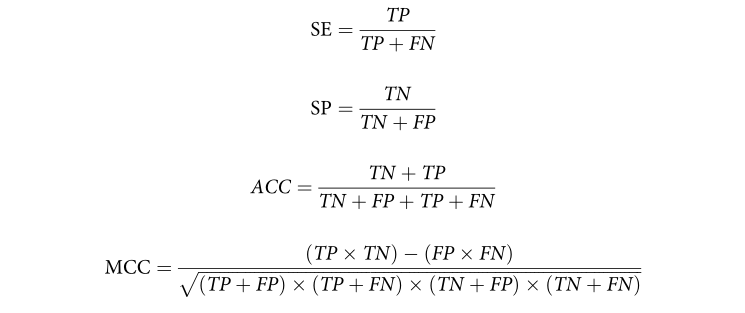
其中 TP、TN、FP 和 FN 分别表示真阳性、真阴性、假阳性和假阴性的数量。此外,曲线下面积 (AUC) 是 ROC 曲线下的面积
Conclusion
设计了一种新的深度学习方法,称为 CRBPDL。(用于环状 RNA-RBP 相互作用位点识别),基于 MSRN 框架,CRBPDL 首先将五种特征连接成一个特征向量。然后使用 MSRB 自动探索更高级的局部或全局上下文依赖关系并获得高级序列特征。随后,将每个 MSRB 的输出组合起来进行全局分层特征融合。并添加 selfAttention 以掌握更多关键和相关的特征并提高预测性能。最后,基于Adaboost算法构建了一个集成的深度学习网络。为了验证 CRBPDL,我们对 circRNA 和线性 RNA 的结合位点进行了预测,并评估了不同方法的性能。 37个环状RNA数据集和31个线性RNA的比较不仅证明了我们方法的有效性,而且显示了该模型在识别环状RNA-RBP相互作用位点方面的潜力。目前,关于已知 RBP 结合位点的数据很少。正负样本不平衡。因此,最重要的是未来的研究是扩展数据集,收集与circRNA、lncRNA或其他RNA结合的RBP结合位点,探索它们的结合特性,开发通用预测软件。







 研究提出了一种名为CRBPDL的新型计算预测器,利用集成深度网络识别circRNA-RBP相互作用位点。特征编码包括k-核苷酸频率、Doc2vec、EIIP、CCN和ANF。模型结构由多尺度残差网络和带有自注意力机制的双向门控循环单元组成,通过AdaBoost算法集成。实验结果证明了CRBPDL在识别circRNA-RBP相互作用位点上的有效性。
研究提出了一种名为CRBPDL的新型计算预测器,利用集成深度网络识别circRNA-RBP相互作用位点。特征编码包括k-核苷酸频率、Doc2vec、EIIP、CCN和ANF。模型结构由多尺度残差网络和带有自注意力机制的双向门控循环单元组成,通过AdaBoost算法集成。实验结果证明了CRBPDL在识别circRNA-RBP相互作用位点上的有效性。


















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








