实验室最近收集了一个大型细胞数据集,准备发一篇论文,导让我跑一下hovernet对细胞进行分割,忙叨了一天啥也没弄出来,准备用CSDN记录一下我的心酸学习历程。
2024年4月11日
一、创建环境
1、


安装了上面的,接下来就开始安装requirements了
PS: pip install torch==1.6.0 torchvision==0.7.0 -i https://pypi.mirrors.ustc.edu.cn/simple/ 这句代码来安装清华镜像源~
pip install -r requirements.txt 安装requirements文件
事已至此,环境已经准备好了! 我去上个厕所了~~~~~
---------------------------------------------------------------------------------------------------------------------------------
二、图像处理

这里写了必须先在这个ex.py进行数据处理,然后得到可以用来训练的数据集
结果如下 生成的新文件~

三、配置文件修改

Step1:设置数据文件路径 在config.py
Step2:设置checkpoints 的存储路径(这个checkpoints是啥我还不太清楚)

Step3:下载权重文件 文件里面写的存在着,我就建了个文件夹然后放进去了

Step4:修改参数之类的 我先没改一会跑跑看看
四、训练前检查

开始报错 一直都是一些数组的问题 所以想到调用一下生成的npy文件看一下

里面的数组是五维的,应该是生成的时候用错了函数,因为 这里明确说了,分割是要四维,分割加分类才需要五维,回去查看ex.py
这里明确说了,分割是要四维,分割加分类才需要五维,回去查看ex.py
ps:报错显示pytorch版本和cuda版本不兼容,升级pytorch版本
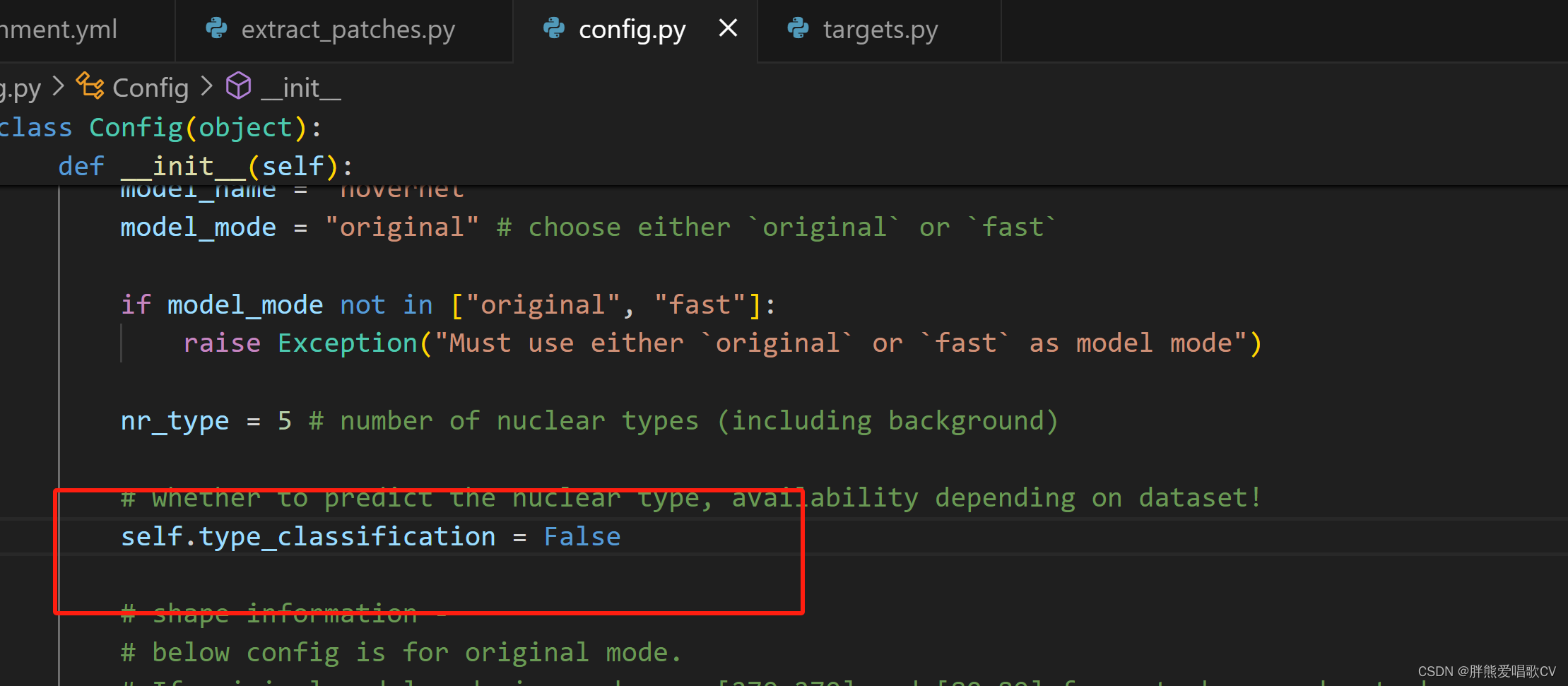
ex.py文件里
不知道是不是这个分类要改成false 一会试试(等待pytorch升级中)
这里也被改了
今天先不改了 ,明天再试试
---------------------------------------------------------------------------------------------------------------------------------
4月15日
气shu我了!环境没配好!重新来一遍!!!!
1、创建pyhton版本为3.6的虚拟环境
2、安装pytorch
pip install torch==1.6.0 torchvision==0.7.3、安装cuda
conda install cudatoolkit==指定版本
查看cudatoolkit版本命令 conda search cudatoolkit
参考:使用虚拟环境conda安装不同版本的cuda,cudnn,pytorch_conda安装cuda-CSDN博客
然后报错了,是通道的问题
查看现有通道命令:conda config --get
下10.2没成功 下的11.8先试试吧
现在extra文件可以跑通,但是还是训练不了。。。。可能是数据集的问题,明天试试下个新的数据集(创建了新的环境hovernet)
我的数据集最后一位标签是0,看见一行代码,0好像是背景,明天改一下改成1试试



---------------------------------------------------------------------------------------------------------------------------------
4月16日
1、 这里面写了三个数据集的数据预处理方案。extract_patches.py里面进行了调用。
这里面写了三个数据集的数据预处理方案。extract_patches.py里面进行了调用。
2、数据集下载 我在Google上搜了一下,有个好人给出了
链接在这里 CPM15 and CPM17 dataset · Issue #5 · vqdang/hover_net · GitHub
引用数据集写论文请注明原论文出处!!!!!!!!!!!!!!!!!!!!!科研不易!!

在.mat 文件中,inst_map 这个键(key)所对应的值包含了实例分割的标注信息。这里的“实例分割”指的是图像中每个独立物体(本例中为细胞核)被识别并区分的过程。
inst_map 中的内容是一个图像,其中每个像素的值代表了不同的实例(细胞核)。这些值是唯一的整数,用来标识每一个独立的细胞核。这些整数的范围从 0 到 N,其中:
- 0 代表背景:也就是说,图像中所有值为0的像素都是背景,不属于任何一个细胞核。
- 1 到 N 代表不同的细胞核:每一个从1到N的整数都对应一个特定的细胞核,不同的整数代表不同的细胞核。这样,图像上相同数值的像素群组就形成了一个细胞核的图像区域。
通过这种方式,可以清楚地区分和识别图像中的每一个细胞核,每个细胞核都有一个独一无二的标识符(整数值),这对于进行生物医学图像分析等应用是非常有用的。
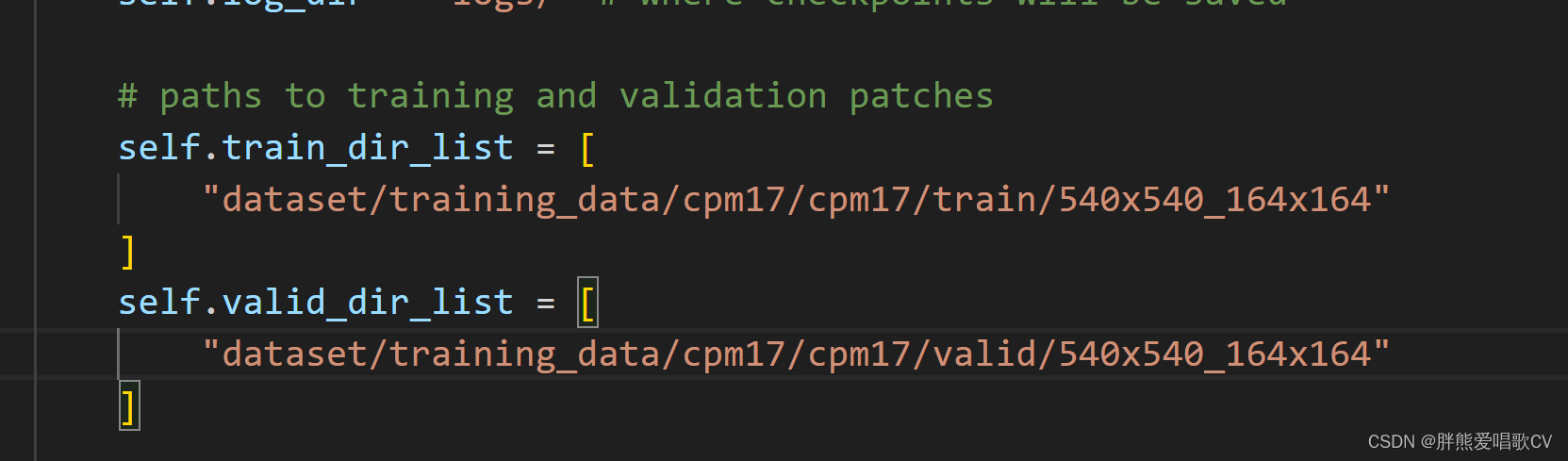
下载了cpm17数据集 然后修改extract_patches.py文件如下

2、运行extract_patches.py 得到了处理后的cpm17
3、修改config文件里的数据路径(下图是还没改的,我先去修改毕业论文了,886!PS:看着电视敲代码真的会效率加倍!小城故事多真好看!)

---------------------------------------------------------------------------------------------------------------------------------
4月17日 喝完酒回来了,敲代码!
昨天下载好了新的数据集,然后按部就班的开始做官方给出的四个步骤。
一、
1、在config.py文件中设置数据路径
2、设置checkpoints存储路径

3、设置预训练权重文件路径
4、设置参数
二、
出现的问题都是因为ex文件处理数据集之后的问题,下面来读一下ex.py文件
4月25 实验有了新进展
import numpy as np
import matplotlib.pyplot as plt
# 修改此路径到你的 .npy 文件位置
data = np.load('xxxx')
# # 输出数据形状
# print('Data Shape:', data.shape)
# # 选择性地可视化数据
# # 例如,可视化第一个通道
# plt.imshow(data[:, :, 0], cmap='gray')
# plt.colorbar()
# plt.title('Channel 1 Visualization')
# plt.show()
# 如果数据不是 uint8 类型或数值范围不在 0-255 之间,需要先进行转换
if data.dtype != np.uint8:
# 归一化到 0-1 范围
normalized_data = (data - data.min()) / (data.max() - data.min())
# 将数据缩放到 0-255 并转换为整型
data = (normalized_data * 255).astype(np.uint8)
# 显示前三个通道作为 RGB 图像
plt.imshow(data[:, :, :3])
plt.title('RGB Visualization')
plt.show()
-----------------------------------------------分割线-------------------------------------------------------------------------
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.colors import ListedColormap
# 加载.npy文件
data = np.load('0.raw.0.npy')
# 获取每个像素点最可能的类别
prediction = np.argmax(data, axis=-1)
# 创建颜色映射
colors = [
(0, 0, 0.5), # 深蓝色,肿瘤
(0.5, 0.7, 1.0), # 浅蓝色,基质
(0, 0.5, 0), # 深绿色,炎症
(0.5, 1, 0.5), # 浅绿色,坏死
(1.0, 1.0, 0) # 黄色,其它区域
]
cmap = ListedColormap(colors)
# 可视化分割结果
plt.figure(figsize=(10, 10))
plt.imshow(prediction, cmap=cmap)
plt.colorbar(ticks=range(len(colors))) # 添加颜色条
plt.title('Semantic Segmentation Map')
plt.show()

>>> from tiatoolbox.models.architecture.hovernet import HoVerNet
>>> import torch
>>> import numpy as np
>>> batch = torch.from_numpy(image_patch)[None]
>>> # image_patch is a 256x256x3 numpy array
>>> weights_path = "A/weights.pth"
>>> pretrained = torch.load(weights_path)
>>> model = HoVerNet(num_types=6, mode="fast")
>>> model.load_state_dict(pretrained)
>>> output = model.infer_batch(model, batch, on_gpu=False)
>>> output = [v[0] for v in output]
>>> output = model.postproc(output)
存在ok.ipynb 文件中,没找到权重文件
checkpoint:hover-net-pytorch-weights - Google 云端硬盘

下载好了权重文件 但是数据没处理好,下班 明天出去开会
---------------------------------------2024.5.1 实验结果图--------------------------------------------------------------
























 4992
4992

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








