动机
免疫疗法的日益应用突显了肿瘤微环境(TME)在影响治疗结果中的关键作用。测序技术的进步能从多个角度分析TME,获得新见解。多组学数据的复杂性和大量性给分析和解释带来了挑战。为了解决问题,推出了IOBR 1.0的升级版本IOBR 2.0,作为集成工具,简化了多组学数据TME分析和可视化的过程。IOBR 2.0能系统地研究TME特征,并识别免疫治疗结果的生物标志物。
亮点
-
IOBR 2.0 提供了用于TME分析和生物标志物识别的流程
-
包含6个模块,用于RNA数据预处理、肿瘤微环境特征分析以及基因组-肿瘤微环境相互作用
-
集成了10种反卷积方法和322个与TME相关的基因特征
-
支持单细胞特征对批量RNA-seq数据表型分析
小结
大规模转录组数据集的使用极大地提高了对TME的理解,有助于开发精准的免疫疗法。多组学、scRNA-seq和空间转录组测序的日益应用带来了许多新见解,仍需要在大样本中临床验证。为了推进多组学在TME研究中的整合,升级了免疫肿瘤学生物研究(IOBR)包至IOBR 2.0,对其分析工作流程重构和标准化。IOBR 2.0提供了基于多组学数据的6个TME分析模块,包括数据预处理、TME估计、TME浸润模式识别、细胞相互作用分析、基因组与TME相互作用以及特征可视化和建模。此外,IOBR 2.0能从scRNA-seq数据中构建基因特征和参考矩阵,用于TME反卷积。用户友好的流程提供了对肿瘤-免疫相互作用的全面见解,并且详细的GitBook (https://iobr.github.io/book/)为每个模块提供了完整的手册和分析指南。
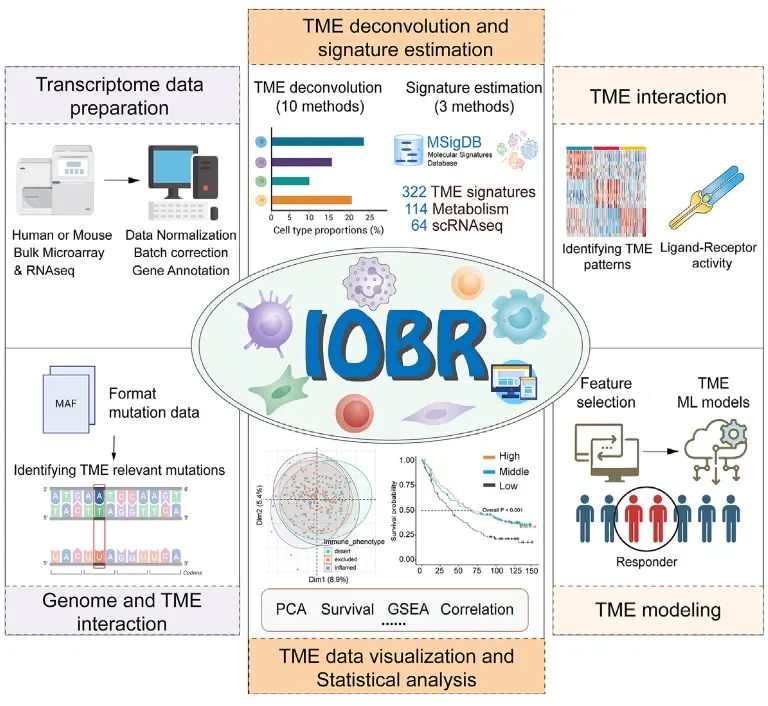
结果
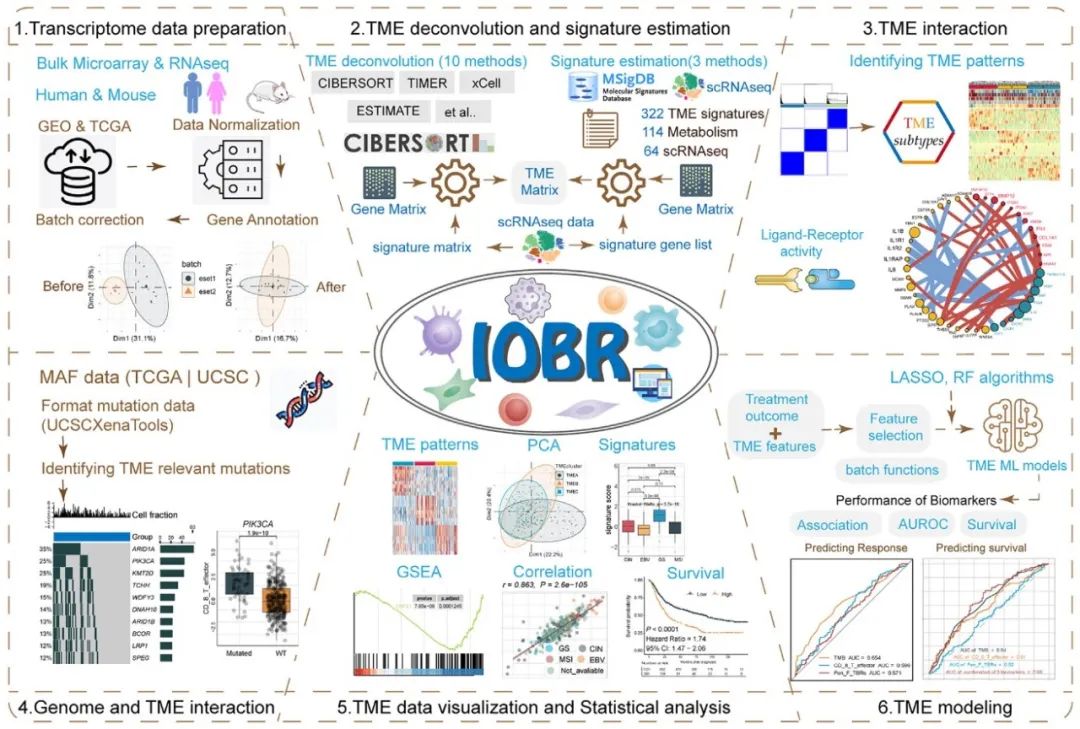
图1 描述IOBR 2.0工作流程的图形方案
IOBR 2.0 包括转录组数据准备、多种反卷积算法和特征估计方法用于微环境分析、TME模式识别、基因组与TME之间相互作用的分析、批量可视化和统计分析,以及TME建模。TME:肿瘤微环境;MAF:突变注释格式;PCA:主成分分析;GSEA:基因集富集分析;RF:随机森林;ML:机器学习;AUROC:接收者操作特征曲线下的面积。
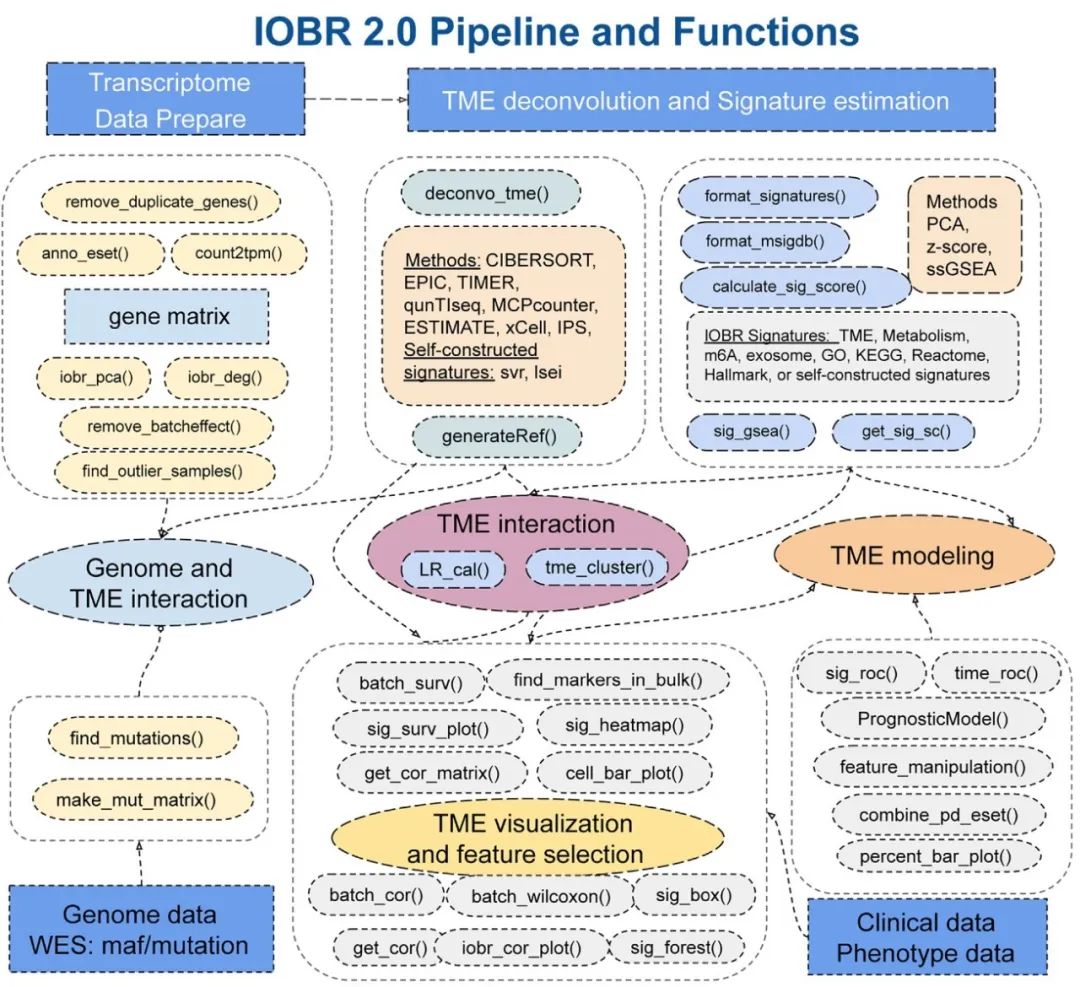
图2 IOBR 2.0由6个与数据预处理和肿瘤免疫微环境相关的分析模块组成
模块的功能包括:
1) 转录组数据的预处理;
2)估计特征评分并识别与表型相关或用户构建的特征,同时解码TME结构;
3)识别TME模式并分析配体-受体相互作用;
4)估计与感兴趣特征相关的特定突变景观;
5)相应的批量可视化和统计分析;
6)模型构建。 WES:全外显子测序。
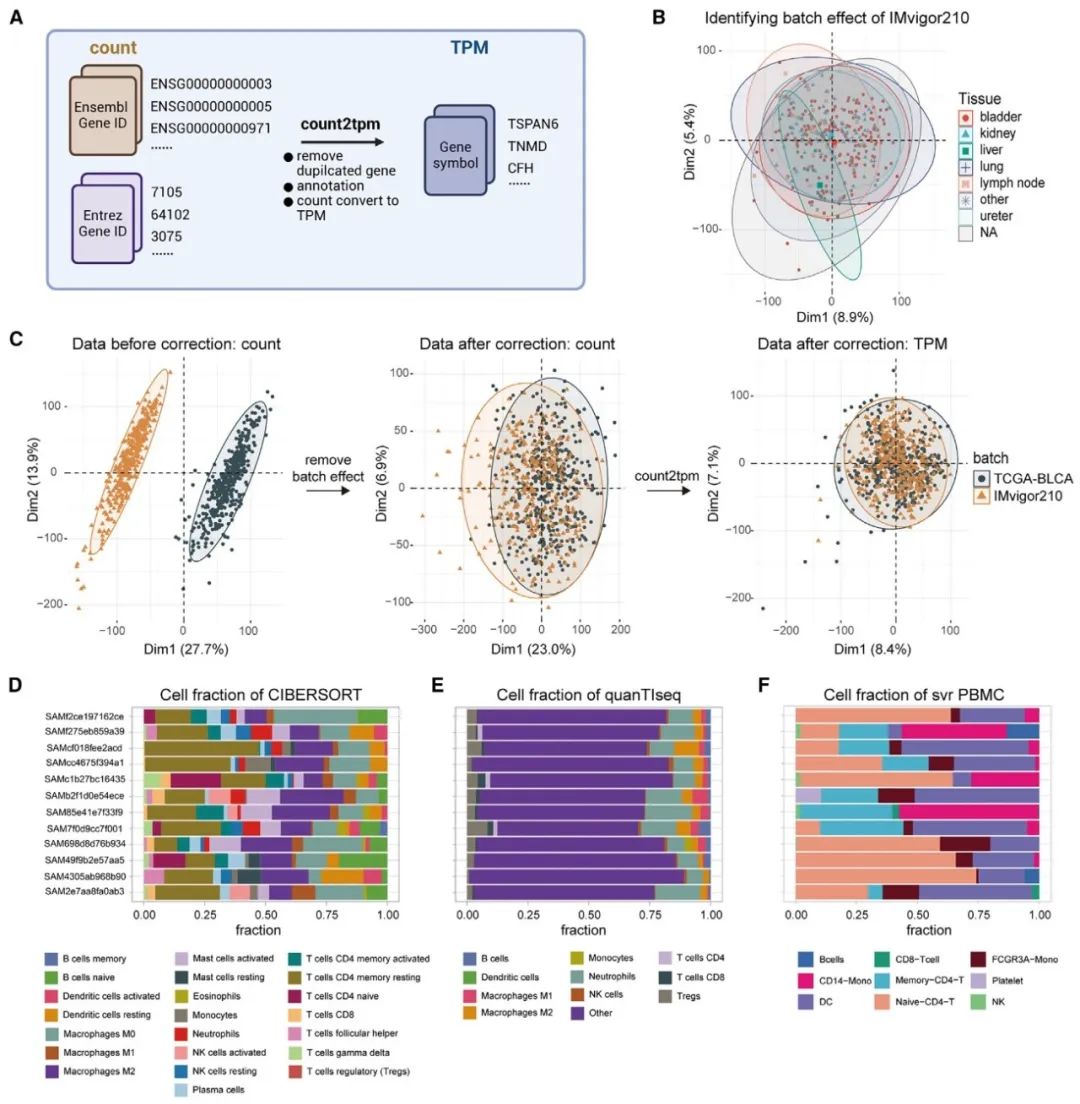
图3 转录组数据的预处理和TME细胞浸润丰度的计算
(A) 流程图展示了使用count2tpm函数将RNA-seq计数数据转换为TPM的过程。 (B) PCA散点图显示了IMvigor210数据集中组织类型的分布。 (C) IMvigor210和TCGA-BLCA数据集在批量校正前后的数据分布比较。 (D 和 E) 基于CIBERSORT (D) 和quanTIseq (E) 的TME细胞百分比条形图。 (F) IOBR 2.0利用从单细胞分析生成的细胞类型特异性基因表达特征,从批量RNA-seq数据中解码IMvigor210队列的TME景观。每种颜色代表不同的细胞类型。NA:不可用;TPM:每百万转录本。
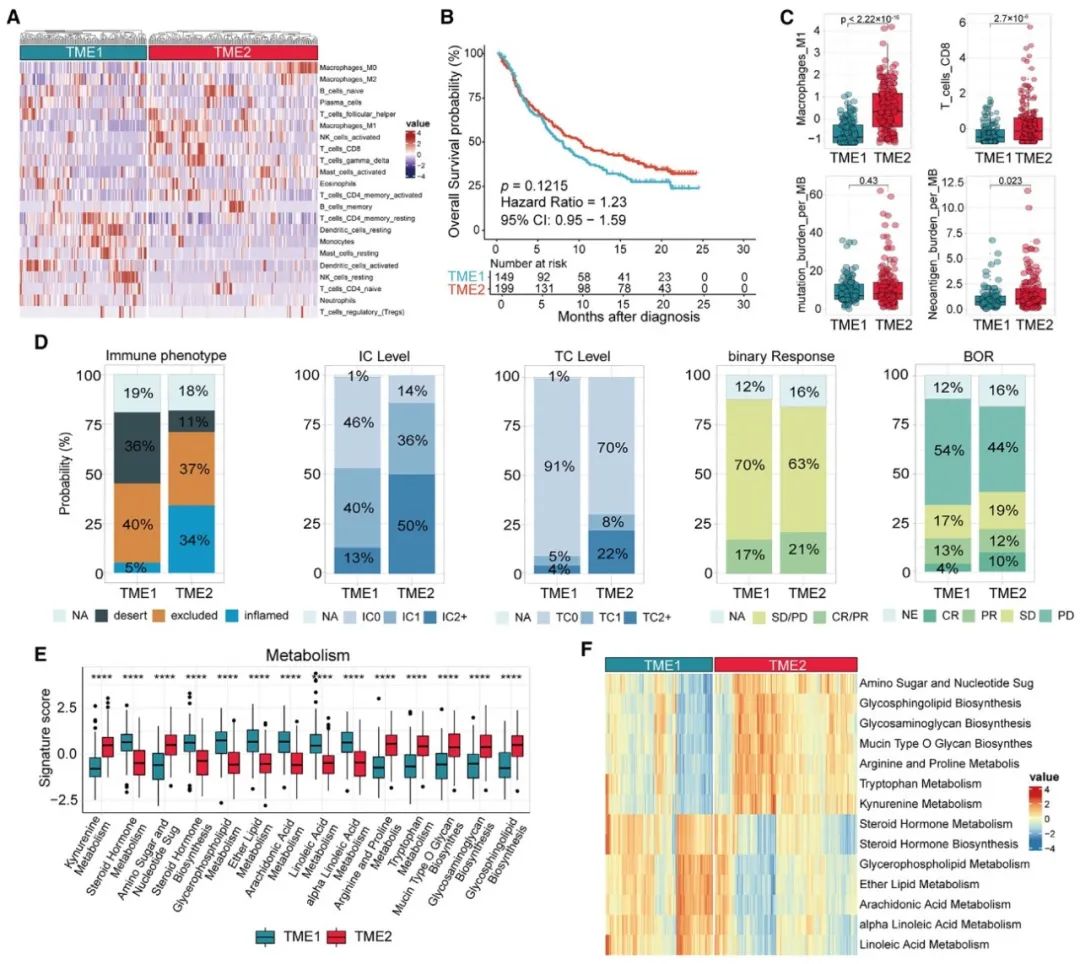
图4 IOBR 2.0识别TME模式并分析不同TME聚类之间的临床特征、表型和TME成分的差异
(A) 热图显示了高(红)和低(蓝)分组中每个患者的TME浸润细胞特征评分(蓝 = TME1;红 = TME2)。热图的行代表使用CIBERSORT算法通过deconvo_tme函数计算的TME浸润细胞特征表达(Z分数)。TME1和TME2是通过基于CIBERSORT的TME浸润细胞特征使用tme_cluster函数识别出的不同TME模式。 (B) Kaplan-Meier曲线比较了TME1和TME2之间的总生存期(OS)。 (C) 箱线图显示了TME1和TME2之间M1巨噬细胞和CD8+ T细胞(Z分数)、每兆碱基(MB)突变负荷和每MB新抗原负荷的表达。 (D) 条形图显示了临床特征和生物标志物,包括免疫表型、IC水平、TC水平以及TME1和TME2之间的BOR。 (E 和 F) 箱线图 (E) 和热图 (F) 描述了IOBR 2.0中包含的代谢特征,并识别与TME模式相关的特征。图 (E) 中的p值使用双侧Mann-Whitney U检验计算。∗p < 0.05;∗∗p < 0.01;∗∗∗p < 0.001;∗∗∗∗p < 0.0001;ns,与同型对照组相比无显著差异。 IC水平:免疫细胞水平;TC水平:肿瘤细胞水平;NA:不可用;NE:无法评估;CR:完全缓解;PR:部分缓解;SD:稳定疾病;PD:进展性疾病;BOR:最佳响应。
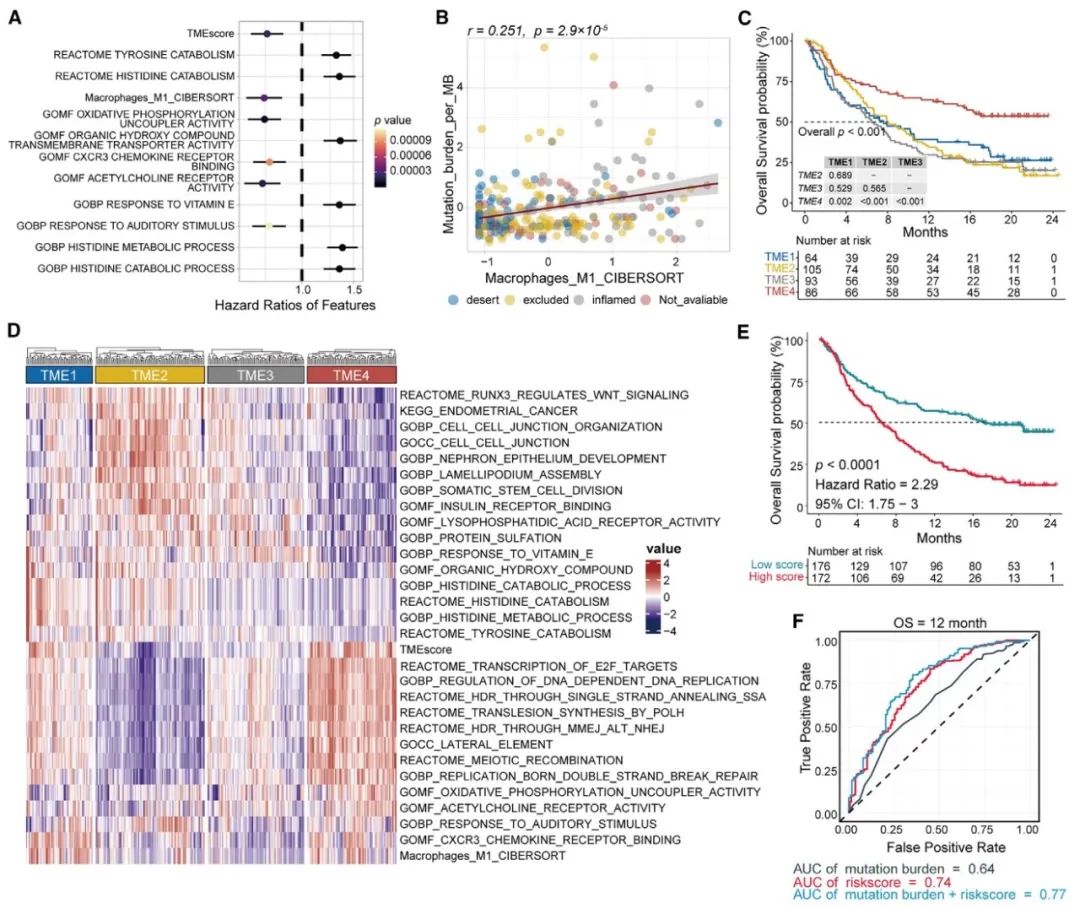
图5 免疫治疗预后特征的筛选和疗效预测模型的构建
(A) 森林图显示了IMvigor210队列中与OS相关的12个基因特征。 (B) 相关性图反映了M1巨噬细胞与每MB突变负荷之间的Spearman相关性。 (C) Kaplan-Meier曲线比较了4个TME聚类(TME1-4)之间的OS。IOBR 2.0基于与免疫治疗预后密切相关的30个特征识别出这4个TME聚类。 (D) 通过IOBR 2.0从ssGSEA获得的4个TME聚类的免疫治疗预后相关特征评分的热图。 (E) Kaplan-Meier曲线比较了高和低分组之间的OS。风险评分的截断值设为平均值。 (F) 时间依赖性的ROC曲线和AUC,用于评估风险评分(红)、每MB突变负荷(灰)以及联合模型(蓝)在12个月时对患者临床获益的预测能力。联合模型是使用Cox回归方法整合风险评分和每MB突变负荷构建的。 ROC:接收者操作特征;ssGSEA:单样本基因集富集分析;OS:总生存期。
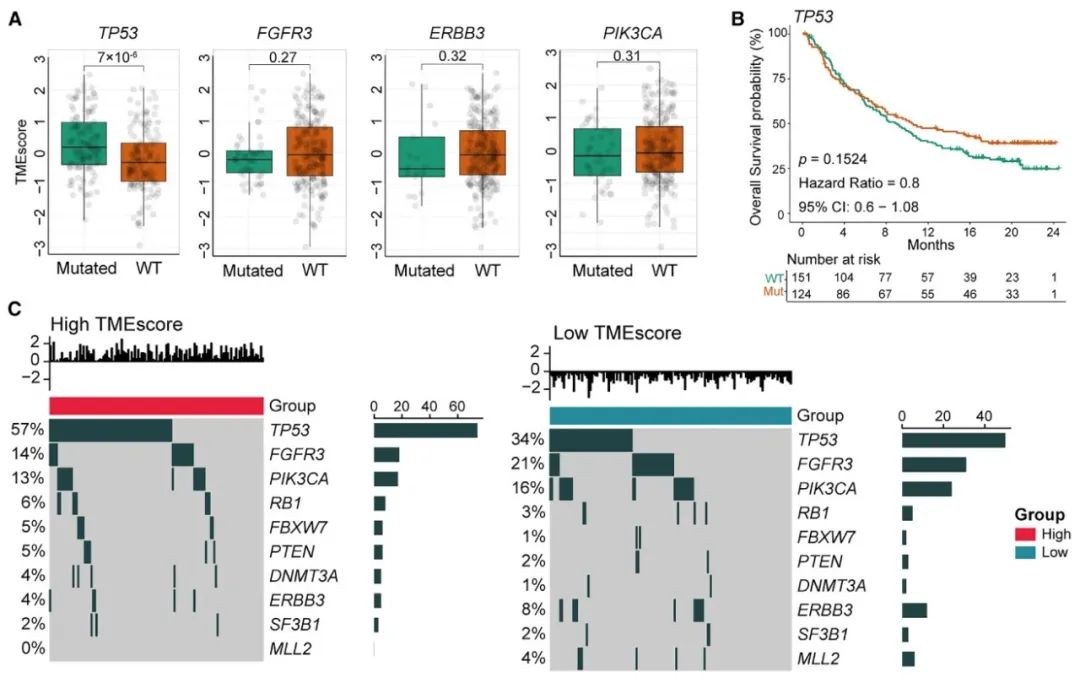
图6 基因组与TME相互作用模块描绘了与TMEscore相关的突变及其对应的肿瘤图谱
(A) 箱线图显示了与TMEscore显著相关的突变,包括TP53、FGFR3、ERBB3和PIK3CA。蓝和黄分别代表突变型和野生型状态。 (B) Kaplan-Meier曲线比较了TP53突变型和野生型状态之间的OS。 (C) 肿瘤图谱展示了在高和低TMEscore背景下的基因组改变景观。左侧绿色条形图和右侧的数字共同展示了每个基因的突变频率。WT:野生型;Mut:突变型。
参考
Cell Rep Methods. 2024 Nov 26:100910. doi: 10.1016/j.crmeth.2024.100910
注:AI辅助创作,如有错误欢迎指出。内容仅供参考,不构成任何建议。
























 1641
1641

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








