用cat12做SBM的皮层分析,这里以指标皮层厚度举例
虽然可以在segment的时候输入已知脑图谱计算脑区的皮层厚度均值,但是由于我们发现的脑区很少刚好对应脑图谱,那么我们想要获得我们分析结果的差异脑区就需要用其他的方法进行进一步分析
用到的工具为spm12,cat12
后面针对提取的顶点值用于绘画的软件为ENIGMA TOOLBOX — ENIGMA TOOLBOX documentation (enigma-toolbox.readthedocs.io),软件没有做可视化,大家学习根据它介绍的代码一一运行就行了,不难的,有耐性即可成功
1.用cat12将得到的差异脑区转为mat文件(此处需要gii文件和dat文件在同一个文件夹才能提取成果哈)
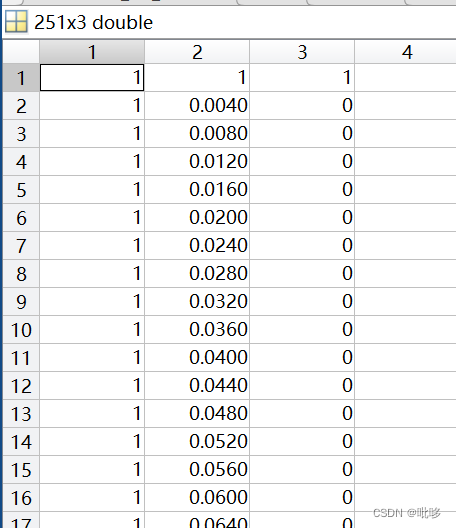
2.打开mat文件得到可以发现Y值就是每个顶点的皮层厚度数值(隐藏的是我后期提取数值的结构体,并非cat12转的mat文件),27是我提取了27个gii文件,也就是27个被试
那么为什么标准模板的顶点坐标是64984,而数值只有59380呢?可以用gifti()函数加载我们得到的统计后的spm_F.gii文件,再find就会发现其实这个统计文件也只有59380个顶点有数值,其他均为零值,而且查看途中ind的结构体就会发现,里面的数值已经对应了64984顶点的值,这是表示每个数值分别位于标准空间的具体哪个顶点

3.知道了每个顶点的数值以后,就需计算我们自己mask的每个顶点的位置了,然后将mask的位置对应到ind相同的位置,并得到59380的位置,因为数值是用59380的保存的,因此需要将位置信息转换来读取每个数值具体代码参考下方
%获取个人mask中的每个顶点的皮层厚度值
load('thickness_HC_Data.mat')%加载从cat12中得到的mat文件
mask_gii=gifti('F:\2024_04_134\statistical_91\SMRI\thickness\F_98_COV\thickness_fdr_005.gii')%读取mask的文件
mask_index=find(mask_gii.cdata')%读取mask不为零的顶点的位置
%循环得到的每个位置
for i=1:length(mask_index)%循环所有位置的总数
cur_vertices_index = find(ind==mask_index(i));%找到ind中与mask同样的数值,并输出在59380的位置信息
thickness_roi_1038(1:27,i)=Y(:,cur_vertices_index);%将得到的mask中每个顶点的皮层厚度输入到新的结构体中
end4.接下来将得到的每个被试的thickness_roi_1038进行循环求平均值,得到一组被试的每个人的平均值后就可以正常做统计了,放到spss里面也可以,数据我算过,都符合方差齐性和正态分布,应该是我们处理数据的流程里有做相应的变换,比如高斯平滑活着z变换,不过cat12里我只自己做了平滑,以前做功能MRI的时候回提供z变换和没有z变换的数值
我提取均值是为了体现我的组别的一个具体差异,画均值柱状图,体现差异,因为纯脑图只能体现区域和是否过矫正,因此用这个补充它无法体现的差异,这个就可以理解为有梯度效应啦
5.接下来就是将我们得到的差异脑区体现在大脑模板上啦,因为gii的文件像常用的基于spm的xjview和brainnet view都无法读取该格式,所以后来我就发现了本文开始的那个基于matlab的工具包——enigma toolbox,真心好用,只要稍微对代码有点了解,画一个小时运行一下就会了。其实以前尝试用过dpabisurf画过cat12的结果图都无法识别,因为两者gii文件所对应的标准空间不一样,但是enigma提供两个标准空间(fsa5和conte69)的文件绘制,因此我选择了enigma,我自己的代码在下面供参考,建议还是跟着网站运行一遍
mask_gii=gifti('F:\2024_04_134\statistical_91\SMRI\thickness\F_98_COV\thickness_fdr_005.gii')%和之前一样读取自己的差异值,注意不是模板哦,是自己的有数值的差异脑区值
curdata=mask_gii.cdata;%提取结构体的cdata
f = figure,%这一不就是画图啦,[0,1]是指colorbar数值的范围,然后不是conte69的标准空间就改为fsa5就行了
plot_cortical(curdata, 'surface_name', 'conte69', 'color_range', ...
[0 1], 'cmap', 'RdBu_r')
6.得到的图片用colormap调色就行了,这个网上搜一下就可以了,下面放一下我的成品,是不是达到了brainnet view的效果啊,这个是蒋田仔模板中的两个脑区,这只是一个面,它会提供四个面的,嘿嘿,开心

调色的话,大家把第一行设置为【1,1,1】就可以得到其余非差异脑区的为白色的底图,差异脑区颜色随意调成自己想要的就行,最后colormap一下到自己的图就行了
有错误的话希望大家帮我指正一下,一起学习,加油哈!























 6005
6005











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










