1. 聚类的指标
2. 聚类常见算法:kmeans聚类、dbscan聚类、层次聚类
3. 三种算法对应的流程
实际在论文中聚类的策略不一定是针对所有特征,可以针对其中几个可以解释的特征进行聚类,得到聚类后的类别,这样后续进行解释也更加符合逻辑。
聚类的流程
1. 标准化数据
2. 选择合适的算法,根据评估指标调参( )
KMeans 和层次聚类的参数是K值,选完k指标就确定
DBSCAN 的参数是 eps 和min_samples,选完他们出现k和评估指标
以及层次聚类的 linkage准则等都需要仔细调优。
除了经典的评估指标,还需要关注聚类出来每个簇对应的样本个数,避免太少没有意义。
3. 将聚类后的特征添加到原数据中
4. 原则t-sne或者pca进行2D或3D可视化
作业: 对心脏病数据集进行聚类。
KMeans 聚类
# 先运行之前预处理好的代码
import pandas as pd
import pandas as pd #用于数据处理和分析,可处理表格数据。
import numpy as np #用于数值计算,提供了高效的数组操作。
import matplotlib.pyplot as plt #用于绘制各种类型的图表
import seaborn as sns #基于matplotlib的高级绘图库,能绘制更美观的统计图形。
import warnings
warnings.filterwarnings("ignore")
# 设置中文字体(解决中文显示问题)
plt.rcParams['font.sans-serif'] = ['SimHei'] # Windows系统常用黑体字体
plt.rcParams['axes.unicode_minus'] = False # 正常显示负号
data = pd.read_csv('D:\python学习\学习资源(代码)\heart.csv') #读取数据
from sklearn.model_selection import train_test_split
X = data.drop(['target'], axis=1) # 特征,axis=1表示按列删除
y = data['target'] # 标签
# # 按照8:2划分训练集和测试集
# X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 80%训练集,20%测试集
import numpy as np
import pandas as pd
from sklearn.cluster import KMeans, DBSCAN, AgglomerativeClustering
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import seaborn as sns
# 标准化数据(聚类前通常需要标准化)
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# X_scaled
import numpy as np
import pandas as pd
from sklearn.cluster import KMeans
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
from sklearn.metrics import silhouette_score, calinski_harabasz_score, davies_bouldin_score
import matplotlib.pyplot as plt
import seaborn as sns
# 评估不同 k 值下的指标
k_range = range(2, 11) # 测试 k 从 2 到 10
inertia_values = []
silhouette_scores = []
ch_scores = []
db_scores = []
for k in k_range:
kmeans = KMeans(n_clusters=k, random_state=42)
kmeans_labels = kmeans.fit_predict(X_scaled)
inertia_values.append(kmeans.inertia_) # 惯性(肘部法则)
silhouette = silhouette_score(X_scaled, kmeans_labels) # 轮廓系数
silhouette_scores.append(silhouette)
ch = calinski_harabasz_score(X_scaled, kmeans_labels) # CH 指数
ch_scores.append(ch)
db = davies_bouldin_score(X_scaled, kmeans_labels) # DB 指数
db_scores.append(db)
print(f"k={k}, 惯性: {kmeans.inertia_:.2f}, 轮廓系数: {silhouette:.3f}, CH 指数: {ch:.2f}, DB 指数: {db:.3f}")
# 绘制评估指标图
plt.figure(figsize=(15, 10))
# 肘部法则图(Inertia)
plt.subplot(2, 2, 1)
plt.plot(k_range, inertia_values, marker='o')
plt.title('肘部法则确定最优聚类数 k(惯性,越小越好)')
plt.xlabel('聚类数 (k)')
plt.ylabel('惯性')
plt.grid(True)
# 轮廓系数图
plt.subplot(2, 2, 2)
plt.plot(k_range, silhouette_scores, marker='o', color='orange')
plt.title('轮廓系数确定最优聚类数 k(越大越好)')
plt.xlabel('聚类数 (k)')
plt.ylabel('轮廓系数')
plt.grid(True)
# CH 指数图
plt.subplot(2, 2, 3)
plt.plot(k_range, ch_scores, marker='o', color='green')
plt.title('Calinski-Harabasz 指数确定最优聚类数 k(越大越好)')
plt.xlabel('聚类数 (k)')
plt.ylabel('CH 指数')
plt.grid(True)
# DB 指数图
plt.subplot(2, 2, 4)
plt.plot(k_range, db_scores, marker='o', color='red')
plt.title('Davies-Bouldin 指数确定最优聚类数 k(越小越好)')
plt.xlabel('聚类数 (k)')
plt.ylabel('DB 指数')
plt.grid(True)
plt.tight_layout()
print(plt.show())

1. 肘部法则图: 找下降速率变慢的拐点,这里都差不多
2. 轮廓系数图:找局部最高点,这里选4
3. CH指数图: 找局部最高点,这里选7之前的都还行
4. DB指数图:找局部最低点,这里选2 4 7 8 9 10都行
综上,选择4比较合适。
- 为什么选择局部最优的点,因为比如簇间差异,分得越细越好,但是k太细了没价值,所以要有取舍。
# 提示用户选择 k 值
selected_k = 4
# 使用选择的 k 值进行 KMeans 聚类
kmeans = KMeans(n_clusters=selected_k, random_state=42)
kmeans_labels = kmeans.fit_predict(X_scaled)
X['KMeans_Cluster'] = kmeans_labels
# 使用 PCA 降维到 2D 进行可视化
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_scaled)
# KMeans 聚类结果可视化
plt.figure(figsize=(6, 5))
sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=kmeans_labels, palette='viridis')
plt.title(f'KMeans Clustering with k={selected_k} (PCA Visualization)')
plt.xlabel('PCA Component 1')
plt.ylabel('PCA Component 2')
plt.show()
# 打印 KMeans 聚类标签的前几行
print(f"KMeans Cluster labels (k={selected_k}) added to X:")
print(X[['KMeans_Cluster']].value_counts())

DBSCAN聚类
import numpy as np
import pandas as pd
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
from sklearn.metrics import silhouette_score, calinski_harabasz_score, davies_bouldin_score
import matplotlib.pyplot as plt
import seaborn as sns
# 评估不同 eps 和 min_samples 下的指标
# eps这个参数表示邻域的半径,min_samples表示一个点被认为是核心点所需的最小样本数。
# min_samples这个参数表示一个核心点所需的最小样本数。
eps_range = np.arange(0.1, 1.0, 0.1)
min_samples_range = range(1, 5)
results = []
for eps in eps_range:
for min_samples in min_samples_range:
dbscan = DBSCAN(eps=eps, min_samples=min_samples)
dbscan_labels = dbscan.fit_predict(X_scaled)
# 计算簇的数量(排除噪声点 -1)
n_clusters = len(np.unique(dbscan_labels)) - (1 if -1 in dbscan_labels else 0)
# 计算噪声点数量
n_noise = list(dbscan_labels).count(-1)
# 只有当簇数量大于 1 且有有效簇时才计算评估指标
if n_clusters > 1:
# 排除噪声点后计算评估指标
mask = dbscan_labels != -1
if mask.sum() > 0: # 确保有非噪声点
silhouette = silhouette_score(X_scaled[mask], dbscan_labels[mask])
ch = calinski_harabasz_score(X_scaled[mask], dbscan_labels[mask])
db = davies_bouldin_score(X_scaled[mask], dbscan_labels[mask])
results.append({
'eps': eps,
'min_samples': min_samples,
'n_clusters': n_clusters,
'n_noise': n_noise,
'silhouette': silhouette,
'ch_score': ch,
'db_score': db
})
print(f"eps={eps:.1f}, min_samples={min_samples}, 簇数: {n_clusters}, 噪声点: {n_noise}, "
f"轮廓系数: {silhouette:.3f}, CH 指数: {ch:.2f}, DB 指数: {db:.3f}")
else:
print(f"eps={eps:.1f}, min_samples={min_samples}, 簇数: {n_clusters}, 噪声点: {n_noise}, 无法计算评估指标")
# 将结果转为 DataFrame 以便可视化和选择参数
results_df = pd.DataFrame(results)
# 绘制评估指标图,增加点论文中的工作量
plt.figure(figsize=(15, 10))
# 轮廓系数图
plt.subplot(2, 2, 1)
for min_samples in min_samples_range:
subset = results_df[results_df['min_samples'] == min_samples] #
plt.plot(subset['eps'], subset['silhouette'], marker='o', label=f'min_samples={min_samples}')
plt.title('轮廓系数确定最优参数(越大越好)')
plt.xlabel('eps')
plt.ylabel('轮廓系数')
plt.legend()
plt.grid(True)
# CH 指数图
plt.subplot(2, 2, 2)
for min_samples in min_samples_range:
subset = results_df[results_df['min_samples'] == min_samples]
plt.plot(subset['eps'], subset['ch_score'], marker='o', label=f'min_samples={min_samples}')
plt.title('Calinski-Harabasz 指数确定最优参数(越大越好)')
plt.xlabel('eps')
plt.ylabel('CH 指数')
plt.legend()
plt.grid(True)
# DB 指数图
plt.subplot(2, 2, 3)
for min_samples in min_samples_range:
subset = results_df[results_df['min_samples'] == min_samples]
plt.plot(subset['eps'], subset['db_score'], marker='o', label=f'min_samples={min_samples}')
plt.title('Davies-Bouldin 指数确定最优参数(越小越好)')
plt.xlabel('eps')
plt.ylabel('DB 指数')
plt.legend()
plt.grid(True)
# 簇数量图
plt.subplot(2, 2, 4)
for min_samples in min_samples_range:
subset = results_df[results_df['min_samples'] == min_samples]
plt.plot(subset['eps'], subset['n_clusters'], marker='o', label=f'min_samples={min_samples}')
plt.title('簇数量变化')
plt.xlabel('eps')
plt.ylabel('簇数量')
plt.legend()
plt.grid(True)
plt.tight_layout()
print(plt.show())

# 选择 eps 和 min_samples 值(根据图表选择最佳参数)
selected_eps = 0.6 # 根据图表调整
selected_min_samples = 2 # 根据图表调整
# 使用选择的参数进行 DBSCAN 聚类
dbscan = DBSCAN(eps=selected_eps, min_samples=selected_min_samples)
dbscan_labels = dbscan.fit_predict(X_scaled)
X['DBSCAN_Cluster'] = dbscan_labels
# 使用 PCA 降维到 2D 进行可视化
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_scaled)
# DBSCAN 聚类结果可视化
plt.figure(figsize=(6, 5))
sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=dbscan_labels, palette='viridis')
plt.title(f'DBSCAN Clustering with eps={selected_eps}, min_samples={selected_min_samples} (PCA Visualization)')
plt.xlabel('PCA Component 1')
plt.ylabel('PCA Component 2')
plt.show()
# 打印 DBSCAN 聚类标签的分布
print(f"DBSCAN Cluster labels (eps={selected_eps}, min_samples={selected_min_samples}) added to X:")
print(X[['DBSCAN_Cluster']].value_counts())
层次聚类
import numpy as np
import pandas as pd
from sklearn.cluster import AgglomerativeClustering
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
from sklearn.metrics import silhouette_score, calinski_harabasz_score, davies_bouldin_score
import matplotlib.pyplot as plt
import seaborn as sns
# 标准化数据
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 评估不同 n_clusters 下的指标
n_clusters_range = range(2, 11) # 测试簇数量从 2 到 10
silhouette_scores = []
ch_scores = []
db_scores = []
for n_clusters in n_clusters_range:
agglo = AgglomerativeClustering(n_clusters=n_clusters, linkage='ward') # 使用 Ward 准则合并簇
agglo_labels = agglo.fit_predict(X_scaled)
# 计算评估指标
silhouette = silhouette_score(X_scaled, agglo_labels)
ch = calinski_harabasz_score(X_scaled, agglo_labels)
db = davies_bouldin_score(X_scaled, agglo_labels)
silhouette_scores.append(silhouette)
ch_scores.append(ch)
db_scores.append(db)
print(f"n_clusters={n_clusters}, 轮廓系数: {silhouette:.3f}, CH 指数: {ch:.2f}, DB 指数: {db:.3f}")
# 绘制评估指标图
plt.figure(figsize=(15, 5))
# 轮廓系数图
plt.subplot(1, 3, 1)
plt.plot(n_clusters_range, silhouette_scores, marker='o')
plt.title('轮廓系数确定最优簇数(越大越好)')
plt.xlabel('簇数量 (n_clusters)')
plt.ylabel('轮廓系数')
plt.grid(True)
# CH 指数图
plt.subplot(1, 3, 2)
plt.plot(n_clusters_range, ch_scores, marker='o')
plt.title('Calinski-Harabasz 指数确定最优簇数(越大越好)')
plt.xlabel('簇数量 (n_clusters)')
plt.ylabel('CH 指数')
plt.grid(True)
# DB 指数图
plt.subplot(1, 3, 3)
plt.plot(n_clusters_range, db_scores, marker='o')
plt.title('Davies-Bouldin 指数确定最优簇数(越小越好)')
plt.xlabel('簇数量 (n_clusters)')
plt.ylabel('DB 指数')
plt.grid(True)
plt.tight_layout()
print(plt.show())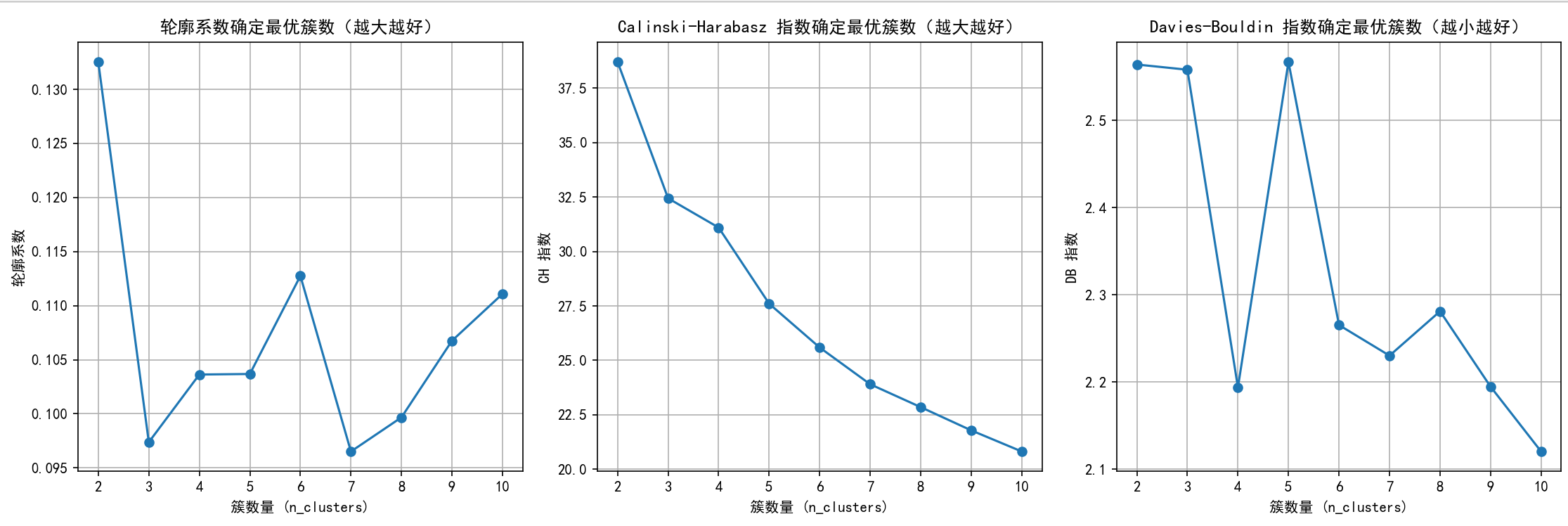
# 提示用户选择 n_clusters 值(这里可以根据图表选择最佳簇数)
selected_n_clusters = 10 # 示例值,根据图表调整
# 使用选择的簇数进行 Agglomerative Clustering 聚类
agglo = AgglomerativeClustering(n_clusters=selected_n_clusters, linkage='ward')
agglo_labels = agglo.fit_predict(X_scaled)
X['Agglo_Cluster'] = agglo_labels
# 使用 PCA 降维到 2D 进行可视化
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_scaled)
# Agglomerative Clustering 聚类结果可视化
plt.figure(figsize=(6, 5))
sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=agglo_labels, palette='viridis')
plt.title(f'Agglomerative Clustering with n_clusters={selected_n_clusters} (PCA Visualization)')
plt.xlabel('PCA Component 1')
plt.ylabel('PCA Component 2')
plt.show()
# 打印 Agglomerative Clustering 聚类标签的分布
print(f"Agglomerative Cluster labels (n_clusters={selected_n_clusters}) added to X:")
print(X[['Agglo_Cluster']].value_counts())
# 层次聚类的树状图可视化
from scipy.cluster import hierarchy
import matplotlib.pyplot as plt
# 假设 X_scaled 是标准化后的数据
# 计算层次聚类的链接矩阵
Z = hierarchy.linkage(X_scaled, method='ward') # 'ward' 是常用的合并准则
# 绘制树状图
plt.figure(figsize=(10, 6))
hierarchy.dendrogram(Z, truncate_mode='level', p=3) # p 控制显示的层次深度
# hierarchy.dendrogram(Z, truncate_mode='level') # 不用p这个参数,可以显示全部的深度
plt.title('Dendrogram for Agglomerative Clustering')
plt.xlabel('Cluster Size')
plt.ylabel('Distance')
print(plt.show())

1. 横坐标代表每个簇对应样本的数据,这些样本数目加一起是整个数据集的样本数目。这是从上到下进行截断,p=3显示最后3层,不用p这个参数会显示全部。
2. 纵轴代表距离 ,反映了在聚类过程中,不同样本或簇合并时的距离度量值。距离越大,意味着两个样本或簇之间的差异越大;距离越小,则差异越小。
@浙大疏锦行

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








