第一次开发类似于生物化学的程序,基于网络服务,使用JAVA语言编写,昨天总算初步完成,我在国内看类似的东西好像不多,我贴几个我程序图片,欢迎同行点评。
主窗口
加载要查询的基因列表
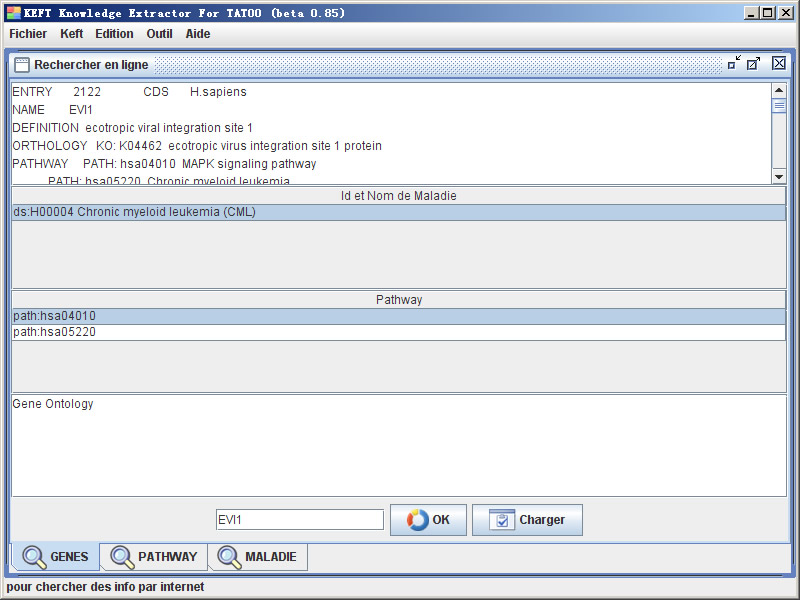
基因查询结果
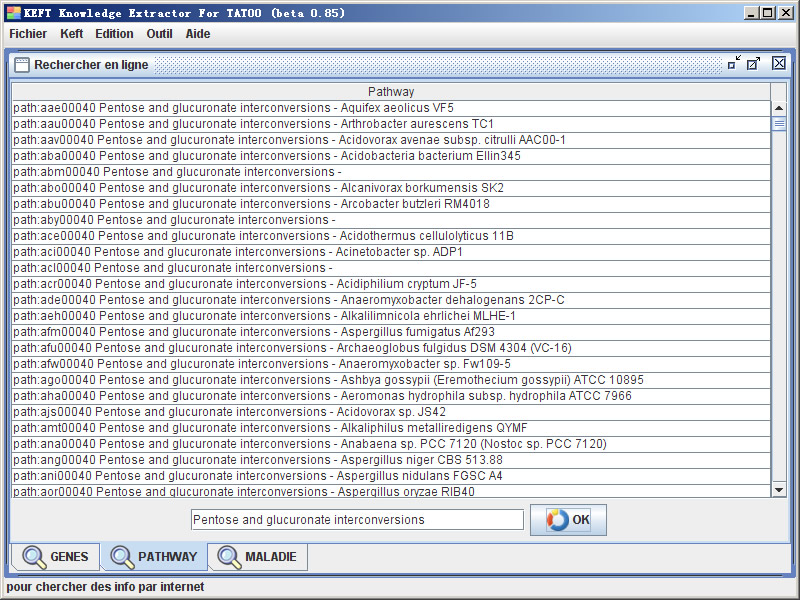
路径搜索结果
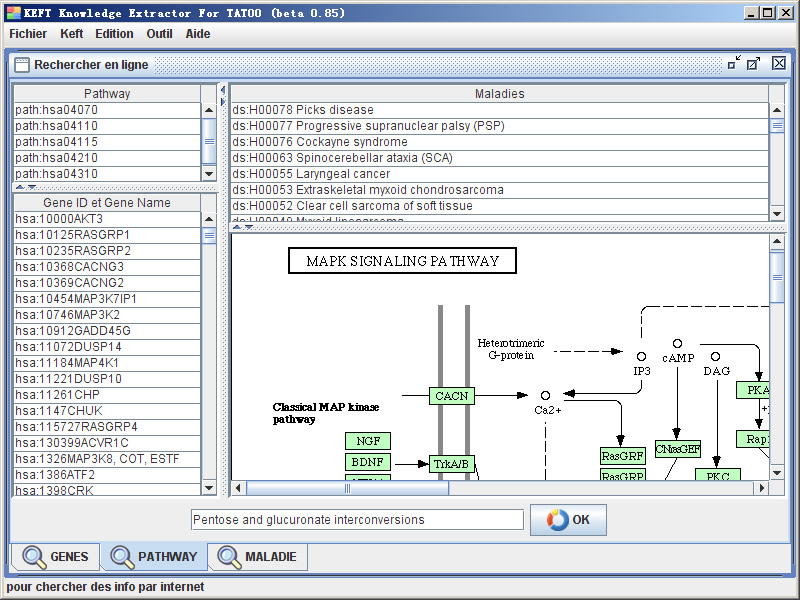
路径搜索结果
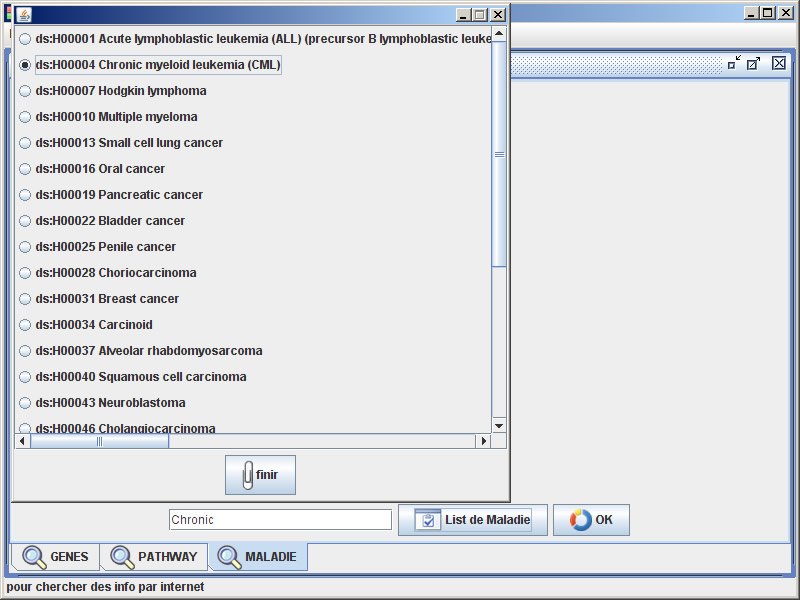
疾病搜索引擎
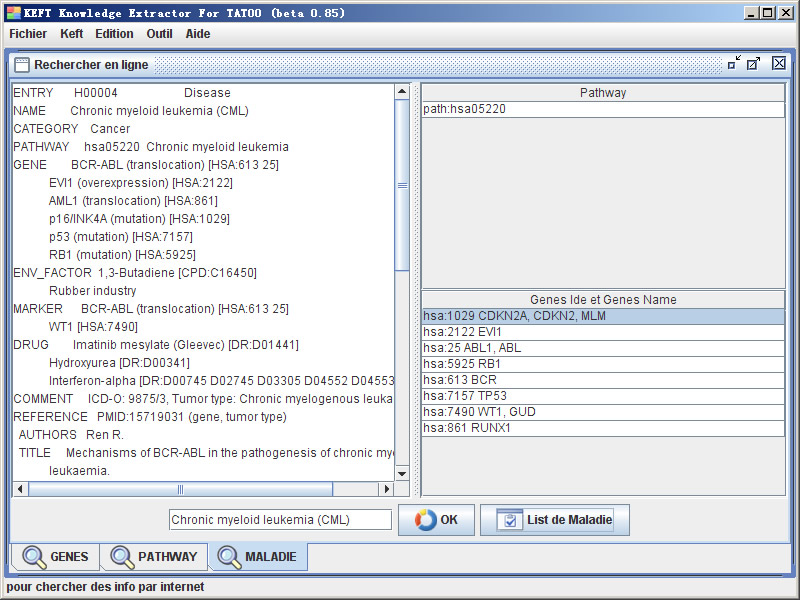
疾病搜索结果展示
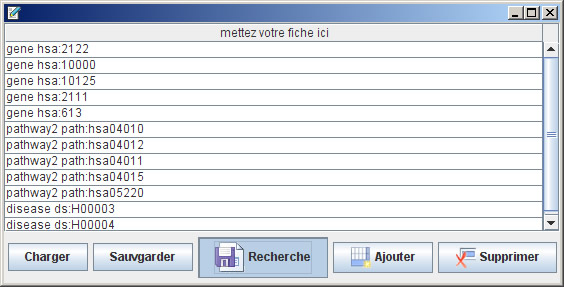
编辑器
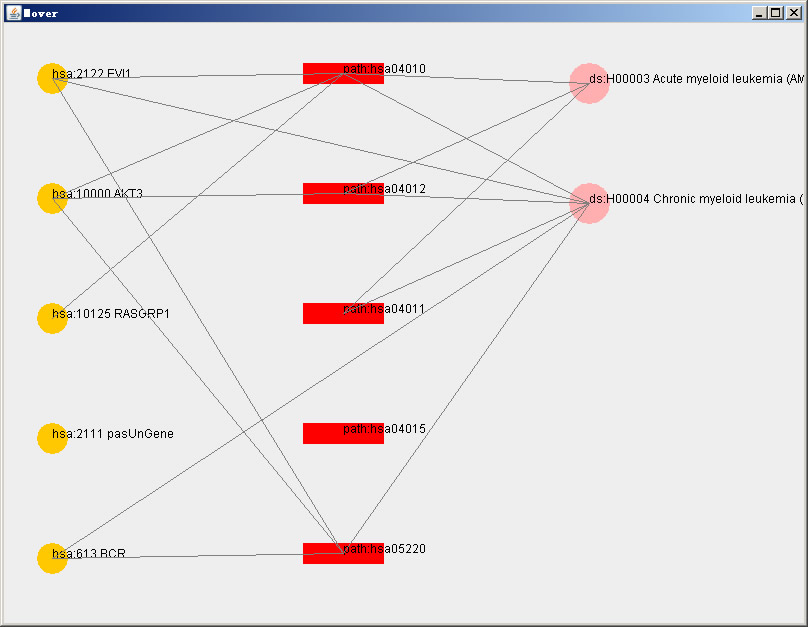
图像表示深层关系查找,每一个元素都可以移动。
第一次开发类似于生物化学的程序,基于网络服务,使用JAVA语言编写,昨天总算初步完成,我在国内看类似的东西好像不多,我贴几个我程序图片,欢迎同行点评。

主窗口
加载要查询的基因列表

基因查询结果

路径搜索结果

路径搜索结果

疾病搜索引擎

疾病搜索结果展示

编辑器

图像表示深层关系查找,每一个元素都可以移动。
 639
639
 601
601
 1031
1031
 722
722

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?


