书接上回:R语言下载TCGA数据 非R包
我们已经获取到TCGA单样品数据,接下来就是合并和处理合并后重复基因名问题。
01.合并前准备
library(rjson)
library(dplyr)
#从原始数据中把*.tsv文件复制到data_in_one文件夹中,便于后续操作
if(!file.exists("data_in_one")) dir.create("data_in_one")
for (dirname in dir("rawdata/")){
mydir <- paste0(getwd(),"/rawdata/",dirname)#注意这里和上书提到的位置一致
file <- list.files(mydir,pattern = "_counts")
myfile <- paste0(mydir,"/",file)
file.copy(myfile,"data_in_one")
}
02.获取TCGA样品编号
#读取json文件,提取样品TCGA编号名称
metadata <- jsonlite::fromJSON("metadata.cart.2023-11-22.json")
metadata_id <- metadata %>%
dplyr::select(c(file_name,associated_entities))
naid_df <- data.frame()
for (i in 1:nrow(metadata)){
naid_df[i,1] <- metadata_id$file_name[i]
naid_df[i,2] <- metadata_id$associated_entities[i][[1]]$entity_submitter_id
}
colnames(naid_df) <- c("filename","TCGA_id")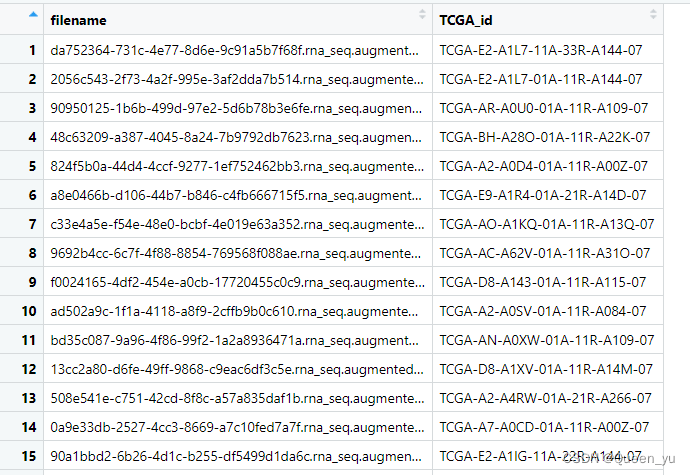
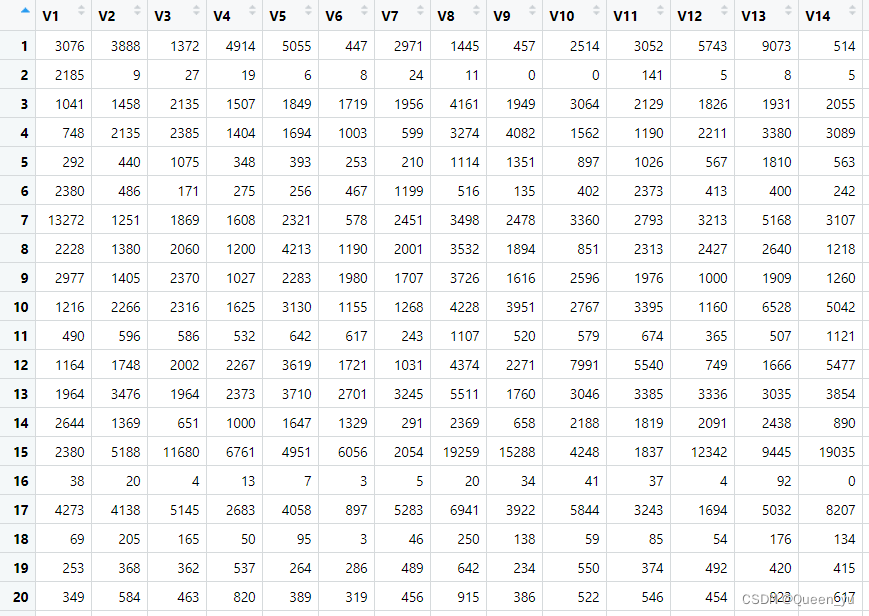
03.获取不同矩阵类型
#提取你想要的类型 counts或者fpkm值
myfread <- function(files){
data = data.table::fread(paste0("data_in_one/",files))
data = data[-seq(1,4),]
data = data$fpkm_unstranded#博主这边是fpkm值,想获取counts矩阵$后边换成unstranded
}
files <- dir("data_in_one")
system.time(f <- lapply(files,myfread))
expr_df <- as.data.frame(do.call(cbind,f))
#查看expr_df,发现我们的矩阵已经获取成功了
04.添加样品TCG编号和基因名
#但是发现样品名称和基因symbol没有,需要进一步处理
#先获取样品名称
rownames(naid_df) <- naid_df[,1]
naid_df <- naid_df[files,]
colnames(expr_df) <- naid_df$TCGA_id
#在获取基因名称
gene_id <- data.table::fread(paste0("data_in_one/",files[1]))$gene_name#这里是基因symbol如果想看到ENS开头的名称,$gene_id就可
gene_id <- gene_id[-seq(1,4)]
expr_df <- cbind(gene_id=gene_id,expr_df)
#结果是这样的基因symbol结果

#基因ENS结果是这样的
05.去除重复基因名
#如果做的是ENSG下边就不用看了,输出结果就行,如果是symbol需要处理一下重复基因名,可参考博主 R去除重复基因名博客
matrix0 <- order(rowMeans(expr_df[,-1]),decreasing = T)
expr_ordered=expr_df[matrix0,]
keep=!duplicated(expr_ordered$gene_id)
expr_max=expr_ordered[keep,]
06.输出
#最终结果输出
expr_max=as.matrix(expr_max)
rownames(expr_max) <- expr_max[,1]
exp=expr_max[,2:ncol(expr_max)]
dimnames=list(rownames(exp),colnames(exp))
data=matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
lea=rbind(id=colnames(data),data)
write.table(lea,file = "TCGA.txt",sep = "\t",quote = F,col.names = F)
到这里矩阵中无重复基因名,用于接下来分析,今天的分享就到这里啦,博主抽空再把获取临床数据,SNP数据和CNV数据获取更新一下,至于时间嘛就待定了,欲知后事如何,请待下回分享~






















 3517
3517











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








