Dayu Tan, Cheng Yang, Jing Wang, Yansen Su, Chunhou Zheng, scAMAC: self-supervised clustering of scRNA-seq data based on adaptive multi-scale autoencoder, Briefings in Bioinformatics, Volume 25, Issue 2, March 2024, bbae068, scAMAC: self-supervised clustering of scRNA-seq data based on adaptive multi-scale autoencoder | Briefings in Bioinformatics | Oxford Academic
论文地址:
论文代码:
摘要
细胞聚类对于分析单细胞RNA测序(scRNA-seq)数据以理解高层次的生物学过程至关重要。基于深度学习的聚类方法近年来在scRNA-seq数据分析中被广泛应用。然而,现有的深度模型往往忽视了网络层之间的相互连接和交互,导致网络层内结构信息的丢失。为此,作者开发了一种基于自适应多尺度自动编码器的新型自监督聚类方法,称为scAMAC。自监督聚类网络利用多尺度注意力机制融合多尺度自动编码器的编码器、隐藏层和解码层的特征信息,这使得能够在同一尺度内探索细胞相关性,并捕捉跨不同尺度的深度特征。自监督聚类网络使用融合后的潜在特征计算成员矩阵,并基于该成员矩阵优化聚类网络。scAMAC采用自适应反馈机制来监督多尺度自动编码器的参数更新,从而获得更有效的细胞特征表示。scAMAC不仅能够实现细胞聚类,还能通过解码层进行数据重建。通过大量实验,证明了scAMAC在数据聚类和重建方面优于多个先进的聚类和插补方法。此外,scAMAC对下游分析,如细胞轨迹推断,也具有重要意义。
方法
数据预处理
收集了14个常用的公共数据集,并移除具有不清晰细胞身份的细胞,以减少未知标签对实验分析公平性的影响。这些数据集的详细信息如表1所示,均可免费下载(网址:https://hemberg-lab.github.io/scRNA.seq.datasets/)。
作者使用Scanpy软件包[32]对真实的scRNA-seq数据进行预处理。scRNA-seq数据是一个二维矩阵,行表示细胞,列表示基因。针对这些数据集,移除在超过95%的细胞中表达值为0的基因,对数据进行归一化和对数转换,然后选择前3000个高变异基因作为输入数据。

scAMAC 模型
scAMAC模型主要由两部分组成:去噪深层多尺度自编码器和自监督聚类网络。多尺度自编码器用于获取原始输入数据的低维表示和重构数据。自监督聚类网络利用MSA模块( Multi-Scale Attention )融合自编码器各层的输出结果,实现不同层次信息的整合。这一融合过程有助于探索细胞之间的关系,减少重要数据特征的损失,从而增强细胞聚类性能。
此外,该网络整合了自监督机制,在训练过程中发挥双重作用。一方面,它引导多尺度自编码器的训练,从输入数据中提取有意义的特征。另一方面,通过根据自监督学习信号迭代更新网络参数,优化整体模型。

如图所示,该模型将基因表达矩阵X作为输入。首先,对预处理数据添加均匀噪声,然后送入自编码器进行训练,以增强网络的鲁棒性。每个网络层的输出为Za、Zb和Zc。通过两个全连接层,Za和Zc分别转换为Za’和Zc’,它们与Zb具有相同的维度。然后,Za’和Zc’和Zb传递给自监督聚类模块。自监督聚类模块利用MSA机制捕捉细胞之间的关系,以及自编码器各层的贡献,获取Z。基于Z计算出成员矩阵U,并通过U进行优化。为了在网络内部实现自监督过程,使用成员矩阵U构建细胞相似性矩阵,以监督自编码器的参数更新。
去噪深层多尺度自编码器
与之前几篇文章介绍的去噪自编码器不一样,去噪深层多尺度自编码器增加了多尺度信息的融合。
对于给定的单细胞RNA测序数据,其基因表达矩阵表示为X ∈ R^{V×G},其中V是细胞的数量,G是每个细胞的基因维度。该自编码器包括编码器、解码器和隐藏层,用于对基因表达数据进行编码和解码,从而获取数据的潜在特征,并通过解码器输出适当的重构数据。具体来说,在编码器中,输入含噪音的数据X0,并在编码层获得输出数据Za,计算如下:
\[ Za = \phi(w_1X_0 + b_1) \quad (1) \]
其中,φ是LeakyReLU激活函数,w1是编码层的权重矩阵,b1是编码层的偏置。这里,X0 = X + N,其中N是均匀分布的噪声。Za通过隐藏层映射为Zb,具体公式如下:
\[ Zb = \phi(w_2Z_a + b_2) \quad (2) \]
其中,φ是LeakyReLU激活函数,w2是隐藏层的权重矩阵,b2是隐藏层的偏置。然后,通过解码层获得输出数据Zc和与编码层相同维度的重构数据X',具体公式如下:
\[ Zc = \phi(w_3Z_b + b_3) \quad (3) \]
\[ X' = \phi(w_4Z_c + b_4) \quad (4) \]
在方程(3)和(4)中,φ是LeakyReLU激活函数,w3和w4是解码层的权重矩阵,b3和b4是解码层的偏置。
为了增强自编码器的训练并有效整合编码和解码层之间的信息,采用以下损失函数来优化网络:

其中,n表示细胞的数量,Xi表示细胞i的输入特征,Xi'表示细胞i的重构特征,Zai表示细胞i的编码层提取的特征,Zci表示细胞i的解码层提取的特征。
MSA mechanism
MSA机制有效地整合了多尺度信息并利用其各自的优势,主要包括两个部分:多尺度协同(MSS)模块和多尺度集成(MSI)模块。在MSA机制中,MSS模块和MSI模块协同工作,以捕捉输入特征图中的空间信息和通道交互。MSS模块负责捕捉空间信息和通道内的依赖关系,而MSI模块则负责获取通道之间的交互。MSS模块和MSI模块的结合可以提高模型的性能,并捕捉更丰富的特征信息。

MSS模块包括空间注意力模块和通道注意力模块,如图2所示。空间注意力模块用于捕捉同一尺度内细胞之间的相似性,关注它们的空间关系。此外,通道注意力模块用于探索跨不同尺度的细胞深层特征。这两个模块协同工作,共享键和查询的权重,减少了参数数量并生成更高效的特征表示。
通过结合空间和通道注意力模块,模型可以有效捕捉数据中的局部和全局依赖关系。空间注意力模块增强了模型识别空间模式和捕捉邻近细胞之间局部关联的能力。同时,通道注意力模块允许模型提取和强调跨不同尺度的最具信息量的特征,从而探索细胞的深层特征。

如图3所示,MSI模块执行一种非降维的局部跨通道交互策略,可以轻量化地捕捉网络不同层的贡献。与MSS模块中的通道注意力不同,MSI模块使用全局上下文信息计算方法来获得每个通道的权重,从而学习多尺度自编码器中每个网络层的重要性。
常规的自编码器架构可以捕捉细胞的主要特征,但可能会忽略自编码器每层中包含的丰富结构信息。为了应对这一问题,模型进一步整合了多尺度自编码器中不同网络层的输出结果,使用MSA机制来探索细胞间更深层的信息,通过融合各尺度的特征来实现。
为了促进特征融合,首先通过全连接层对解码层及其输出Za和Zc进行降维,公式如下:
\[ Z_a'= \phi(w_{11}Z_a + b_{11}) \quad (6) \]
\[ Z_c' = \phi(w_{22}Z_c + b_{22}) \quad (7) \]
在公式(6)和(7)中,φ表示LeakyReLU激活函数,w_{11}、w_{22}、b_{11}和b_{22}分别是全连接层网络的权重矩阵和偏置。
将Z_a'、Z_c'和Zb连接起来,并进行归一化:
\[ Z = Z_a' + Z_c' + Z_b \quad (8) \]
然后,使用MSS模块探索细胞的深层特征及其相互关系,公式如下:

其中,SA代表空间注意力模块,CA代表通道注意力模块。Q_{shared}、K_{shared}、V_{spatial}和V_{channel}分别表示共享查询、共享键、空间值和通道值向量矩阵。w_q、w_k、w_{vs}和w_{vc}表示四个不同初始化的全连接层的权重矩阵,b_q、b_k、b_{vs}和b_{vc}表示四个不同初始化的全连接层的偏置。
空间注意力模块SA定义如下:

空间投影的权重表示为w_p和w_s,空间投影的偏置表示为b_p和b_s。d_k的维度与潜在特征Z相同,用于防止softmax值过大,导致注意力机制的偏导数接近0。
通道注意力CA的公式如下:

对于MSS模块的输出Z_e,再次对其进行归一化,并将其传递到MSI模块,以学习自编码器中每个网络层的重要性,并获得最终的低维潜在特征Z。
自监督聚类网络
模型使用低维潜在特征 \( Z \) 在 FKM (Fuzzy k-means)算法中计算隶属矩阵 \( U \),并通过 \( U \) 优化自监督聚类网络。其损失函数如下:
\[ L_{C-N} = \sum_{i=1}^{n} \sum_{j=1}^{k} H_{ij} u_{ij} \left\| Z_i - C_j \right\|^2 \]
在公式(12)中, 是自监督聚类优化的权重。\( Z_i \) 表示模型获得的第 \( i \) 个细胞的潜在特征,\( C_j \) 是第 \( j \) 个簇的质心,\( u_{ij} \) 是第 \( i \) 个细胞在第 \( j \) 个簇中的隶属度,\( \epsilon \) 是影响自监督聚类网络鲁棒性的平衡因子。
是自监督聚类优化的权重。\( Z_i \) 表示模型获得的第 \( i \) 个细胞的潜在特征,\( C_j \) 是第 \( j \) 个簇的质心,\( u_{ij} \) 是第 \( i \) 个细胞在第 \( j \) 个簇中的隶属度,\( \epsilon \) 是影响自监督聚类网络鲁棒性的平衡因子。
簇中心 \( C_j \) 的更新公式为:
\[ C_j = \frac{\sum_{i=1}^{n} H_{ij} u_{ij} Z_i}{\sum_{i=1}^{n} H_{ij} u_{ij}} \]
隶属度 \( u_{ij} \) 的更新公式为:
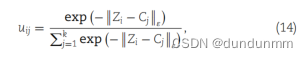

为了实现多尺度自编码器的自监督学习并将聚类信息纳入网络以改进数据重构,引入了一种自适应反馈机制。相应的自监督损失函数定义如下:

其中,\( \cdot \) 表示向量间的点积,IM 表示单位向量。![]() 表示不同细胞间的相似度分数。
表示不同细胞间的相似度分数。
实验结果

很有新意的在于没有用之前的GCN或者GAT学习细胞之间的关联,而是用MSA机制来实现。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








