原文标题:Cm-siRPred: Predicting chemically modified siRNA efficiency based on multi-view learning strategy
原文链接:https://www.sciencedirect.com/science/article/pii/S0141813024014417?via%3Dihub
摘要
siRNA修饰是指对siRNA分子进行化学或结构上的改变,以提高其在体内的稳定性和生物利用度,减少脱靶效应,并增强其特异性。这些修饰可以包括对siRNA分子的碱基、糖骨架或磷酸骨架的改变,以改善其药代动力学性质和减少免疫原性。
siRNA分子的合理修饰对于确保其药物特性至关重要。基于机器学习的化学修饰 siRNA(cm-siRNA)效率预测可以显著优化 siRNA 化学修饰的设计过程,节省 siRNA 药物开发的时间和成本。然而,现有的机器学习方法存在数据集小、数据表示能力不足和缺乏可解释性等局限性。因此,在本研究中,我们开发了基于多视角学习策略的 Cm-siRPred 算法。该算法采用多视角策略来表示 cm-siRNA 的双链序列、化学修饰和理化性质。它结合了一个交叉注意模型来全局关联不同的表示向量,以及一个双层 CNN 模块来学习局部关联特征。该算法在交叉验证实验、独立数据集和已获批准的 siRNA 药物案例研究中表现出卓越的性能,展示了其鲁棒性和泛化能力。此外,我们还开发了一个用户友好型网络服务器,可有效预测 cm-siRNA 的效率,并协助设计 siRNA 药物化学修饰。总之,Cm-siRPred 是一个实用的工具,为 siRNA 化学修饰和药物效率研究提供了宝贵的技术支持,同时有效地帮助了新型小核酸药物的开发。Cm-siRPred可在https://cellkno wledge.com.cn/sirnapredictor/免费获取。
1.引言
小干扰 RNA(small interfering RNA,siRNA)是一种由 21-23 个核苷酸组成的双链非编码 RNA,可以特异性地靶向 mRNA,通过 RNA 干扰(RNAi)使基因沉默[1,2]。这种机制使得 siRNA 可以被设计成核苷酸药物,在 mRNA 水平上靶向致病基因 [3]。与小分子药物和抗体药物相比,siRNA 具有靶向范围广、特异性强、研发周期短等多重优势,已受到医学界和工业界的广泛关注,成为分子治疗领域的重要分子[4,5]。
尽管 siRNA 疗法潜力巨大,但其生物半衰期短、易被核糖核酸酶消化、脱靶效应、免疫原性和细胞毒性等各种局限性阻碍了其发展和临床应用[6,7]。这些缺陷导致大型制药公司在 2010 年代初退出该领域,造成严重的资金危机。然而,一些公司坚持不懈,不断改进序列设计、核苷酸化学修饰和给药机制,为更安全有效的 RNAi 疗法铺平了道路[8]。2018 年,美国食品和药物管理局(FDA)批准了首个 siRNA 药物 Patisiran,用于治疗遗传性变异性转甲状腺素淀粉样变性病(hATTRv)[9]。这一里程碑不仅为 hATTRv 患者带来了希望,也重新点燃了医学界和工业界对 siRNA 治疗研究的热情。截至 2023 年 5 月,全球已有五种 siRNA 药物获得批准,还有许多 siRNA 疗法正处于不同的临床试验阶段(表 1)。
越来越多的证据表明,对 siRNA 进行化学修饰可以增强 siRNA 药物的稳定性、特异性和安全性,从而提高其治疗效率[1,11]。例如,硫代磷酸酯(PS)和甲基膦酸酯(MP)等膦酸酯修饰可以保护磷酸二酯键中脆弱的磷原子,减少磷酸基团上的负电荷,提高抗核酸酶能力、药物动力学特性和血清稳定性[12,13]。核糖修饰,如 2'-O- 甲氧基乙基(2'-OME)和 2′-氟嘧啶(2'-F)修饰,可改变核糖的分子构象,促进分子间相互作用,提高热稳定性和靶向效率[14-17]。在尿苷的 5 位进行溴或碘的碱基修饰,可加强腺嘌呤-尿苷(A-U)碱基对的连接,提高 siRNA 的 mRNA 靶向效率[18],这些化学修饰的组合可显著提高 siRNA 的临床疗效[19],Alnylam、诺华和 Ionis 等大公司都采用多种化学修饰的组合来提高其 siRNA 治疗药物的类药物特性[20-25]。
合理安排化学修饰对确保 siRNA 的药物特性至关重要[26]。药物开发者早期对预定义化学修饰模式的依赖导致了实际应用中的高失败率以及巨大的时间和经济成本[3,27]。目前,随着人工智能技术的爆炸式发展,利用机器学习方法预测化学修饰 siRNA(cm-siRNA)的效率并辅助 siRNA 化学修饰设计正成为核酸药物开发的新策略[28-31]。然而,与专注于预测未修饰 siRNA 活性的算法相比,专门针对 cm-siRNA 效率预测的研究和算法非常有限。现有的三种方法,即 Dar 等人基于支持向量机(SVM)的方法(SMEpred)[28]、Shmushkovich 等人基于线性回归的方法[29]和 Dong 等人基于偏最小二乘法回归的方法[30],均采用统计模型或特征提取方法来表示 siRNA 序列,其模型性能取决于研究者对特征的提取,因此要全面捕捉 cm-siRNA 分子的隐含特征具有挑战性[32]。此外,这些方法缺乏可解释性,没有考虑 siRNA 有义链的信息,而有义链和反义链之间的热稳定性以及与目标 mRNA 的分子亲和力都会影响沉默效率[19]。
在此,我们介绍一种基于多视角学习策略的 cmsiRNA 效率预测算法 Cm-siRPred。该算法利用多视角策略来表示正反义链序列、siRNA 的理化性质以及化学修饰的位置和分子指纹,然后利用交叉注意模型对不同的表示向量进行全局相关性分析,再利用 CNN 模块学习局部相关性特征。此外,我们还开发了一个精心设计的网络服务器,用于预测cm-siRNA的效率,并协助设计化学修饰siRNA药物。该网络服务器可在 https ://cellknowledge.com.cn/sirnapredictor/ 免费获取。
2.材料与方法
2.1.研究概述

(1)从 siRNAmod 中收集数据,并将其格式化为 fastq;
(2)使用单次编码、分子指纹和理化性质提取特征;
(3)构建共注意力多视角回归模型;
(4)通过 5 倍交叉验证、独立数据集测试和 FDA 药物数据验证对模型进行评估;
(5)开发用于 cm-siRNA 药物效率预测的网络服务器。
本研究分为 5 个不同阶段,包括 cm-siRNA 数据集的收集、特征提取、模型构建、模型评估和网络服务器的建立(图 1)。第一阶段包括从 siRNAmod 数据库中收集 cm-siRNA 数据集和化学修饰信息。然后将数据整理成 fastq 格式。接下来,使用单次编码、分子指纹和理化性质提取每条 cm-siRNA 的分子表征。在对数据进行清理和充分表征后,开发了一个基于共注意力的多视角回归模型来预测 cm-siRNA 的效率。该模型采用多种方法进行了评估,包括 5 倍交叉验证、独立数据集验证以及与美国食品药物管理局批准的临床药物进行验证。为了便于进一步研究 cm-siRNA 药物,我们为科学界开发了一个用户友好型网络服务器。
2.2.数据集收集
首先,我们从 siRNAmod 数据库下载 siRNA,并进行预处理以去除低质量和未修改的 siRNA[33]。这样就得到了总共 4278 个 cm-siRNA 条目。该数据集涵盖了多种化学修饰,包括 128 种不同类型,其中有 8 种糖类磷酸连接修饰、77 种糖类修饰、3 种磷酸骨架修饰、26 种碱基修饰以及 14 种可对碱基和/或糖分子进行的修饰。siRNA 中修饰类型和数量的分布如图 2 所示。

A. cm-siRNA 有义链和反义链中化学修饰数量的分布。
B. 有义链中化学修饰类型的分布。
C. 反义链中化学修饰类型的分布。
- 有义链:在DNA双链中,不作为模板链的那条链称为有义链。有义链的核苷酸序列与转录生成的RNA序列一致,但不包含遗传信息。在转录过程中,有义链作为模板指导RNA的合成。
- 反义链:作为模板链的那条链称为反义链。反义链包含遗传信息,但在转录过程中不直接参与RNA的合成。
我们创建了三个基准数据集,以确保对 Cm-siRPred 与之前的方法进行公平的评估和比较。首先,我们根据效率得分将数据分为 10 个分区。我们从每个分区中随机抽取 90% 的条目作为数据集 1,其余 10% 的条目构成数据集 2。数据集 1 用于 5 倍验证,数据集 2 作为独立测试集。此外,我们还编制了一个单独的数据集,即数据集 3,其中包括四种经 FDA 批准的化学修饰 siRNA 药物(见表 1)。

2.3.特征呈现
我们采用了多视角学习策略来表示 cmsiRNA 的特征,包括 siRNA 的双链序列(有义链和反义链)、化学修饰分子及其理化性质。
2.3.1.双链序列
cm-siRNA 的双链序列由核糖核苷酸(AGUC)和脱氧胸苷(T)组成。我们使用自然语言处理中常用的 One-hot 编码方法对序列进行数字转换。在我们的研究中,我们将 K 值设为 5,代表四个核糖核苷酸和一个脱氧胸苷。我们将 cm-siRNA 序列窗口设为 25,这样双链 siRNA 就可以用 50 × 5 矩阵表示(每条链用 25 × 5 矩阵表示)。
![]()
2.3.2.分子指纹
我们使用分子存取系统(MACC)编码 siRNA 有义/反义链的分子结构,同时捕捉 RNA 序列碱基组成和化学修饰信息。我们使用 Python 中的 rdkit 软件包(版本 2022.9.5)将碱基和化学修饰转换成 MACC。具体来说,rdkit 将 MACC 中的 166 个亚结构字典与目标分子进行匹配,并分配一个二进制值(0/1)来表示每个亚结构的存在或不存在。因此,cm-siRNA 使用 rdkit 表示为 166 维的二进制向量。

2.3.3.理化性质
我们从 PubChem 数据库中检索了四种核糖核苷酸和脱氧胸苷的理化性质。[34] 共获得了十种不同的理化性质,如分子量和拓扑表面积(见表 S1)。

然后,我们根据 cm-siRNA 的属性对其理化性质进行表征。与双链序列类似,我们将 cm-siRNA 序列窗口设置为 25,从而将双链 siRNA 表示为 50 × 10 矩阵(每条链表示为 25 × 10 矩阵)。
2.4.Cm-siRPred 架构
为了有效整合和处理 cmsiRNA 的多视角特征,我们采用了交叉注意机制,而不是简单地连接特征向量。这种机制有助于识别和强调这些特征之间的相互关系,从而获得更全面的表征。在交叉注意层之后,一个三维卷积神经网络(3D-CNN)被用来学习表征向量的局部特征。最后,一个两层全连接网络被用于预测(见图 3)。
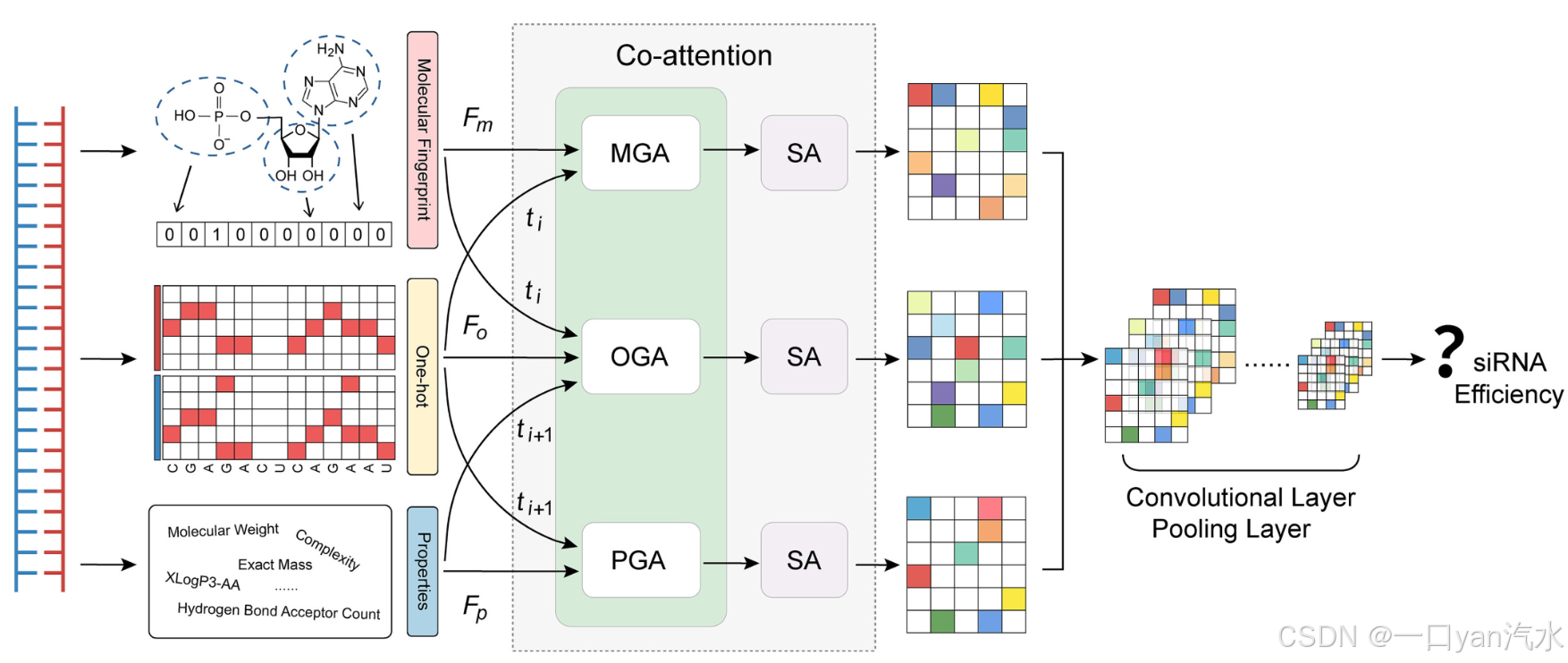
分子指纹—one-hot编码—属性
2.4.1.交叉注意模块
交叉注意机制的基本单元是自我注意(SA)。自注意模块以查询(Q)、键(K)和值(V)向量为输入,通过下式得到输出的被注意特征:

其中,Q,K,V∈,
∈
。
是每个 "头 "的输出维度,d 是 SA 模块的输入维度。h 代表并联 "头 "的数量,h =
。我们设定 h = 4。

多头注意力机制—相加和归一化
在交叉关注的背景下,该机制可以在不同的视图模块之间交换查询(Q)向量(图 4)。这使得每个模块在处理自身模块的同时,还能关注其他模块的重要特征,从而加强不同视图之间的信息交互和整合,正如后面的等式所示:

其中,,
∈
表示特征矩阵,这些特征矩阵由嵌入层根据one-hot编码、MACCS 或
中的
、
、
进行变换。我们设置 d = 512,l = 50。
2.4.2.3D-CNN 模块
Cm-siRPred 在交叉注意机制之后集成了一个 3D-CNN 模块。该模块由两层组成,每层包括一个卷积操作、一个整流线性单元(ReLU)激活函数和一个最大池化层,如下式所示:

其中, 表示 n 层的卷积运算,
表示上一层的输入。ReLU 表示 ReLU 激活函数,
表示 n 层的最大池化操作。具体来说,该模型从一个卷积层开始,该层接收三个输入通道并生成 16 个特征图。随后是第二个卷积层,将通道从 16 个扩展到 32 个。每个卷积层都采用 3 × 3 内核,步长为 1,并填充 1 个像素,以保持特征图的尺寸。卷积后,整流线性单元(ReLU)激活函数产生非线性,然后进行 2 × 2 最大池化,以实现高效的向下采样。
2.4.3.分类模块
3D-CNN 部分的输出经扁平化后输入全连接部分,该部分由两层组成,用于高级特征学习和最终预测。初始全连接层接受大小为 32 × 12 × 128 的输入,输出 128 个节点。然后通过 ReLU 激活函数和 50 % 的剔除层对其进行处理,从而增强模型的泛化能力并减少过度拟合。最后的输出层有一个节点,使用 sigmoid 激活函数,使模型的输出介于 0 和 1 之间。
2.5.比较研究
在比较研究中,我们在独立数据集(数据集 2)上将 Cm-siRPred 与其他几种方法进行了比较。基于多头注意力的方法去除了不同视图模块之间的查询向量交换处理,因此可以作为对比实验中的对照模型,从而更好地突出交叉注意力作为特征融合策略的优势。此外,我们还使用了不同数据表示的 SMEpred 方法,包括 SMEpred(MNC)、SMEpred(MNC + BIN-AS(种子 13))和 SMEpred(MNC + BIN AS(后 8))。在 SMEpred 中,siRNA 序列由五个常见核苷酸和化学修饰核苷酸组成。MNC 按每种核苷酸的比例表示序列。BIN 表示法将 MNC 转化为二进制向量。BINAS(种子 13)表示 siRNA 反义链 5′ 端前 13 个核苷酸的二进制载体,而 BIN AS(最后 8)表示 3′ 端最后 8 个核苷酸。由于 SMEpred 在线服务器出现故障,我们根据上述原则重建了模型,并使用相同的数据集进行了训练。
2.6.性能评估指标
我们采用了 5 个指标来评估模型的性能,包括皮尔逊相关系数(PCC)、斯皮尔曼秩相关系数(SCC)、曲线下面积(AUC)、平均绝对误差(MAE)和平均平方误差(MSE)。

其中,cov 是协方差函数, 是 X 的标准偏差,
是 Y 的标准偏差。此外,
代表特定 cm-siRNA 的预测效率,
代表同一 cm-siRNA 的实际观察效率。
2.7.模型解释
为了阐明 Cm-siRPred 的回归过程,我们使用了 SHapley Additive exPlanations(SHAP),这是一种解释机器学习和深度学习模型输出的方法。它通过比较有特征和无特征的预测结果来量化每个特征对预测的贡献,并在一个合作游戏中计算回报,在这个游戏中,特征 "合作 "来实现预测。我们使用函数 shap.DeepExplainer()初始化了深度学习模型的专用组件 SHAP DeepExplainer。初始化时使用了我们的 Cm-siRPred 模型和由 500 个随机样本组成的数据集子集作为背景数据。解释器建立后,我们计算了整个数据集的 SHAP 值,以确保对模型的预测行为进行全面分析。我们使用函数 explainer. shap_values()进行了这一综合计算,该函数可为每个预测结果生成详细的特征贡献。
3.结果
3.1.性能评估

注:粗体数字代表相应指标下的最佳性能,括号中的数字代表标准偏差。
为了证明基于多视角注意力算法的 Cm-siRPred 所提高的性能和能力,我们在数据集 1 上将 Cm-siRPred(交叉注意力)与另一种模型(多头注意力)和当前先进的算法(使用不同特征描述模型的 SMEpred)进行了比较。评估性能时使用了多种指标,包括 PCC、SCC、AUC、MAE 和 MSE。如表 2 和图 5A 所示,Cm-siRPred 在所有指标上都优于其他算法,PCC、SCC、AUC、MAE 和 MSE 值分别为 0.8283、0.8263、0.9147、0.1152 和 0.0288。在比较分析中,Cm-siRPred 的表现优于大多数其他模型,在大多数指标上都有显著的统计学差异(图 5C)。虽然缺乏多视角策略的多头注意力模型的有效性略低于 Cm-siRPred,但它仍能超越其他算法。不过,Cm-siRPred 和多头注意力模型之间的性能差距在统计学上并不明显,尽管 Cm-siRPred 略占优势。此外,使用单核苷酸组成(MNC)和二进制序列编码(BIN)作为输入的 SMEpred 与仅使用 MNC 的 SMEpred 相比取得了更高的性能,这表明采用更全面的特征提取策略有可能进一步提高模型的能力。

A. 数据集 1 的 ROC 曲线。
B. 数据集 2 上的 ROC 曲线
C. 5 倍验证中 SCC、PCC、AUC、MAE 和 MSE 的方框图,
方框图上的 p 值由双侧 Student's t 检验估计。
*"表示 p < 0.05,"**"表示 p < 0.01,
"***"表示 p < 0.001,否则显示精确的 p 值。
3.2.独立数据集上的模型性能比较
为了评估 Cm-siRPred 的泛化能力,我们将其用于独立数据集(数据集 2),并与其他方法进行比较。所有用于比较的模型和方法都专门在数据集 1 上进行了训练,以确保比较的公平性。如表 3 和图 5B 所示,Cm-siRPred 在多个指标上都优于其他方法,其 PCC、SCC、AUC、MAE 和 MSE 值分别为 0.7700、0.7670、0.8931、0.1300 和 0.0379。值得注意的是,与 5 倍验证相比,该模型在独立数据集上的性能略有下降。

注:粗体数字代表相应指标下的最佳性能。
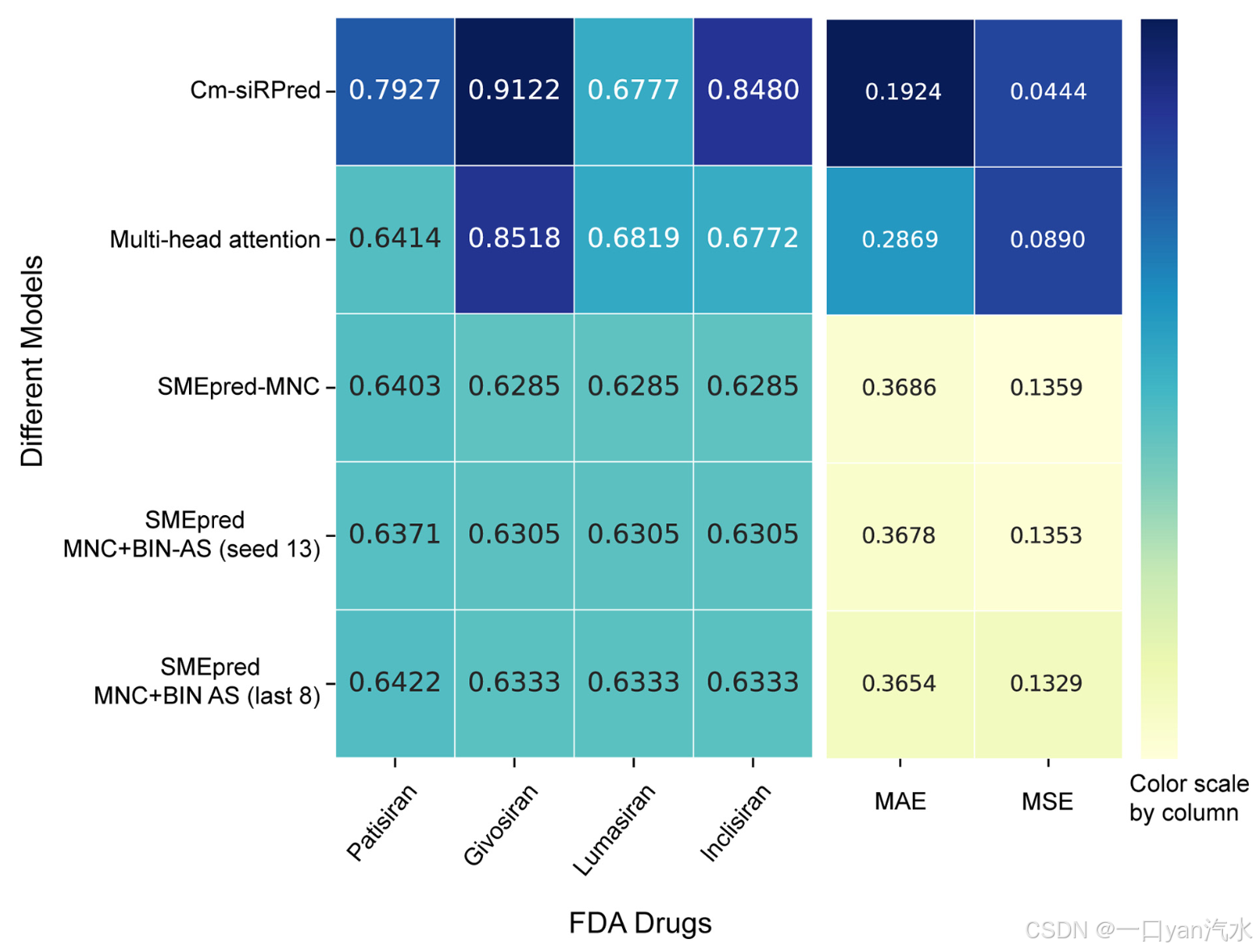
3.3.在 FDA 批准药物中的应用

注:粗体数字代表相应指标下的最佳表现。模型生成的药物效率预测分数范围为 0 至 1,数值越大,效率越高。
为全面评估 Cm-siRPred 在实际研发场景中的适用性,我们将 Cm-siRPred 和其他方法应用于四种 FDA 批准的 cm-siRNA 药物。如表 4 和图 6 所示,与其他方法相比,Cm-siRPred 的表现优于三种药物:Patisiran (0.7927)、Givosiran (0.9122) 和 Inclisiran (0.8480),仅在 Lumasiran (0.6819) 上略微落后于多头注意力方法。此外,就总体指标而言,Cm-siRPred 的 MAE 和 MSE 也优于其他方法。结果表明,Cm-siRPred 在实际应用中仍然保持了较高的准确性和鲁棒性,展示了其潜在的实用价值。
3.4.模型解释
鉴于深度学习模型的复杂性和不透明性,我们尝试使用 SHAP [35]探究 Cm-siRPred 的可解释性。我们评估了 cm-siRNA 的三个不同视角:双链序列(one-hot)、化学修饰分子(指纹)和理化性质(属性)。图 7 显示了基于三种视图的绝对平均特征重要性的前 10 个特征。在单击视图中,"反义 21 "和 "有义 21 "被确定为影响力最大的两个特征(图 7A)。同时,在指纹图谱视图中,前 10 个特征全部位于有义链(图 7B),而在属性视图中,大多数特征分布在 siRNA 序列的两端(图 7C)。这些结果表明,siRNA 的两端以及有义链对 cm-siRNA 的效率有重要影响。

A、B 和 C 分别表示双链序列特征、分子指纹特征和理化性质特征的平均绝对 SHAP 值。
3.5.用于 cm-siRNA 设计的网络服务器
为方便用户,Cm-siRPred 提供了一个精心设计的网络服务器,用于预测 cm-siRNA 的效率,并协助设计化学修饰的 siRNA 药物。该网络服务器有两个不同的模块:化学修饰设计模块和 cm-siRNA 效率预测模块。图 8A 展示了化学修饰设计模块,该模块具有可在线动态设计 cm-siRNA 药物的功能。用户首先输入 siRNA 序列的有义/反义链。点击 "生成修饰选择器 "后,会显示 siRNA 每个碱基的化学修饰选择器。然后,用户可以用鼠标为每个碱基选择无修饰、单一修饰或多重修饰。设计阶段完成后,"提交到服务器 "按钮将启动预测过程。此外,图 8B 展示了 cm-siRNA 效率预测模块,用户可通过该模块输入一批 cmsiRNA 序列(fasta 格式)及其相应的化学修饰信息(SMILES 格式),以进行效率预测。
4.讨论
基于机器学习技术预测Cm-siRNA的药效可以大大节省siRNA化学修饰设计的经济和时间成本,优化siRNA药物的研发过程。然而,目前现有的预测算法大多依赖于传统的机器学习方法,存在训练集小、数据表示能力有限、忽略 siRNA 的有义链信息、模型缺乏可解释性等局限性。在本研究中,我们开发了基于多视图学习策略的 Cm-siRPred 算法,该算法在交叉验证实验和独立数据集中均表现出最佳性能,显示出良好的准确性、鲁棒性和泛化能力。我们认为,Cm-siRPred 的优异性能主要归功于以下因素:(1) 采用深度学习框架,高效学习数据的非线性特征;(2) 采用多视角学习策略,全面表示 cm-siRNA 双链序列和化学修饰数据。(3) 利用交叉注意模型对不同表示向量之间的内在关系进行全局关联和探索。
此外,本研究还利用 SHAP(SHapley Additive exPlanations)进一步探讨了所提模型的可解释性。结果表明,siRNA 序列两端和有义链的修饰对 cmsiRNA 的功效有显著影响。siRNA 的末端修饰是最常见的修饰策略之一 [36,37]。目前的研究表明,在 siRNA 的 3′或 5′末端引入化学修饰基团可改变其结构和亲水性,从而提高 siRNA 在细胞中的稳定性和效率[38,39]。例如,许多研究表明,在 siRNA 的反义链 5′端和有义链 3′端加入硫代磷酸酯(PS)修饰,可显著提高反义链的选择性和 siRNA 的活性 [40,41]。此外,siRNA 有义链和反义链之间的热稳定性以及与靶 mRNA 的分子亲和力也会影响沉默效率[19]。这些结果表明,在开发 cm-siRNA 效能预测方法时,不应忽视有义链信息。
此外,本研究还开发了一个用于预测 cmsiRNA 效力和辅助 siRNA 药物化学修饰设计的网络服务器。该服务器包括两个模块:化学修饰设计模块和 cm-siRNA 效能预测模块。化学修饰设计模块允许用户在 siRNA 序列的不同位置自由添加各种化学修饰并预测其药效,提供了一个用户友好的在线工具。
然而,cm-siRPred 算法仍然存在一些局限性。首先,由于训练集中不同化学修饰的分布不均,目前的模型对一些罕见化学修饰的预测能力有限。此外,大部分训练数据来自体外实验,因此该算法无法准确模拟 cm-siRNA 在体内的疗效。今后,我们将进一步收集和改进 cm-siRNA 数据集,扩大数据集中化学修饰类型的覆盖范围,增加体内实验数据,并不断更新和优化算法,以解决上述局限性。
值得注意的是,用于开发靶向药物和 mRNA 疫苗的生成模型已经取得了重大进展[42,43]。因此,未来版本的 CmsiRPred 必须纳入这些技术。整合生成模型将提高 Cm-siRPred 预测 siRNA 疗效和特异性的准确性,尤其是在递送机制、免疫系统相互作用和分子设计方面。这种整合将扩大 Cm-siRPred 在治疗开发方面的潜力,特别是在设计更有效的基于 siRNA 的疗法方面。
总之,Cm-siRPred 是预测 cm-siRNA 药效和协助设计 cm-siRNA 药物的实用工具。我们相信,它能为 siRNA 化学修饰和药物活性研究提供技术支持,也能为基于小 RNA 的药物开发提供有效帮助。
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








