一起来品我的shit代码
实验题目
用遗传算法解决20个城市的TSP问题。
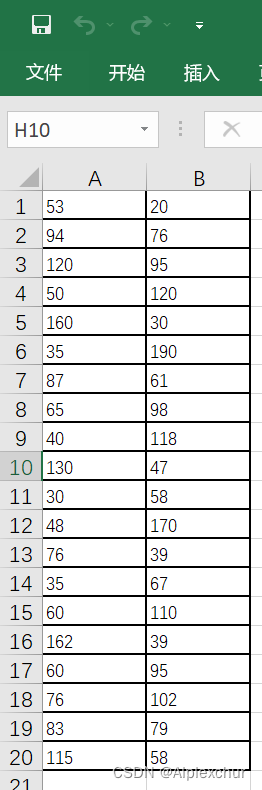
20个城市的坐标
| x | 53 | 94 | 120 | 50 | 160 | 35 | 87 | 65 | 40 | 130 |
| y | 20 | 76 | 95 | 120 | 30 | 190 | 61 | 98 | 118 | 47 |
| x | 30 | 48 | 76 | 35 | 60 | 162 | 60 | 76 | 83 | 115 |
| y | 58 | 170 | 39 | 67 | 110 | 39 | 95 | 102 | 79 | 58 |
问题分析
TSP问题又译为旅行推销员问题、货郎担问题,是数学领域中著名问题之一。假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路径的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。路径的选择目标是要求得的路径路程为所有路径之中的最小值。
算法设计
本实验采用遗传算法来解决旅行商问题。遗传算法是一种模拟自然选择和遗传机制的优化搜索算法,适用于寻找最优解的问题。
1. 初始化种群
首先,需要随机生成一个种群,种群中的个体代表问题的可能解。在本实验中,种群中的每个个体都是问题的一个可能解,即一条路径,其包含所有城市且每个城市只访问一次。因为旅行商需要途径所有城市并回到起点,所以每条路径以0号城市为首尾。在这里随机生成popsize个个体。
2. 适应度函数
接下来,定义一个适应度函数,用于评估种群中每个个体的优劣。适应度函数根据问题的特性来设计,通常是与问题相关的目标函数。在TSP问题中,适应度函数设计为路径长度的倒数,即路径长度越短,适应度越高。
3. 选择
在选择阶段,根据个体的适应度,选择适应度较高的个体作为父代,用于产生下一代个体。吧适应度归一化后采用累积概率的轮盘赌方法选择个体。
4. 交叉
选出的父代个体进行交叉操作,产生新的个体。交叉操作模拟了生物的交配过程,通过交叉将父代的遗传信息传递给子代。在本实验中,随机选取两个亲本,在随机位点进行互换,这里可能会出现无效解,经过去重处理、补足缺失处理后使其成为有效解。
5. 变异
在变异阶段,对新生成的个体进行随机变异操作,以增加种群的多样性。变异操作模拟了生物的基因突变过程,通过变异引入新的遗传信息。本实验采用两个基因位点互换的方式进行变异操作。
6. 更新种群
将新生成的个体与原种群进行合并,形成新一代种群。然后,根据预定的终止条件(例如达到最大迭代次数或找到满意的解),继续执行选择、交叉和变异操作,直到满足停止条件。
运算结果
采用如图数据,存放在city_coordinate.csv中。
实验总结
在本次实验中,首先对遗传算法进行了详细的设计和实现,包括种群初始化、适应度函数的设计、选择、交叉和变异等关键步骤。然后,根据问题的特点,编写了针对TSP的遗传算法实现代码,并在实验中进行了验证和测试。在实验过程中发现了一些影响算法性能的关键因素,如种群大小、交叉率、变异率等。通过调节这些参数,可以对算法的性能产生重要影响。另外,在实际应用中,需要根据具体问题的特点进行参数调优和算法设计,以获得更好的解决方案。
附录
import numpy as np
from matplotlib import pyplot as plt
import pandas as pd
plt.rcParams['font.sans-serif'] = ['SimHei'] # 运行配置参数中的字体(font)为黑体(SimHei)
class GA:
def cal_fitness(self, pop):
# 适应度函数
fitness = np.zeros((self.pop_size, 1))
for i in range(self.pop_size):
dis = 0
for j in range(self.gene_length - 2): # sqrt((x1-x2)**2+(y1-y2)**2) 还要回到起点
dis += np.sqrt((self.matrix[pop[i][j], 0] - self.matrix[pop[i][j + 1], 0]) ** 2 + (
self.matrix[pop[i][j], 1] - self.matrix[pop[i][j + 1], 1]) ** 2)
fitness[i] = 1 / dis # 取倒数 距离越小适应度越大
return fitness
def roulette_wheel_selection(self, pop, fitness):
# 轮盘赌
fit1 = (fitness - np.min(fitness)) / (np.max(fitness) - np.min(fitness)) # 区间归一化为[0,1]
fit2 = fit1 / np.sum(fit1) # 求每个个体的适应度占整体适应度之和的百分比
fit3 = np.cumsum(fit2) # 求累积概率
new_pop = pop.copy()
for i in range(self.pop_size):
r = np.random.rand()
for index in range(len(fit3)):
# 找到第一个比随机数小的个体
if r <= fit3[index]:
new_pop[i] = pop[index]
break
return new_pop
def __init__(self, city_coordinate, pop_size, max_generation, crossover_rate, mutation_rate):
self.matrix = city_coordinate
self.pop_size = pop_size
self.city_num = len(city_coordinate)
self.gene_length = self.city_num + 1
self.max_generation = max_generation
self.crossover_rate = crossover_rate
self.mutation_rate = mutation_rate
def generate_init_pop(self):
# 初始化种群
init_pop = np.zeros((self.pop_size, self.gene_length), dtype=int)
for i in range(self.pop_size):
# 生成以0号城市为首尾的随机排列
init_pop[i, :] = np.append(np.append(0, np.random.permutation(np.arange(1, self.city_num))), 0)
return init_pop
# selected_individual = roulette_wheel_selection(init_pop, fit3)
def crossover(self, pop):
# 交叉互换
for i in range(self.pop_size):
if np.random.rand() < self.crossover_rate: # 随机数小于交叉概率则进行交叉
father = pop[np.random.randint(self.pop_size)] # 随机选两个交叉对象
mother = pop[np.random.randint(self.pop_size)]
cross_point = np.random.randint(self.gene_length)
son = np.concatenate((father[0:cross_point], mother[cross_point:self.gene_length])) # 指定取父本前段基因和母本后段基因
# 可能存在无效解 就开始修复 把缺失的城市按顺序加到son里
unique_genes = list(set(son))
if len(unique_genes) != self.city_num: # 如果交叉后存在重复元素
for j in range(self.city_num):
if j not in unique_genes:
unique_genes.append(j) # 添加缺失的基因
unique_genes.append(0) # 回到起点0
son = np.array(unique_genes) # 将修复后的基因重新赋值给 son
pop[i] = son
return pop
def mutation(self, pop):
# 变异 这里采取的是两个基因位点互换
for i in range(self.pop_size):
if np.random.rand() < self.mutation_rate: # 随机数小于变异概率则进行变异
pos1 = np.random.randint(1, self.gene_length - 1)
pos2 = np.random.randint(1, self.gene_length - 1)
pop[i][pos1], pop[i][pos2] = pop[i][pos2], pop[i][pos1]
return pop
def solution(self):
pop = self.generate_init_pop()
dis = np.zeros((self.max_generation, 1))
best_dis = 1 / self.cal_fitness(pop)[0]
best_path = pop[0]
for i in range(self.max_generation):
fitness = self.cal_fitness(pop)
pop = self.roulette_wheel_selection(pop, fitness)
pop = self.crossover(pop)
pop = self.mutation(pop)
if 1 / max(fitness) < best_dis:
idx = np.where(fitness == max(fitness)) # 可能存在多个 取0号元素就行
best_dis = 1 / max(fitness)
best_path = pop[idx[0][0]]
dis[i] = best_dis
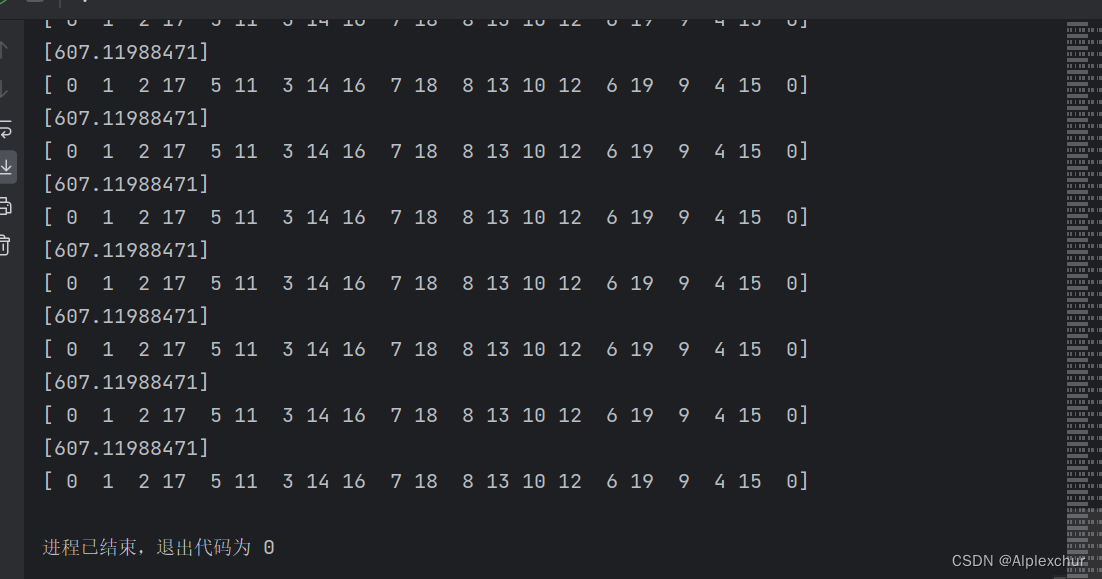
print(best_dis)
print(best_path)
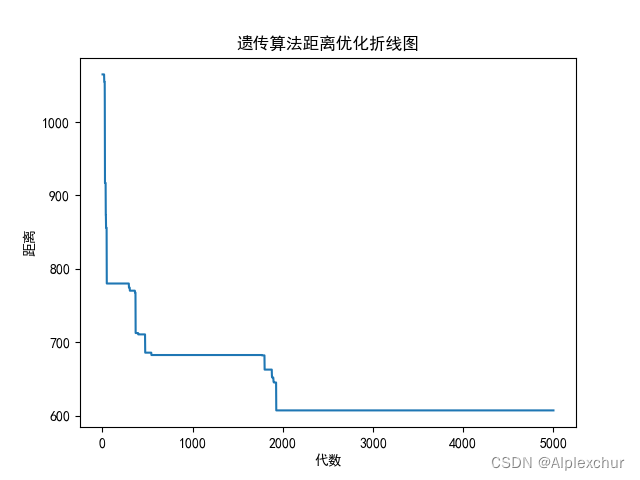
plt.plot(range(1, self.max_generation + 1), dis)
plt.title("遗传算法距离优化折线图")
plt.xlabel('代数')
plt.ylabel('距离')
plt.show()
city_coordinate = pd.read_excel('city_coordinate.xlsx', header=None).values
ga = GA(city_coordinate, pop_size=100, max_generation=500, crossover_rate=0.95, mutation_rate=0.2)
ga.solution()








 本文介绍了如何运用遗传算法解决20个城市TSP问题,详细描述了算法设计的各个环节,包括初始化种群、适应度函数、选择、交叉和变异。作者通过实验验证了算法的有效性,并探讨了关键参数对性能的影响。
本文介绍了如何运用遗传算法解决20个城市TSP问题,详细描述了算法设计的各个环节,包括初始化种群、适应度函数、选择、交叉和变异。作者通过实验验证了算法的有效性,并探讨了关键参数对性能的影响。














 1217
1217











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








