1. K-Means聚类
原理:
K-Means聚类通过迭代优化使得每个数据点到其最近质心的距离之和最小。该算法假设簇是球形的,并且每个簇的大小大致相等。
推导:
- 初始化K个质心,可以随机选择K个数据点作为初始质心。
- 将每个数据点分配到距离最近的质心,形成K个簇。
- 更新每个簇的质心为簇内所有数据点的平均值。
- 重复步骤2和3,直到质心位置不再变化或达到最大迭代次数。
数学表示:
 。
。
优点:
- 简单易理解:K-Means算法简单,易于实现和理解。
- 高效:K-Means算法的时间复杂度为O(nki),其中n是样本数量,k是聚类数,i是迭代次数。通常情况下,算法收敛速度较快。
- 适用广泛:适用于大多数数值型数据的聚类任务,特别是球形聚类效果较好。
缺点:
- 需要预设K值:需要提前指定聚类数K值,这在实际应用中可能不容易确定。
- 对初始值敏感:对初始质心的选择敏感,不同的初始质心可能导致不同的聚类结果。
- 只适用于线性分离:假设数据是线性可分的,无法处理复杂的非线性结构。
- 对噪声和离群点敏感:对噪声和离群点比较敏感,这些点可能会显著影响聚类结果。
算法步骤
- 初始化:随机选择K个初始质心。
- 分配数据点:将每个数据点分配到最近的质心,形成K个簇。
- 更新质心:计算每个簇的质心,即所有簇内数据点的平均值。
- 迭代:重复步骤2和3,直到质心不再发生变化或达到最大迭代次数。
示例
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
# 生成示例数据
centers = [[2, 2], [8, 8], [5, 5]]
X, _ = make_blobs(n_samples=300, centers=centers, cluster_std=0.7, random_state=42)
# 数据标准化处理
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 使用K-Means算法进行聚类(假设K=3)
k = 3
kmeans = KMeans(n_clusters=k, random_state=123)
kmeans.fit(X_scaled)
# 获取聚类标签
labels = kmeans.labels_
# 可视化聚类结果
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=labels, cmap='viridis')
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1], s=300, c='red', marker='X')
plt.title(f'K-Means Clustering (k={k})')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()

2. 均值漂移聚类 (Mean Shift Clustering)
原理:
均值漂移聚类是一种基于密度的聚类方法,通过迭代移动质心到数据点密度最高的区域,最终质心收敛到密度峰值。
推导:
- 初始化每个数据点为一个质心。
- 对每个质心,计算其邻域内所有数据点的密度梯度,移动质心到密度最大的方向。
- 重复步骤2,直到质心位置不再变化或移动距离小于设定阈值。
- 合并距离较近的质心,形成最终的簇。
数学表示:

优点:
- 不需要预设簇数:均值漂移聚类不需要像K-Means那样提前指定簇的数量。
- 适用于任意形状的簇:可以处理非球形簇,适用于任意形状的簇。
- 处理噪声:可以很好地处理噪声和离群点,因为它聚焦于高密度区域。
缺点:
- 计算量大:均值漂移聚类的计算量较大,尤其是对大规模数据集,计算速度较慢。
- 带宽选择困难:算法效果对带宽参数非常敏感,选择合适的带宽较为困难。
- 边界模糊:簇与簇之间的边界可能不清晰。
算法步骤:
- 初始化:将每个点初始化为一个质心。
- 计算密度梯度:在每个质心周围计算密度梯度。
- 更新质心位置:将质心移动到密度梯度最大的位置。
- 合并质心:当质心不再发生变化或质心移动距离小于设定阈值时,停止移动。将距离较近的质心合并为一个簇。
示例
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import MeanShift, estimate_bandwidth
from sklearn.datasets import make_blobs
# 生成示例数据
centers = [[2, 2], [8, 8], [5, 5]]
X, _ = make_blobs(n_samples=300, centers=centers, cluster_std=0.7, random_state=42)
# 数据标准化处理
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 使用均值漂移算法进行聚类
bandwidth = estimate_bandwidth(X_scaled, quantile=0.2, n_samples=200)
meanshift = MeanShift(bandwidth=bandwidth, bin_seeding=True)
meanshift.fit(X_scaled)
# 获取聚类标签和质心
labels = meanshift.labels_
cluster_centers = meanshift.cluster_centers_
# 可视化聚类结果
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=labels, cmap='viridis')
plt.scatter(cluster_centers[:, 0], cluster_centers[:, 1], s=300, c='red', marker='X')
plt.title('Mean Shift Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()

3. 基于密度的聚类方法 (DBSCAN)
原理: DBSCAN通过寻找密度高的区域进行聚类。它定义了核心点、边界点和噪声点,并根据这些定义形成簇。
推导:
- 标记核心点:对于数据集中每个点,计算其
eps邻域内的点数,如果邻域内点的数量不小于min_samples,则标记为核心点。 - 形成簇:将核心点及其
eps邻域内的所有点组成一个簇,并将这些点标记为已访问。 - 扩展簇:继续检查簇中的每个点,扩展其邻域内的所有核心点,直到没有新的点可以加入。
- 标记边界点和噪声点:剩余未访问的点如果在某个核心点的
eps邻域内,则标记为边界点,否则标记为噪声点。
优点:
- 不需要预设簇数:DBSCAN不需要像K-Means那样预设簇的数量。
- 能够发现任意形状的簇:能够识别任意形状的簇,而不仅仅是球形簇。
- 处理噪声和离群点:可以很好地处理噪声和离群点,不受离群点的影响。
缺点:
- 参数选择困难:需要指定两个参数
eps(邻域半径)和min_samples(最小样本数),参数选择不当会影响聚类效果。 - 高维数据处理困难:在高维数据中,DBSCAN效果可能不如其他聚类算法。
算法步骤:
- 标记核心点:对于数据集中每个点,计算其
eps邻域内的点数,如果邻域内点的数量不小于min_samples,则标记为核心点。 - 形成簇:将核心点及其
eps邻域内的所有点组成一个簇,并将这些点标记为已访问。 - 扩展簇:继续检查簇中的每个点,扩展其邻域内的所有核心点,直到没有新的点可以加入。
- 标记边界点和噪声点:剩余未访问的点如果在某个核心点的
eps邻域内,则标记为边界点,否则标记为噪声点。
示例
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.datasets import make_blobs
# 生成示例数据
centers = [[2, 2], [8, 8], [5, 5]]
X, _ = make_blobs(n_samples=300, centers=centers, cluster_std=0.7, random_state=42)
# 数据标准化处理
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 使用DBSCAN算法进行聚类
dbscan = DBSCAN(eps=0.3, min_samples=5)
dbscan.fit(X_scaled)
# 获取聚类标签
labels = dbscan.labels_
# 可视化聚类结果
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=labels, cmap='viridis')
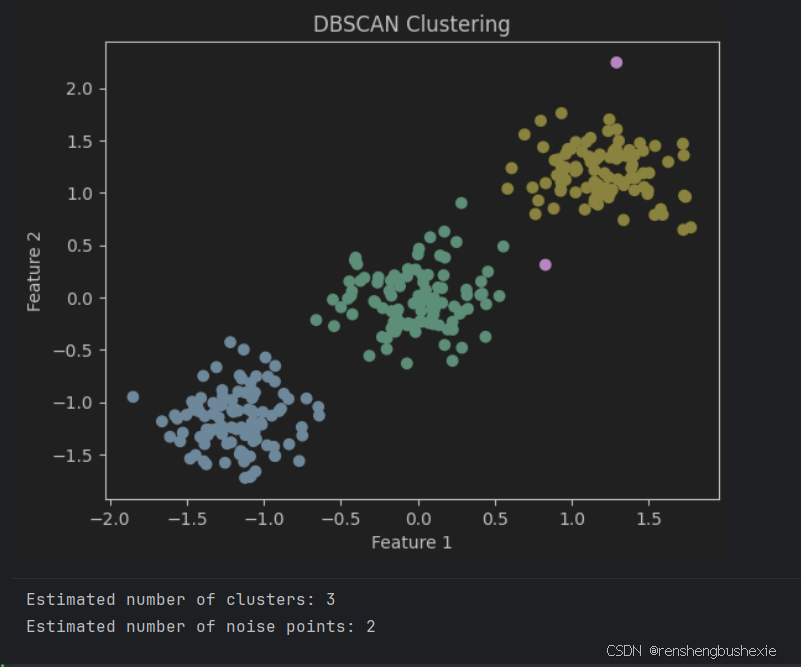
plt.title('DBSCAN Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()
# 打印聚类结果信息
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
n_noise_ = list(labels).count(-1)
print(f'Estimated number of clusters: {n_clusters_}')
print(f'Estimated number of noise points: {n_noise_}')
4. 高斯混合模型 (GMM)
原理: GMM是一种基于概率模型的聚类方法,它假设数据由多个高斯分布的混合模型生成。通过最大期望(EM)算法来估计模型参数。
推导:
- 初始化:选择初始参数,包括均值、协方差矩阵和混合系数。
- E步(期望步):计算每个数据点属于每个簇的后验概率。

3.M步(最大化步):更新参数,使得给定后验概率时,数据的对数似然最大化。

- 迭代:重复E步和M步,直到对数似然函数收敛或达到最大迭代次数。
优点:
- 适用于任意形状的簇:GMM可以生成椭圆形的簇,因此比K-Means更灵活,能够更好地拟合非球形簇。
- 软聚类:GMM提供了每个点属于每个簇的概率,可以进行软聚类,而不仅仅是硬聚类。
- 统计基础:GMM基于概率模型,提供了丰富的统计信息,如每个簇的均值、协方差等。
缺点:
- 参数估计复杂:需要估计多个参数(均值、协方差矩阵、混合系数),计算量大。
- 对初始值敏感:对初始参数值较敏感,可能导致局部最优解。
- 需要预设簇数:与K-Means类似,GMM也需要预设簇数K。
算法步骤:
- 初始化:选择初始参数,包括均值、协方差矩阵和混合系数。
- E步(期望步):计算每个数据点属于每个簇的后验概率。
- M步(最大化步):更新参数,使得给定后验概率时,数据的对数似然最大化。
- 迭代:重复E步和M步,直到对数似然函数收敛或达到最大迭代次数。
示例
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.mixture import GaussianMixture
from sklearn.datasets import make_blobs
# 生成示例数据
centers = [[2, 2], [8, 8], [5, 5]]
X, _ = make_blobs(n_samples=300, centers=centers, cluster_std=0.7, random_state=42)
# 数据标准化处理
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 使用高斯混合模型进行聚类(假设K=3)
k = 3
gmm = GaussianMixture(n_components=k, random_state=123)
gmm.fit(X_scaled)
# 获取聚类标签
labels = gmm.predict(X_scaled)
# 获取每个数据点的簇概率
probabilities = gmm.predict_proba(X_scaled)
# 可视化聚类结果
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=labels, cmap='viridis')
plt.scatter(gmm.means_[:, 0], gmm.means_[:, 1], s=300, c='red', marker='X')
plt.title('GMM Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()
# 打印聚类结果信息
print(f'Estimated number of clusters: {k}')

5. 凝聚层次聚类
原理: 凝聚层次聚类是一种自底向上的聚类方法,它通过迭代合并最相似的簇来生成层次结构(树状图)。
推导:
- 初始化:将每个数据点看作一个单独的簇。
- 计算距离:计算所有簇之间的距离(可以使用各种距离度量,如欧氏距离、曼哈顿距离等)。
- 合并最近的簇:找到最近的两个簇并将它们合并为一个簇。
- 更新距离矩阵:更新距离矩阵,计算新簇与其他所有簇之间的距离。
- 重复:重复步骤3和4,直到所有簇合并为一个簇或达到预定的簇数。
距离计算方法:

优点:
- 不需要预设簇数:与K-Means不同,凝聚层次聚类不需要预先设定簇的数量。
- 生成层次结构:可以生成一个树状结构(树状图),方便观察不同层次的聚类结构。
- 适用于任意形状的簇:可以识别任意形状的簇,适用性强。
缺点:
- 计算复杂度高:凝聚层次聚类的计算复杂度较高,尤其是对于大规模数据集,计算速度较慢。
- 对噪声和离群点敏感:对噪声和离群点比较敏感,这些点可能会影响聚类结果。
- 无法撤销合并:一旦两个簇合并,就无法撤销,这可能导致次优的聚类结果。
算法步骤:
- 初始化:将每个数据点看作一个单独的簇。
- 计算距离:计算所有簇之间的距离(可以使用各种距离度量,如欧氏距离、曼哈顿距离等)。
- 合并最近的簇:找到最近的两个簇并将它们合并为一个簇。
- 更新距离矩阵:更新距离矩阵,计算新簇与其他所有簇之间的距离。
- 重复:重复步骤3和4,直到所有簇合并为一个簇或达到预定的簇数。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import AgglomerativeClustering
from scipy.cluster.hierarchy import dendrogram, linkage
# 生成示例数据
from sklearn.datasets import make_blobs
centers = [[2, 2], [8, 8], [5, 5]]
X, _ = make_blobs(n_samples=300, centers=centers, cluster_std=0.7, random_state=42)
# 数据标准化处理
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# 使用凝聚层次聚类(假设K=3)
k = 3
agg_cluster = AgglomerativeClustering(n_clusters=k, metric='euclidean', linkage='ward')
agg_cluster.fit(X_scaled)
# 获取聚类标签
labels = agg_cluster.labels_
# 可视化聚类结果
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=labels, cmap='viridis')
plt.title('Agglomerative Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()
# 绘制树状图
linked = linkage(X_scaled, method='ward')
dendrogram(linked)
plt.title('Dendrogram')
plt.xlabel('Samples')
plt.ylabel('Distance')
plt.show()
# 打印聚类结果信息
n_clusters_ = len(set(labels))
print(f'Estimated number of clusters: {n_clusters_}')


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








