小罗碎碎念
方向分析
在医学AI研究领域,HPV相关口咽鳞状细胞癌(OPSCC)的精准预后预测一直是关键难题。这篇发表于eBioMedicine 2025年的论文聚焦于此,提出了一种创新的解决方案。

可能部分老师/同学,对于这个期刊不熟悉,这里稍微介绍一下。

这篇文章发表在eBioMedicine,但是,文章的数据集只有277例患者,并且,没有基因组学的数据,没有外部验证的队列,现在清楚为啥我要单独把这个拿出来介绍一下了吧。
| 作者身份 | 姓名 | 单位 |
|---|---|---|
| 第一作者 | Bolin Song | 佐治亚理工学院和埃默里大学华莱士·H·库尔特生物医学工程系 |
| 通讯作者 | Anant Madabhushia | 佐治亚理工学院和埃默里大学华莱士·H·库尔特生物医学工程系;亚特兰大退伍军人事务医疗中心 |
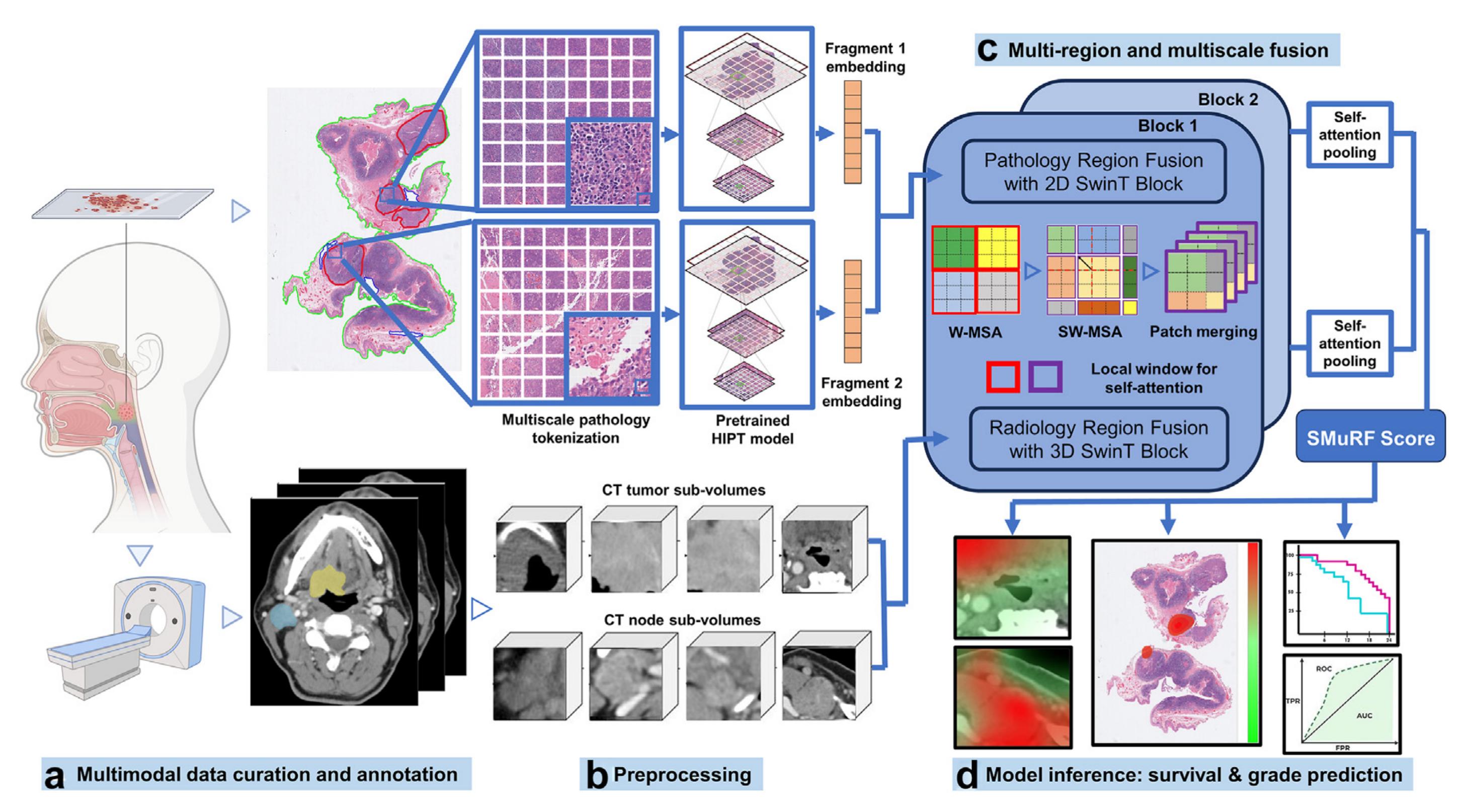
SMuRF:基于Swin Transformer的多模态和多区域数据融合框架
该模型整合了放射学CT与病理学全切片图像(WSI)的特征,通过交叉模态和跨区域窗口的多头自注意力机制,捕捉肿瘤不同区域和图像尺度间的相互作用,以此预测OPSCC患者的生存情况和肿瘤分级。
在277例患者的队列研究中,SMuRF展现出强大性能,其DFS预测的C指数达0.81,肿瘤分级分类的AUC为0.75,显著优于单模态模型,且是独立的预后生物标志物。
对于从事医学AI研究的人员而言,此研究意义重大。SMuRF的成功应用,为多模态数据融合在癌症预后预测方面提供了新的思路和方法。其多尺度和多区域的成像融合策略,可拓展到其他癌症研究中,助力解决多模态学习和数据整合的难题。
不过研究也存在局限,如未纳入基因组数据、缺乏外部验证等,这也为后续研究指明了方向,激励科研人员进一步优化模型,推动医学AI在癌症诊疗领域的深入发展。
交流群
欢迎大家加入【医学AI】交流群,本群设立的初衷是提供交流平台,方便大家后续课题合作。
目前小罗全平台关注量61,000+,交流群总成员1400+,大部分来自国内外顶尖院校/医院,期待您的加入!!
由于近期入群推销人员较多,已开启入群验证,扫码添加我的联系方式,备注姓名-单位-科室/专业,即可邀您入群。
知识星球
如需获取推文中提及的各种资料,欢迎加入我的知识星球!
限时优惠,通过个人微信订阅,可立减20!
一、文献概述
本文旨在通过整合放射学和病理学数据,利用深度学习预测HPV相关口咽鳞状细胞癌(OPSCC)的预后,开发出Swin Transformer 为基础的多模态框架(SMuRF)。
- 研究背景:HPV相关OPSCC发病率上升,现有分期系统虽区分HPV状态但患者仍面临生存挑战,仅依靠分子生物标志物存在局限性,而整合病理学和放射学信息可提供多尺度肿瘤洞察,此前多模态研究在该领域关注较少。
- 文献调研: (“deep learning” OR“machine learning” OR “artificial intelligence” OR “AI”) AND (“multimodal learning” OR “data integration” OR “data fusion”) AND (“medical images” OR “radiology”) AND (“pathology” OR “whole slide images” OR “biopsy”)
- 研究方法
- 数据收集:经机构审查委员会批准,回顾性收集277例患者的CT扫描、病理全切片图像、临床和生存结局信息,划分训练集、验证集和测试集,确定DFS和肿瘤分级等终点指标。
- 数据预处理:病理WSI先排除背景、分割轮廓,再用HIPT方法降维;放射学CT重采样、归一化,扩展并裁剪肿瘤和淋巴结体积,进行数据增强。
- 模型构建:采用SwinT块提取层次嵌入,通过注意力机制融合多区域和多尺度图像嵌入,使用多任务学习网络输出风险评分,对比多种融合方法,并计算IG和注意力热图辅助模型解释。
- 模型训练与评估:基于PyTorch框架,使用特定超参数训练模型,进行多种统计分析评估模型性能。
- 研究结果
- 临床特征:277例患者中多数为男性,中位年龄59岁,各数据集临床特征无显著差异。
- 模型性能:SMuRF在DFS预测和肿瘤分级分类上表现良好,C指数达0.81 ,AUC为0.75 ,优于单模态模型。多变量分析显示,SMuRF是独立预后生物标志物,在预测DFS和肿瘤分级时贡献最大,且对批效应有抗性。
- 模型可解释性:IG热图表明原发肿瘤的瘤内和瘤周区域、淋巴结内部特征具有预后价值;注意力热图显示不同尺度下病理WSI的形态学特征。
- 研究结论:SMuRF能有效整合多尺度成像数据,显著提升OPSCC风险分层和预后预测能力,有望指导治疗策略调整。但研究存在局限性,如未纳入基因组数据、缺乏外部验证等,未来可进一步研究拓展模型应用。
二、基于多模态数据的SMuRF模型流程
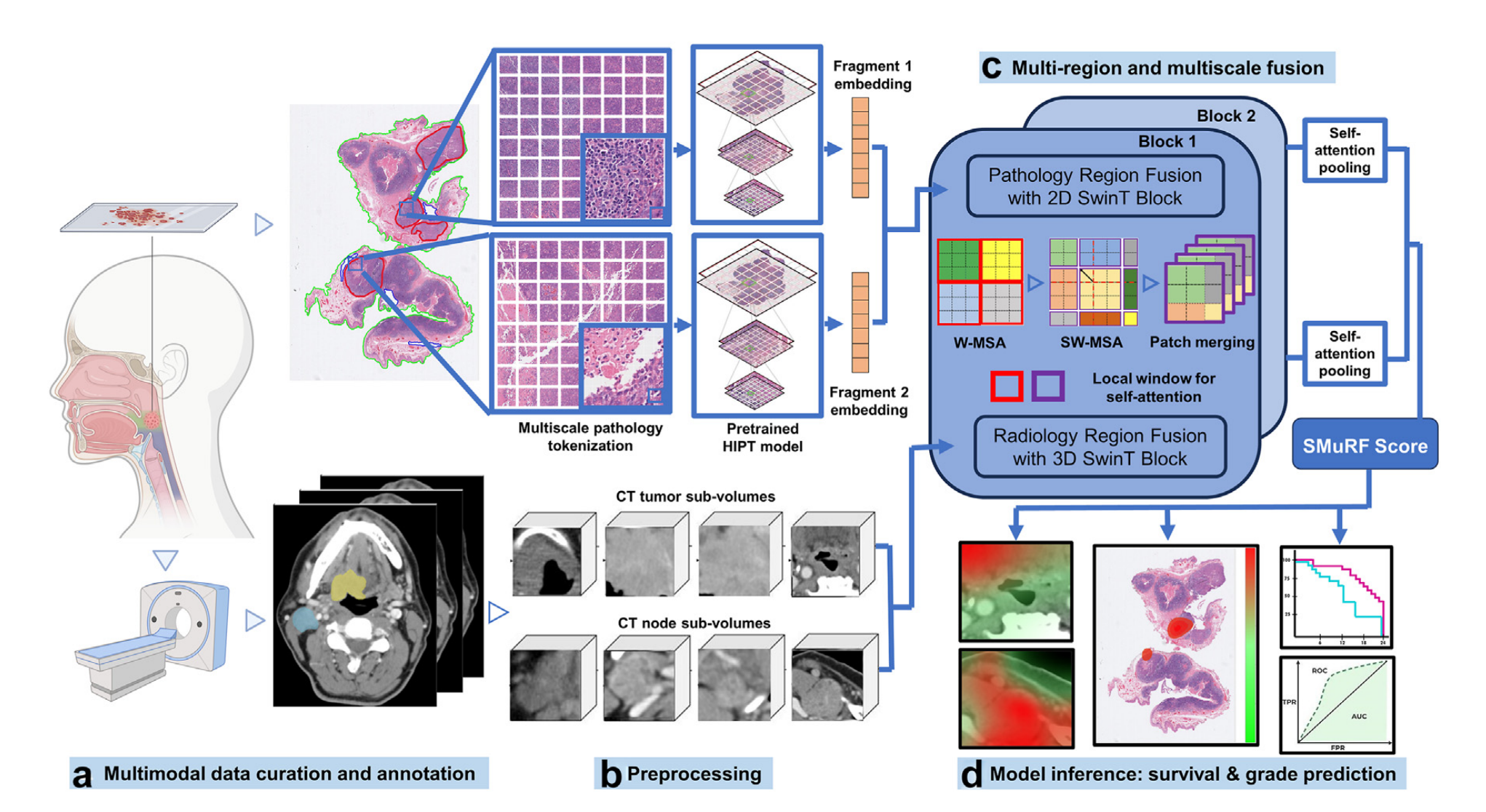
- 多模态数据管理与标注(Multimodal data curation and annotation):整合头颈部的病理切片数据和CT扫描数据,对病理切片中的肿瘤区域和CT影像中的肿瘤及淋巴结区域进行标注。
- 预处理(Preprocessing):病理切片进行多尺度标记化处理,通过预训练的HIPT模型生成片段嵌入;CT影像则分割出肿瘤子体积(CT tumor sub - volumes)和淋巴结子体积(CT node sub - volumes) 。
- 多区域和多尺度融合(Multi - region and multiscale fusion):使用2D SwinT模块进行病理区域融合,3D SwinT模块进行放射学区域融合,通过窗口多头自注意力(W - MSA)、滑动窗口多头自注意力(SW - MSA)和补丁合并(Patch merging)等操作,结合自注意力池化,得出SMuRF分数。
- 模型推理:生存和分级预测(Model inference: survival & grade prediction) :利用SMuRF分数对患者的生存率和肿瘤分级进行预测,通过可视化热图和生存曲线等展示预测结果。
三、HPV相关口咽鳞状细胞癌患者的多因素分析
这一部分展示了不同变量与生存风险比和肿瘤分级比值比的关系。
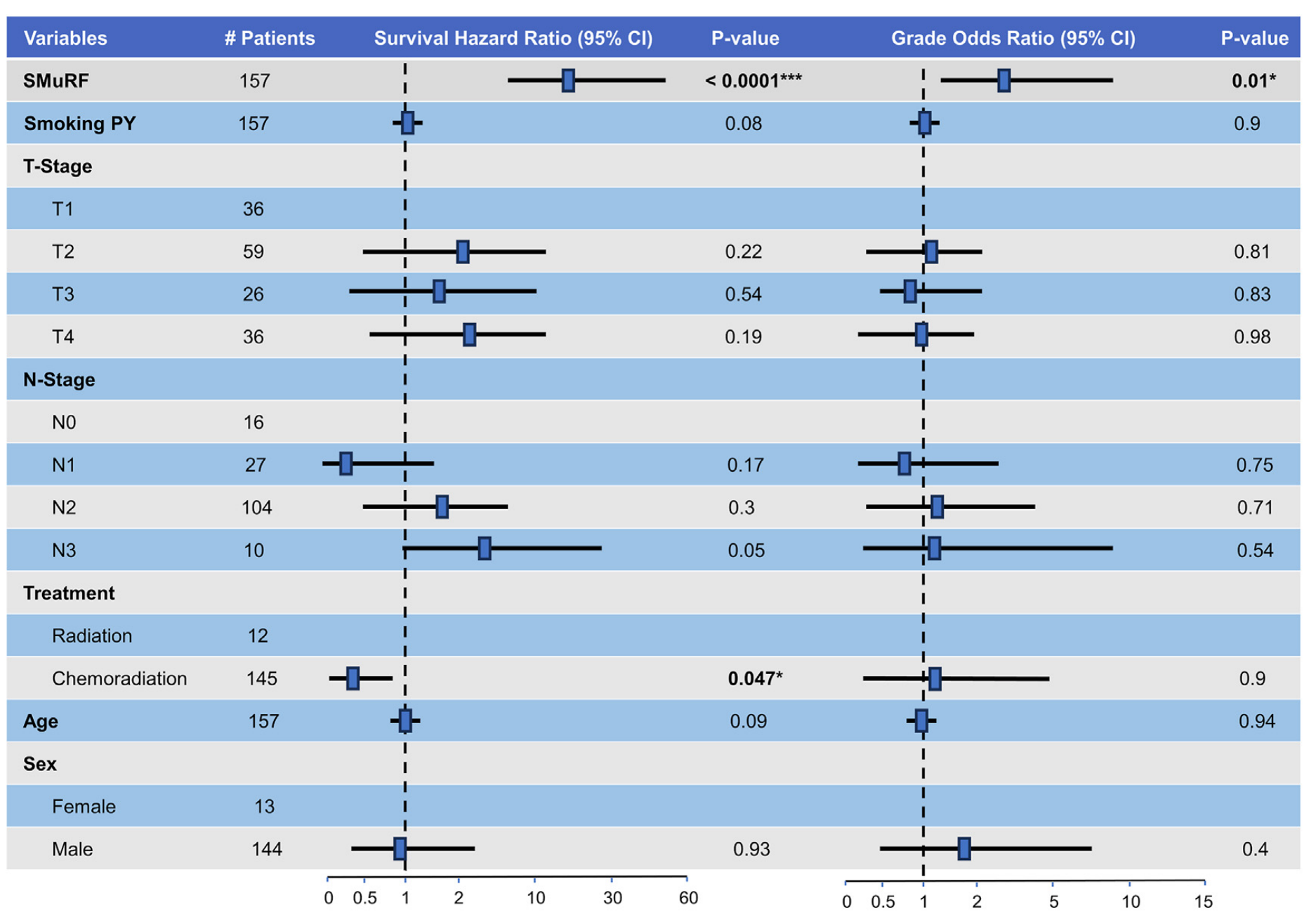
- SMuRF:涉及157名患者,生存风险比的P值小于0.0001(极显著),分级比值比的P值为0.01(显著) ,说明SMuRF与患者生存和肿瘤分级有显著关联。
- 吸烟包年数(Smoking PY):涉及157名患者,生存风险比和分级比值比的P值分别为0.08和0.9,与患者生存和肿瘤分级无显著关联。
- T分期(T - Stage):不同T分期(T1 - T4)患者数量不同,各分期的生存风险比和分级比值比的P值均大于0.05,与患者生存和肿瘤分级无显著关联。
- N分期(N - Stage):不同N分期(N0 - N3)患者数量有差异,各分期的生存风险比和分级比值比的P值均大于0.05,与患者生存和肿瘤分级无显著关联。
- 治疗方式(Treatment):分为放疗(Radiation )和放化疗(Chemoradiation),放化疗组生存风险比的P值为0.047(显著),分级比值比的P值为0.9,说明放化疗与患者生存有显著关联,与肿瘤分级无显著关联。
- 年龄(Age):涉及157名患者,生存风险比和分级比值比的P值分别为0.09和0.94,与患者生存和肿瘤分级无显著关联。
- 性别(Sex):女性13名,男性144名,生存风险比和分级比值比的P值分别为0.93和0.4 ,与患者生存和肿瘤分级无显著关联。
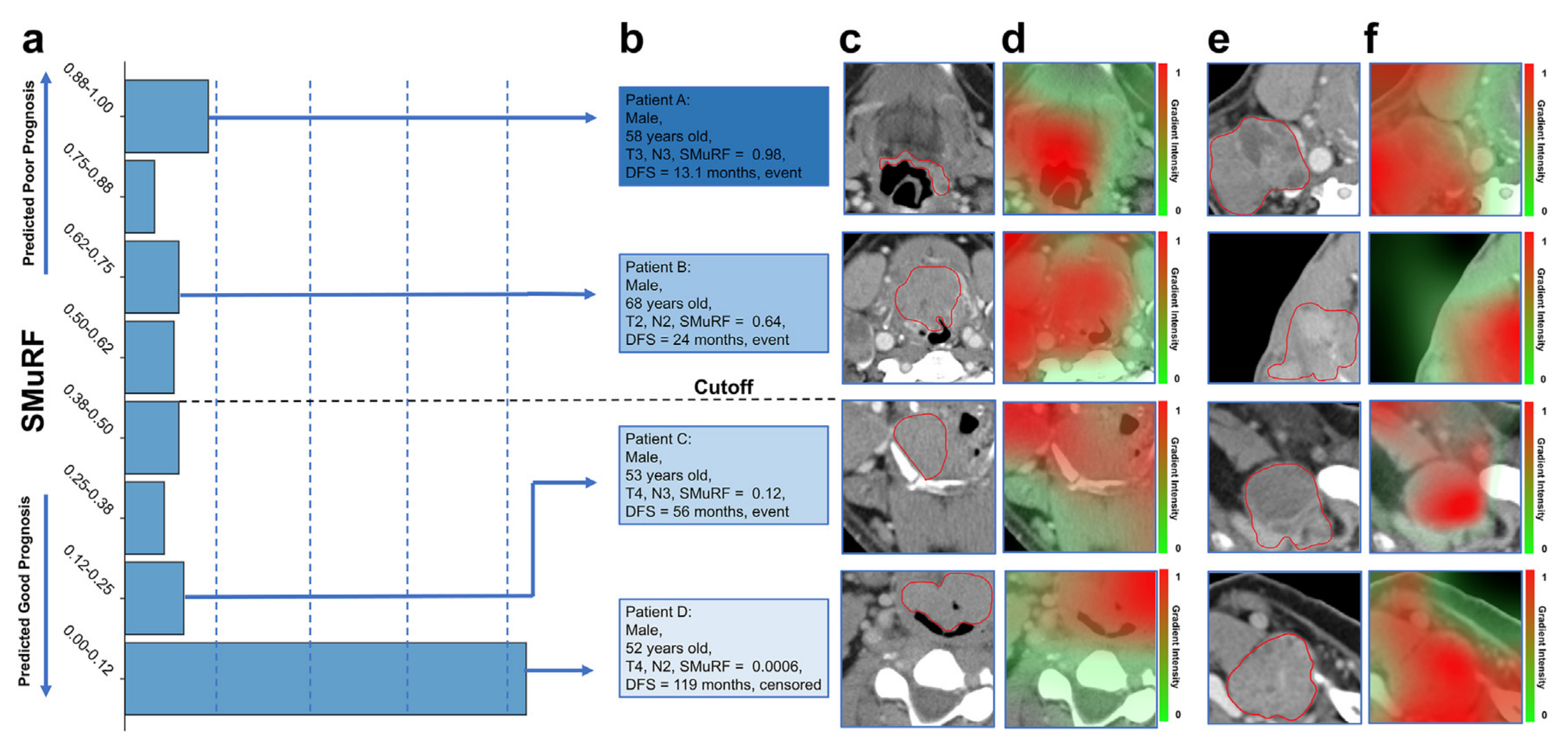
四、SMuRF模型在病理全切片图像(WSI)分析中的应用
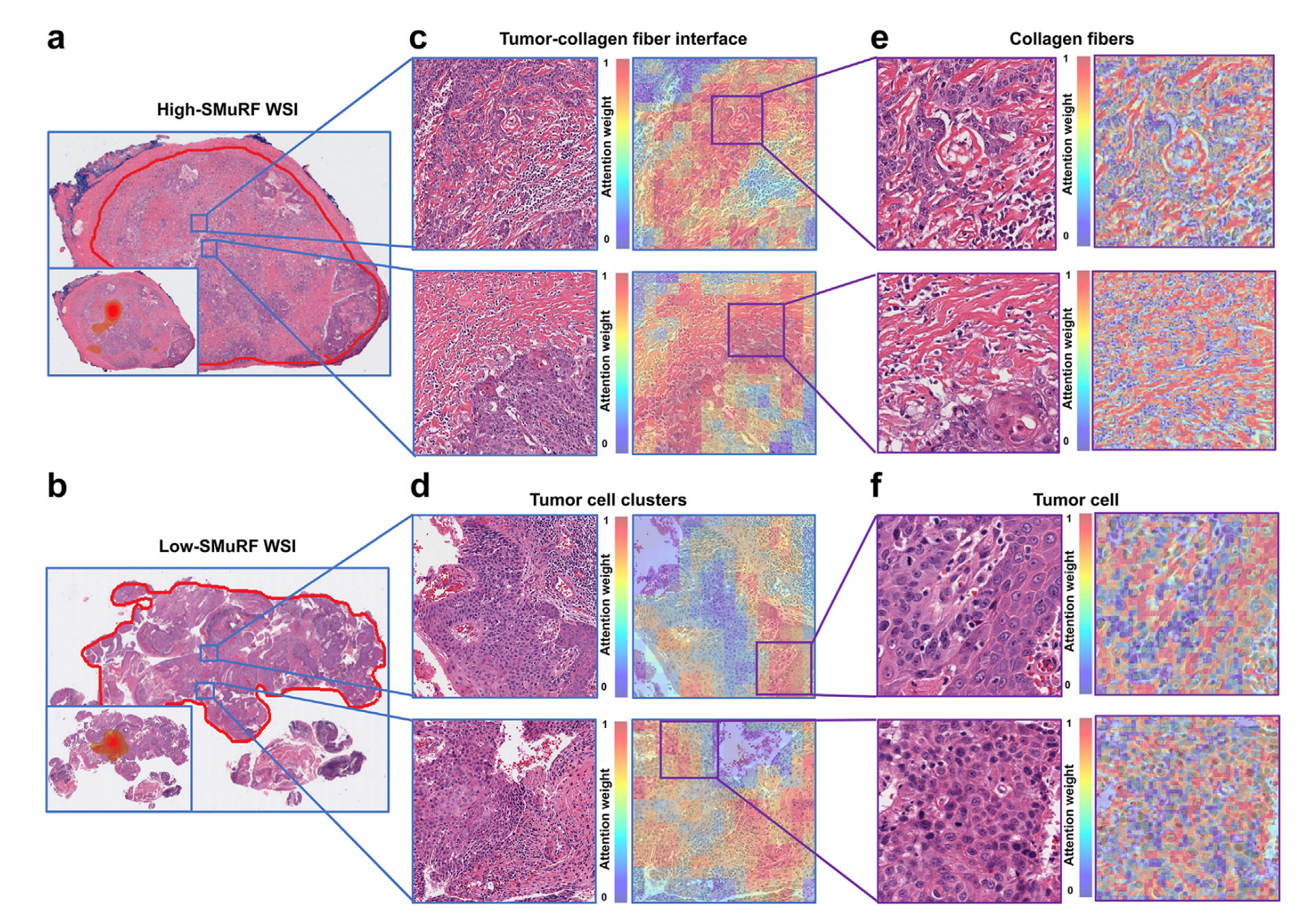
- 图像标注与区域检测:图a和图b分别展示了一个高SMuRF值和一个低SMuRF值的病理WSI,红色边界标注了原发肿瘤区域,并且通过集成梯度(IG)叠加突出了SMuRF检测到的与预后相关的区域(左下角小图)。
- 注意力热图展示:对于每个病理WSI,图c、d和图e、f分别展示了在预后相关区域内,大小为2048×2048的两个示例区域及其对应的注意力热图。在256像素的宏观尺度下,高SMuRF值患者的HIPT模型注意力热图显示,与肿瘤 - 胶原纤维界面相关的高注意力区域更密集,而低SMuRF值患者的WSI主要包含肿瘤细胞簇。在16像素的微观尺度下,高SMuRF值的注意力热图突出单个胶原纤维,而低SMuRF值的注意力热图主要强调肿瘤细胞。这些形态特征(如肿瘤 - 胶原纤维界面、肿瘤细胞簇)的存在,都经过病理学家评估和确认。
五、模型搭建
注意,这不是项目复现教程,只是在分析作者的模型构建思路。
5-1:需要安装的依赖项
einops
h5py
numpy
pandas
matplotlib
medpy
opencv-python
openslide-python
Pillow
scikit-learn
scikit-image
lifelines
pynrrd
torch
torchvision
torchaudio
tqdm
webdataset
5-2:数据预处理与加载模块
- 自定义数据对齐函数(
custom_collate)
def custom_collate(data):
ct_tumor, ct_lymphnodes, pathology, y, time, event, ID = zip(*data) # 解包数据元组
max_sizes = (max([path.shape[0] for path in pathology]), # 计算最大病理特征尺寸
max([path.shape[1] for path in pathology]))
# 病理数据维度调整与填充
for i in range(len(pathology)):
pathology[i] = torch.moveaxis(pathology[i], -1,0) # 调整维度顺序
pad_2d = max_sizes[1] - pathology[i].shape[2]
pad_3d = max_sizes[0] - pathology[i].shape[1]
padding = (floor(pad_2d/2), ceil(pad_2d/2), # 对称填充计算
floor(pad_2d/2), ceil(pad_2d/2),
floor(pad_3d/2), ceil(pad_3d/2))
m = torch.nn.ConstantPad3d(padding, 0) # 创建填充层
pathology[i] = m(pathology[i])
pathology[i] = torch.permute(pathology[i], (1,0,2,3)).float() # 维度置换
关键步骤说明:
- 使用
zip(*data)解压批次数据 - 计算最大特征尺寸用于标准化填充
- 通过
ConstantPad3d实现三维对称填充 - 维度置换适配神经网络输入要求
- 放射-病理联合数据集类(
RadPathDataset)
class RadPathDataset(Dataset):
def __init__(self, df, root_data, index=None, dim=[128,128,3], ring=15):
self.transforms = transforms.Compose([ # 定义数据增强
transforms.ToTensor(),
transforms.RandomHorizontalFlip(0.5),
transforms.RandomVerticalFlip(0.5)])
def get_radiology(self, ct_image, index):
# CT图像分区域采样
for location in ['tumor', 'lymph']: # 肿瘤与淋巴结双区域处理
X_min, X_max = self.df["X_min_"+location][index] - self.ring, # 扩展采样边界
Z_1 = Z_min + int((Z_max - Z_min)/4) # 四等分深度采样
center1 = [center_Y[0], center_X[0], np.random.randint(Z_min, Z_1+1)] # 随机中心点
sub_vol1 = self.transforms(utils.random_crop(ct_image, self.dim, center1)) # 随机裁剪
数据流说明:
- 从CSV读取元数据(
df参数) - 加载
.nii.gz格式CT影像 - 使用软窗技术预处理CT图像(
soft_tissue_window) - 对肿瘤和淋巴结区域进行四层随机采样
- 加载病理学特征嵌入数据(
embeddings.npy)
5-3:模型输入特征处理
手工特征数据集(
HandCraftedFeaturesDataset)
class HandCraftedFeaturesDataset(Dataset):
def __getitem__(self, idx):
# 特征标准化与噪声添加
features_mod1 += np.random.randn(*features_mod1.shape) * self.random_noise_sigma
mean1 = np.mean(features_mod1, 0)
std1 = np.std(features_mod1, 0)
features_mod1 = (features_mod1 - mean1) / std1 # Z-score标准化
数据处理流程:
- 从DataFrame提取放射组学特征(
rad0-rad6) - 添加高斯噪声增强鲁棒性
- 执行标准化处理消除量纲差异
5-4:数据加载器配置
train_loader = DataLoader(train_set,
batch_size=1,
shuffle=True,
collate_fn=custom_collate)
参数解析
• batch_size=1:小批量适合医疗数据的高方差特性
• shuffle=True:防止医学数据顺序偏差
• collate_fn:处理非等长病理特征
六、特征提取和数据处理
6-1:CT影像边界框提取模块
build_ct_boxes函数
def build_ct_boxes(path):
mkdirs(os.path.join(path, 'ct_boxes')) # 创建存储目录
df = pd.read_csv(os.path.join(path, 'data_table.csv'))
# 遍历肿瘤/淋巴结标注文件
for label_name in ['label.nii.gz', 'node.nii.gz']:
for patient in df["radiology_folder_name"]:
label_path = os.path.join(path, "radiology", patient, label_name)
if os.path.exists(label_path):
label, _ = load(label_path) # 加载NIFTI格式标注
# 计算三维边界坐标
x_min = np.min(np.nonzero(label)[1])
...
else: # 处理缺失标注的情况
x_min, x_max = 0,0
...
- 使用示例
path = r"C:\data"
build_ct_boxes(path) # 生成ct_boxes目录及坐标文件
6-2:病理图像特征提取模块
- HIPT模型初始化
def patch_embedding(dir_data, dir_hipt_pth, device):
# 加载双阶段ViT模型
model = HIPT_4K(pretrained_weights256, pretrained_weights4k, device256, device4k)
model.eval()
# 遍历病理切片
for slide in slide_list:
image = openslide.OpenSlide(slide_path) # 打开WSI全切片图像
mask = Image.open(mask_path) # 加载肿瘤区域标注
downscale = find_downscale(image, mask) # 计算最佳下采样率
- 特征生成流程
# 处理每个图像块
for h5file in h5_files: # h5文件存储图像块坐标
with h5py.File(h5file) as f:
h5_arr = f[a_group_key][()] # 读取坐标数组
# 生成嵌入向量
for coord in h5_arr:
region = image.read_region(coord, level, size) # 提取图像块
embedding = model(eval_transforms()(region)) # 前向传播
df.iloc[i, 2:] = embedding.cpu().numpy()
6-3:特征标准化模块
- 特征片段重组
def fragment_embeddings(dir_embeddings, size):
for slide in os.listdir(dir_embeddings):
img_stack = {"x_shape": [], "y_shape": [], "img": []}
# 对齐不同尺寸的特征图
max_shape = np.max([max_x, max_y])
final_img = np.zeros((len(imgs), max_shape, max_shape, 192))
# 中心对齐填充
for i, img in enumerate(img_stack['img']):
final_img[i, (max_shape - x)//2:(max_shape - x)//2 + x,
(max_shape - y)//2:(max_shape - y)//2 + y, :] = img
np.save("embeddings.npy", final_img)
6-4:系统集成要点
- 环境配置检查表
# OpenSlide DLL路径配置(Windows特殊处理)
OPENSLIDE_PATH = r'C:\openslide-win64\bin'
if hasattr(os, 'add_dll_directory'):
with os.add_dll_directory(OPENSLIDE_PATH):
import openslide
else:
import openslide
# GPU设备检测
device = torch.device('cuda:0' if torch.cuda.is_available() else 'cpu')
- 数据目录结构规范
数据根目录/
├─ radiology/
│ └─ 患者ID/
│ ├─ CT_img.nii.gz # CT原始影像
│ ├─ label.nii.gz # 肿瘤标注
│ └─ node.nii.gz # 淋巴结标注
├─ pathology/
│ ├─ slides/ # 原始病理切片
│ ├─ tumor_annotation/ # 病理区域标注
│ └─ clam_output/ # 特征输出
│ ├─ embeddings/ # HIPT嵌入向量
│ └─ patches/ # 图像块坐标
6-5:完整执行流程示例
-
数据预处理:
build_ct_boxes(r"C:\data") # 生成CT坐标 -
病理特征提取:
patch_embedding( dir_data=r"C:\data\pathology\clam_output", dir_hipt_pth=r"C:\HIPT\Checkpoints", device=device ) -
特征标准化:
fragment_embeddings( dir_embeddings=r"C:\data\embeddings", size=2048 ) -
模型训练:
dataset = RadPathDataset(df, root_data=r"C:\data") loader = DataLoader(dataset, collate_fn=custom_collate) for batch in loader: # 多模态数据输入 ct_tumor, ct_lymph, pathology, labels = batch outputs = model(ct_tumor, ct_lymph, pathology) loss = criterion(outputs, labels) ...
七、SMuRF模型主程序
7-1:代码结构概览
├── 数据加载模块
│ ├── RadPathDataset:放射+病理联合数据集
│ ├── RadDataset:纯放射数据集
│ └── PathDataset:纯病理数据集
├── 模型架构
│ ├── FusionModelBi:双模态融合模型
│ ├── RModel:放射专用模型
│ └── PModel:病理专用模型
├── 训练流程
│ ├── one_epoch:单epoch前向过程
│ ├── train_model:完整训练循环
│ └── train_val:训练验证主入口
└── 辅助功能
├── compute_metrics:性能指标计算
└── save_results_to_mat:结果存储
7-2:核心功能解析
- 多模态数据加载器
class RadPathDataset(Dataset):
def __getitem__(self, index):
# 加载CT影像
ct_image, _ = load(os.path.join(root, "CT_img.nii.gz"))
# 软窗预处理
ct_image = utils.soft_tissue_window(ct_image)
# 双区域采样(肿瘤+淋巴结)
ct_vol = self.get_radiology(ct_image, index)
# 加载病理嵌入
pathology = np.load(embeddings_path)
return ct_tumor, ct_lymph, pathology, grade, time, event, ID
- 多任务学习机制
class MultiTaskLoss(nn.Module):
def forward(self, task_type, pred_grade, pred_hazard, true_grade, time, event):
if task_type == "grade":
return F.cross_entropy(pred_grade, true_grade.long())
elif task_type == "survival":
return cox_loss(pred_hazard, time, event)
else: # 多任务加权
return 0.5*CE_loss + 0.5*cox_loss
- 模态对齐策略
def custom_collate(data):
# 动态填充病理特征
max_sizes = (max([p.shape[0] for p in pathology]),
max([p.shape[1] for p in pathology]))
for i in range(len(pathology)):
padding = (floor(pad_2d/2), ceil(pad_2d/2), ...)
m = nn.ConstantPad3d(padding, 0)
pathology[i] = m(pathology[i])
return torch.stack(ct_tumor), torch.stack(ct_lymph), ...
7-3:使用教程
步骤1:数据准备
数据目录结构:
raptomics/
├── data/
│ ├── radiology/
│ │ └── Patient001/
│ │ ├── CT_img.nii.gz
│ │ ├── label.nii.gz
│ │ └── node.nii.gz
│ └── pathology/
│ ├── embeddings/
│ └── tumor_annotation/
步骤2:配置文件设置
# parameters.py中配置关键参数
parser.add_argument('--feature_type', type=str, default='radiology',
choices=['radiology', 'pathology', 'both'])
parser.add_argument('--task', type=str, default='multitask',
choices=['grade', 'survival', 'multitask'])
parser.add_argument('--batch_size', type=int, default=8)
parser.add_argument('--lr', type=float, default=1e-4)
步骤3:启动训练
# 纯放射模型训练
python main.py --feature_type radiology --task grade
# 多模态联合训练
python main.py --feature_type both --task multitask --mmo_loss
步骤4:结果分析
# 查看训练曲线
plt.plot(metric_logger['train']['cindex'], label='Train C-index')
plt.plot(metric_logger['val']['grad_auc'], label='Val AUC')
# 解释模型预测
with open('pred_test.pkl','rb') as f:
preds = pickle.load(f)
print(f"Test C-index: {compute_cindex(preds[3], preds[4], preds[1])}")
7-4:关键参数说明
| 参数 | 类型 | 默认值 | 说明 |
|---|---|---|---|
--ring | int | 15 | CT采样区域扩展半径(mm) |
--gamma | float | 0.5 | 模态对齐损失权重 |
--dim | list | [128,128,3] | 切片采样尺寸[height, width, channels] |
--mmo_loss | flag | False | 启用模态间最大均值差异正则 |
结束语
本期推文的内容就到这里啦,如果需要获取医学AI领域的最新发展动态,请关注小罗的推送!如需进一步深入研究,获取相关资料,欢迎加入我的知识星球!
限时优惠,通过个人微信订阅,可立减20!


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








