样本点之间的距离 首先利用欧氏距离计算距离矩阵
d<-c(1,2,5,7,9,10)
### 样本点之间的距离
### 首先利用欧氏距离计算距离矩阵
d.mat <- dist(d, method='euclidean',diag=TRUE, upper=F)
> d.mat
1 2 3 4 5 6
1 0
2 1 0
3 4 3 0
4 6 5 2 0
5 8 7 4 2 0
6 9 8 5 3 1 0
R 中用 dist(data, method=) 函数计算观测间的距离。在 dist() 函数中, diag选项决定显示距离阵时是否显示对角线元素, upper 决定显示距离阵时是否显示上三角元素, method 是距离种类 :
• euclidean 欧式距离 ;
• manhattan 曼哈顿距离 ;
• maximum 最大(上确界)距离 ;
• minkowski 闵可夫斯基距离 ;
• canberra 兰氏距离。
本例的 6 个评分取值范围相近,所以不需要标准化。如果变量取值范围差别很大,
在计算距离前一般应该把每列标准化 (scale() 函数 )
用最短距离法聚类
> res1 <- hclust(d.mat, method='single'); res1
Call:
hclust(d = d.mat, method = "single")
Cluster method : single
Distance : euclidean
Number of objects: 6
hclust() 函数中选项 method 选择系统聚类的类间距离,包括:
• single 最短距离法,
• complete 最大距离法
• average 类平均法
• median 中间距离法
• centrold 重心法
• ward Ward 法
对 hclust() 的输出结果用 plot() 画聚类树状图:

观测的左右关系不代表距离,两类相连时纵轴的高度才代表两类之间的距离,距离越大,越不应该合并到一起。
对 hclust() 的输出结果用 cutree(tree, k=) 得到每个观测的分类号,输出类号组成的向量。注意聚类方法的结果是一些分类,具体的类编号并没有意义。
> cut1 <- cutree(res1, k=2); cut1
[1] 1 1 2 2 2 2
结果 cut1 是分为两类后每个观测的类编号。
用并列盒形图验证分类:
> boxplot(t(as.matrix(d)))
用最长距离法聚类
> res2 <- hclust(d.mat, method='complete'); res2
Call:
hclust(d = d.mat, method = "complete")
Cluster method : complete
Distance : euclidean
Number of objects: 6
plot(res2)
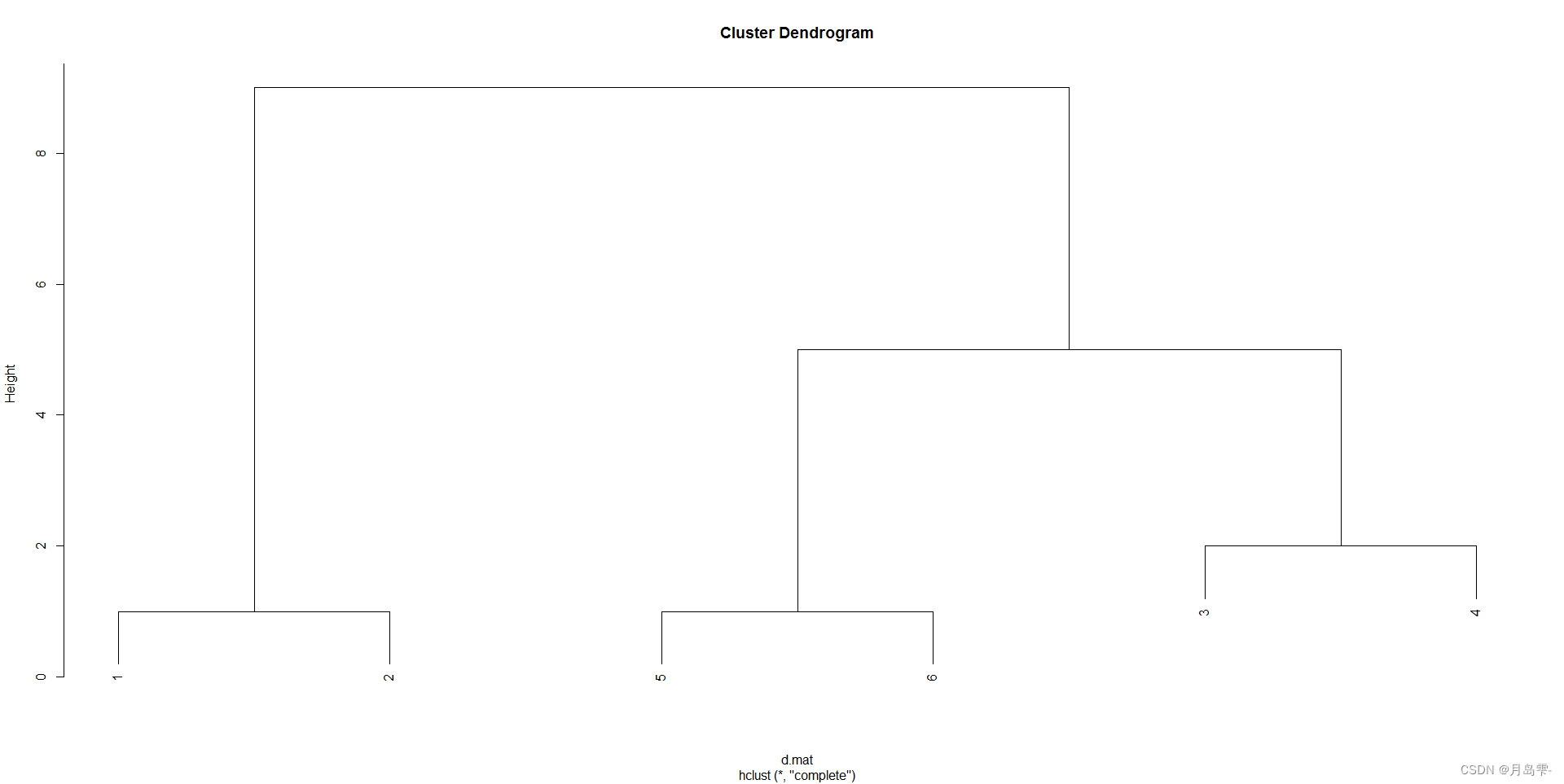
> cut2 <- cutree(res2, k=2); cut2
[1] 1 1 2 2 2 2
> cut2b <- cutree(res2, k=3); cut2b
[1] 1 1 2 2 3 3
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








