简单来说三个步骤:
1.把要作为Library包的工程和主工程放在同一个文件夹下(最好这样做,当然不这样做也行,有时不稳定)
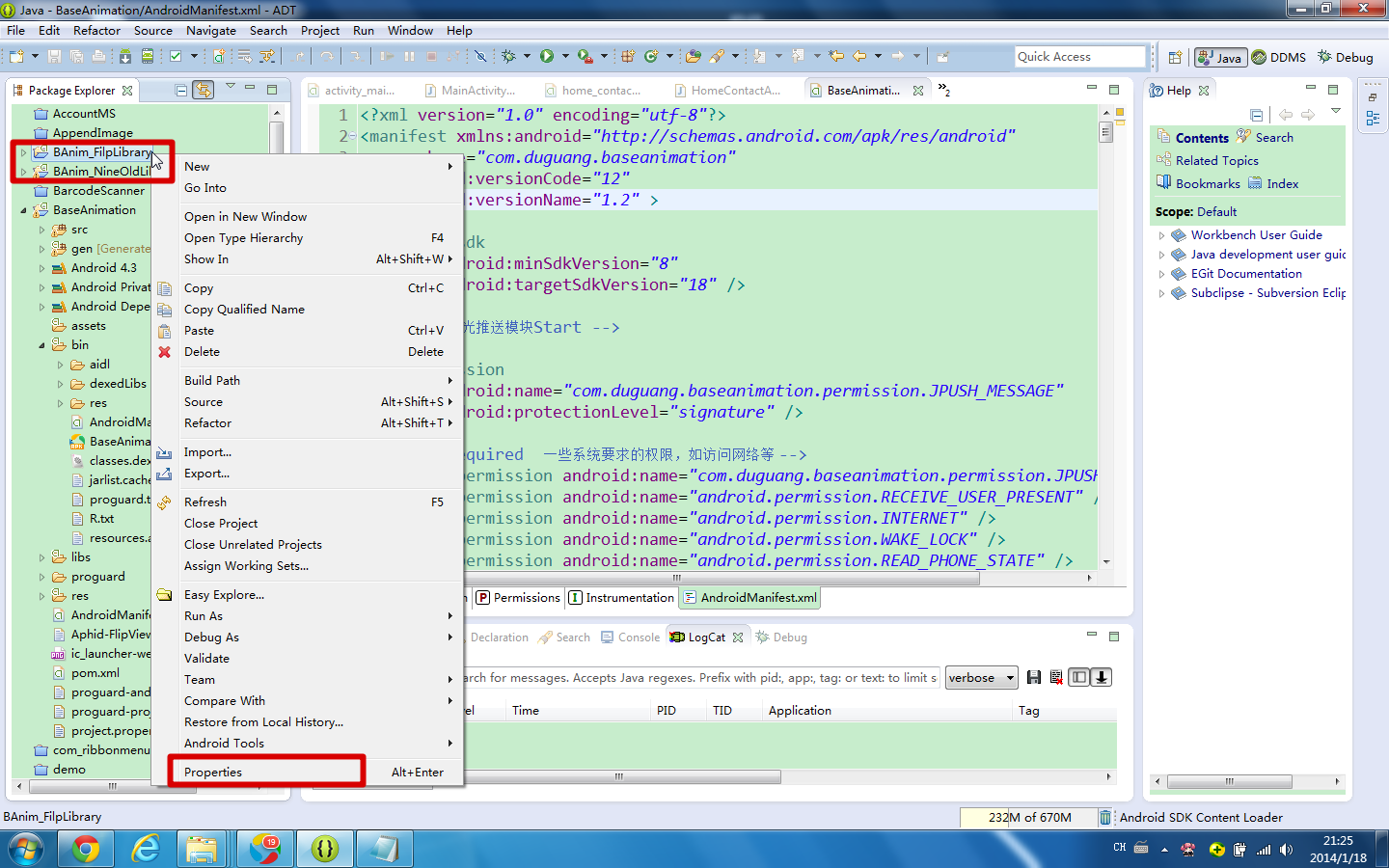
分别对BAnim_FilpLibrary , BAnim_NineOldLibrary 这两个工程进行一下操作:右键--properties
2.选择左侧Android红框中的选上对勾
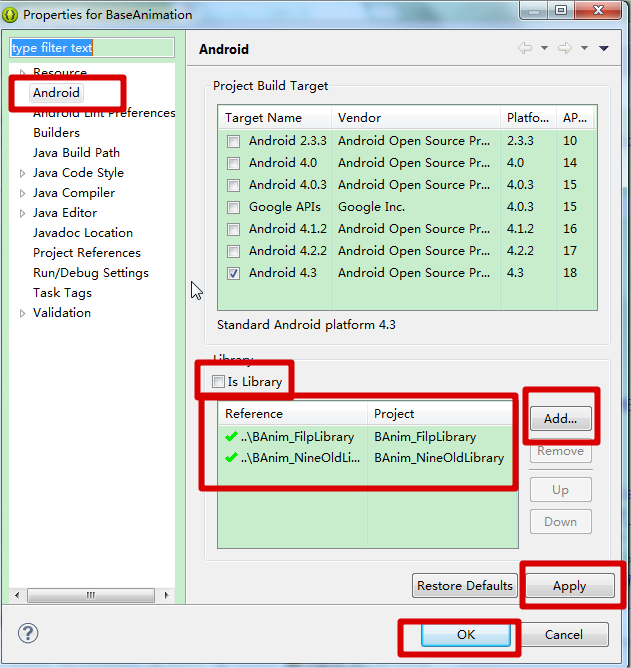
3、对主工程BaseAnimation的操作
同样右键--properties--Android--Is Library这里不要打对勾--点击Add (把BAnim_FilpLibrary , BAnim_NineOldLibrary添加进来 )--点击Appky--点击OK就行了
错误就会消除了!






















 461
461

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








