本文讨论了微生物组学研究的各个阶段,从设计实验到收集和储存样本,到最后序列数据的图形展示等,有助于研究人员考虑实验和分析中遇到的各种影响因素,指导我们更好地进行微生物组学研究。
分析微生物组的最佳做法
Best practices for analyzing microbiomes
作者:Rob Knight, Alison Vrbanac, et al.
时间:2018.5
期刊:Nature Reviews Microbiology
IF:31.851
DOI: 10.1038/s41579-018-0029-9
一、摘要
复杂的微生物群落塑造了从哺乳动物胃肠道到土壤各种环境的动态变化。与早期方法相比,DNA测序技术和数据分析的进步为微生物组分析提供了极大改进,例如,在分类学分辨率、错误发现率控制和其他特性上。在这篇综述中,作者讨论了进行微生物组学研究的最佳方法,包括实验设计、分子分析技术的选择、数据分析方法和多组学数据集的整合。最近研究结果表明,以可操作分类单元为基础的分析,应替换为基于精确的序列变异、整合宏基因组和代谢组学,以及围绕成分数据分析问题的新方法,而这些新方法的进展也非常迅速。同时,作者也指出,尽管其中一些方法是新的,但重要的是要注意在实验设计过程中出现的经典问题,并要与研究重现性相关。作者描述了如何将这些议题聚焦,使研究人员能够从他们的微生物组数据中获得更多信息。
二、主要结果
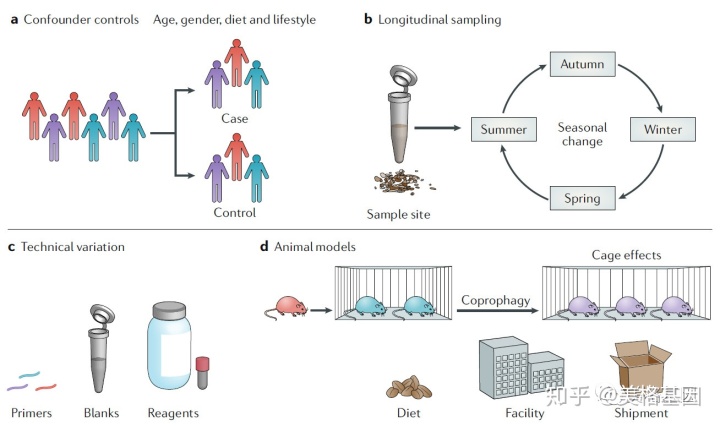
开展一个强有力的微生物组实验,需要对许多因素进行仔细考虑。





 本文深入探讨了微生物组学研究的各个方面,包括实验设计、分子技术选择、数据分析和多组学整合。作者强调了在处理微生物群落数据时考虑经典问题和重现性的重要性,并提出基于精确序列变异的新方法。文中详细阐述了如何通过分层潜在混淆因素、纵向研究、标准化技术以及元数据收集来优化研究。此外,还介绍了16S rRNA测序后的处理方法和工具,如Deblur和Kraken,以及多组学分析的应用,如三维可视化、相关网络分析和代谢活性网络建模,旨在帮助研究人员从微生物组数据中获取更多见解。
本文深入探讨了微生物组学研究的各个方面,包括实验设计、分子技术选择、数据分析和多组学整合。作者强调了在处理微生物群落数据时考虑经典问题和重现性的重要性,并提出基于精确序列变异的新方法。文中详细阐述了如何通过分层潜在混淆因素、纵向研究、标准化技术以及元数据收集来优化研究。此外,还介绍了16S rRNA测序后的处理方法和工具,如Deblur和Kraken,以及多组学分析的应用,如三维可视化、相关网络分析和代谢活性网络建模,旨在帮助研究人员从微生物组数据中获取更多见解。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








