今天推荐一个不错的绘制生存曲线的R包——ggsurvfit包,这个包最大的优点就是基于ggplot2语法来绘制生存曲线,相比survminer包的ggsurvplot()函数,ggsurvfit包的可定制化程度更高,而且绘制起来更加简单,强力推荐。
特别是组合不同的生存曲线图形,极其方便。
昨天我们学习了下怎么在图形中插入子图,今天来学习个稍微复杂点的,怎么将带有风险表的生存曲线插入到主图中。
也就是实现下面这个图形。

下面来学习下。
1. 加载数据
使用survival包的lung数据集,数据集包括了NCCTG晚期肺癌患者的生存率。
运行以下代码加载数据:
library(survival) # 加载包
head(lung) # 预览数据
2. 拟合曲线
使用survfit2()函数来拟合曲线。
library(ggsurvfit)
fit <- survfit2(Surv(time, status) ~ sex, data = lung)
fit # 可输出部分信息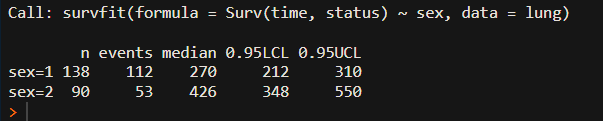
3. 简单生存曲线
将拟合模型转化为整洁数据框,然后提取其中的数据使用ggplot2包进行绘图。
ggsurvfit包将绘图代码打包成了一个函数——ggsurvfit()函数,使用ggsurvfit()函数可以轻松绘制生存曲线。
fit %>%
ggsurvfit()
4. 生存曲线排版
可以在主图上面添加带有风险表的子图。
p1 <- survfit(Surv(time, status) ~ sex, data = lung) %>%
ggsurvfit(linewidth = 0.8) +
add_risktable(risktable_stats = "n.risk") +
theme_classic(base_size = 14) +
theme(legend.position=c(0.2,0.2)) +
ggsci::scale_fill_nejm()
p2 <- survfit(Surv(time, status) ~ sex, data = lung) %>%
ggsurvfit(linewidth = 0.8, type = "cumhaz") +
add_risktable(risktable_stats = "n.risk",
risktable_height = 0.20) +
theme_classic(base_size = 10) +
theme(legend.position='none') +
ggsci::scale_fill_nejm()绘制很简单,一行代码即可搞定。
library(patchwork)
wrap_plots(ggsurvfit_build(p1)) +
inset_element(ggsurvfit_build(p2),
left = 0.50, bottom = 0.55, right = 1.0, top = 1.0)
如上所示,即绘制好了我们需要的图形。
这个R包是完全基于ggplot2语法来绘制生存曲线的,只要熟练掌握ggplot2语法,可定制化生存曲线各个不同的图形组成元件。
绘制生存曲线非常完美,最强的R包。
本公众提供各种科研服务了!
一、课程培训 2022年以来,我们召集了一批富有经验的高校专业队伍,着手举行短期统计课程培训班,包括R语言、meta分析、临床预测模型、真实世界临床研究、问卷与量表分析、医学统计与SPSS、临床试验数据分析、重复测量资料分析、nhanes、孟德尔随机化等10余门课。如果您有需求,不妨点击查看: |
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








