一、项目简介与生物学背景
Jilin_China队2020年以"IGAME"为题设计了一款以合成生物学为基础的生命游戏。其利用在player中植入对光敏感的基因来创造出在不同光照下有不同状态(攻击/防守)的大肠杆菌。
在游戏中玩家通过不同颜色的光来控制“Player”和“Enemy”进行对抗,“Enemy”持续进行攻击,蓝光照射后“Player”开始防御,红光照射后“Player”开始反击,最终通过KNN算法计算菌的数目判别胜负【1】。
二、数学建模及计算机技术
Model 1--KNN算法
(1)算法简介:
KNN又叫邻近算法,或K最近邻(kNN,k-NearestNeighbor)分类算法,它是数据挖掘分类技术中最简单的方法之一。所谓K最近邻,就是k个最近的邻居的意思,说的是每个样本都可以用它最接近的k个邻居来代表。
KNN算法的核心思想是如果一个样本在特征空间中的k个最相邻的样本中的大多数属于某一个类别,则该样本也属于这个类别,并具有这个类别上样本的特性。
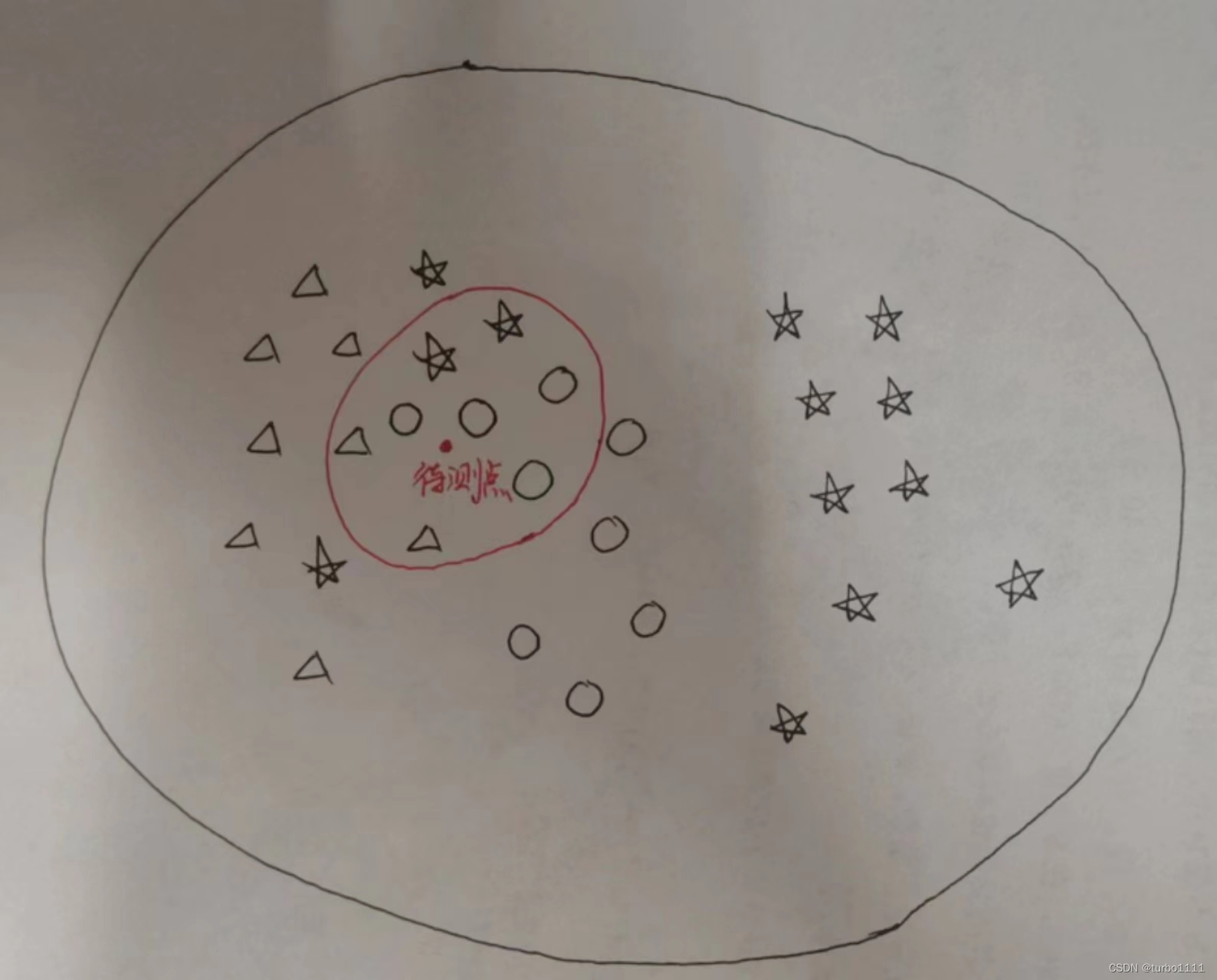
如上图,待测点要分在哪一类呢?我们查看离它最近的K(这里K=8)个样本点,在这八个样本点中有四个⚪,三个▲,两个⭐,⚪的个数最多,则待测点分在⚪这一类中。
样本点间距离计算公式:
1、超参数距离:
![[外链图片转存失败(img-t3USjKMW-1565944939062)(C:\Users\爱拼才会赢\Desktop\python\神经网络基础及架构初步\图片\8.jpg)]](https://i-blog.csdnimg.cn/blog_migrate/bf47d00862be07cf893032d77fac9fac.jpeg)
2、欧氏距离:
![[外链图片转存失败(img-OTUstmw6-1565944939062)(C:\Users\爱拼才会赢\Desktop\python\神经网络基础及架构初步\图片\9.jpg)]](https://i-blog.csdnimg.cn/blog_migrate/2659d611a87cbec9381905a47b4fe236.jpeg)
3、曼哈顿距离:

(2)在项目中的作用:
采用KNN算法来模拟平板上菌落数量的分布,并以二进制图片的形式呈现,以快速评估细菌间的博弈结果。如果最后玩家的领土大于敌人的领土,则玩家获胜。如下图所示(图源网站:Team:Jilin China - 2020.igem.org)

(3)代码实现:
##KNN算法代码实现
##语言:python
##日期:2023.03.22
##创作:turbo1111
import numpy as np
import operator
def knn_classify(inputX, data, labels, k):
# 1.计算测试数据与各训练数据之间的距离。
dataSize = data.shape[0]#12
x = np.tile(inputX, (dataSize, 1)) - data#计算待测点与样本点的差值(x1-x2)
xPositive = x ** 2#差值的平方((x1-x2)^2)
xDistances = xPositive.sum(axis=1)#求和
distances = np.sqrt(xDistances)#计算距离
# 2.按照距离的大小进行排序。
sortDisIndex = distances.argsort()
# 3.选择其中距离最小的k个样本点。4.确定K个样本点所在类别的出现频率。
classCount = {} # 创建字典:label为键,频数为值
for i in range(k):#循环k次,取出k个距离最小的样本点,获取他的类别,计算数目
getLabel = labels[sortDisIndex[i]]
classCount[getLabel] = classCount.get(getLabel, 0) + 1
# 5.返回K个样本点中出现频率最高的类别作为最终的预测分类。
sortClass = sorted(classCount.items(), key=operator.itemgetter(1), reverse=True)
print(sortClass[0][0])
return sortClass[0][0]
if __name__ == '__main__':
# 测试数据
inputX = np.array([1,0.9,1])
# 训练数据
data = np.array([[1, 0.9, 1], [0.8, 0.9, 0.7], [1.3, 1, 1.2], [1.2, 0.9, 1], [2, 2.2, 2.1], [2.3, 2.2, 2],
[2, 2.2, 1.9], [1.9, 2.2, 2.1], [3.1, 3.1, 3], [2.8, 2.9, 3.1], [2.9, 3, 3.2], [3.1, 3, 3.1]])
# 训练数据标签
labels = np.array([1, 1, 1, 2, 2, 2, 2, 2, 3, 4, 3, 4])
k = 4
knn_classify(inputX, data, labels, k)
Model 2--ODE(常微分方程分析)
(1)预备知识:
米氏方程:
米氏方程(Michaelis-Menten equation)是表示一个酶促反应的起始速度与底物浓度关系的速度方程。
在酶促反应中,在低浓度底物情况下,反应相对于底物是一级反应(与浓度的大小成正比);而当底物浓度处于中间范围时,反应(相对于底物)是混合级反应(一级反应与零级反应的混合)。当底物浓度增加时,反应由一级反应向零级反应(生成速度与酶的浓度无关,表示酶已经过量了)过渡。
Hill方程(希尔方程):
希尔方程是一个线性二阶常微分方程。

其中,J(z)是一个周期为pai的偶函数,可做傅里叶级数展开:

(2)项目中的ODE推导:
1、enemy攻击player
enemy通过分泌木聚糖酶(xylanase)到细胞外水解木聚糖,水解产物是阿拉伯糖(ara),阿拉伯糖可以进入player细胞中,与Arac相结合,改变其构象。
Arac会进一步结合pBAD基因,激活relE的表达,产生ReLE(一中mRNA酶),使得player自身的mRNA裂解从而抑制生长而死亡。
常微分方程公式推导如下:

2、红光下,player攻击enemy
在红光照射下,player内部的群体感应系统相关的基因--luxl基因被激活,其表达出的产物--AHL会与enemy的luxR基因表达出的luxR蛋白结合,从而激活prLUX基因(个人理解为luxR蛋白会抑制prLUX基因的表达),使得enemy的relE基因表达,产生ReLE裂解enemy的RNA,从而杀死enemy。
常微分方程推导如下:

3、蓝光下,player进行防御:
蓝光照射下,player内的抗毒素基因开始表达,其表达的产物RelB能与体内的RelE相结合,抑制RelE的RNA裂解作用。
具体微分方程推导如下:
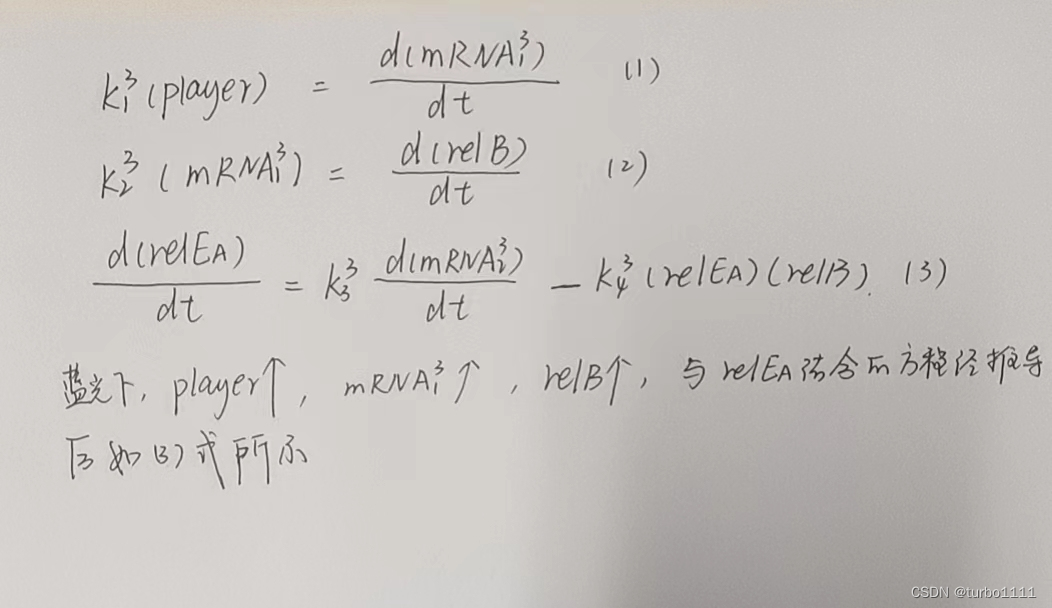
本文引用博客/推文:
【1】Jilin_China|iGEM项目分析 (qq.com)
【3】Team:Jilin China - 2020.igem.org
本文如有疏漏、错误之处,欢迎指正。







 文章介绍了Jilin_China团队2020年设计的一款基于合成生物学的“IGAME”游戏,玩家通过光控大肠杆菌的攻击与防御。游戏中应用了KNN算法来模拟菌落分布,以Python实现。同时,文章还探讨了常微分方程在模拟生物反应过程中的应用,如酶促反应和群体感应机制。
文章介绍了Jilin_China团队2020年设计的一款基于合成生物学的“IGAME”游戏,玩家通过光控大肠杆菌的攻击与防御。游戏中应用了KNN算法来模拟菌落分布,以Python实现。同时,文章还探讨了常微分方程在模拟生物反应过程中的应用,如酶促反应和群体感应机制。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








