重点
使用ggplot2包构建富集气泡图
能够分别抽取三个不同过程的前10个数据并用rbind()合并数据框
pp <- ggplot(data = slimgo,mapping = aes(x=Term,y=Percentage,fill=Ontology))#数据来源是slimgo,aes是映射,将Term的数据映射到x轴,映射之前先将变量转换为因子,Ontology映射为填充色
geom_bar(stat=“identity”)#在数据框已有计数的条件下创建条形图
coord_flip()翻转坐标轴
cale_x_discrete(limits=rev(slimgo$Term))改变过程内的排序顺序
guides(fill='none')去掉图例
theme_bw()切换黑白背景为主题
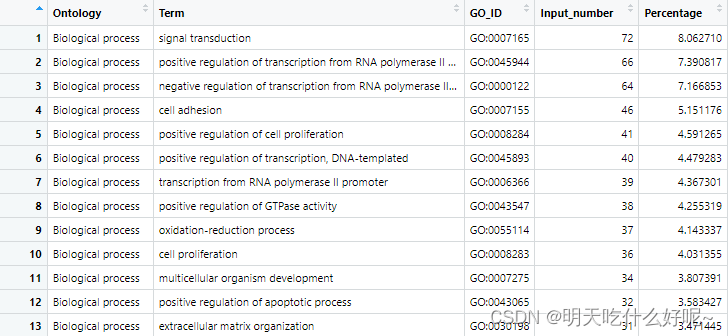
数据概览
代码
#GO注释图
go <- read.csv(file.choose(),header=T)#go.csv
head(go)
View(go)
#绘制GO富集分析图
library(ggplot2)
#对数据框进行排序
go_sort <- go[order(go$Ontology,-go$Percentage),]#对列进行排序要加逗号,加负号降序排列
head(go_sort)
#取前十行最显著的进行画图
#分子功能
m <- go_sort[go_sort$Ontology=="Biological process",][1:10,]
c <- go_sort[go_sort$Ontology=="Cellular component",][1:10,]
b <- go_sort[go_sort$Ontology=="Molecular function",][1:10,]
slimgo <- rbind(b,c,m)#使用rbind进行合并
slimgo
View(slimgo)
#将Term列转化为因子
slimgo$Term <- factor(slimgo$Term,levels = slimgo$Term)
colnames(slimgo)
pp <- ggplot(data = slimgo,mapping = aes(x=Term,y=Percentage,fill=Ontology))#数据来源是slimgo,aes是映射,将Term的数据映射到x轴,Ontology映射为填充色
pp
#
pp+geom_bar(stat = "identity")#geom_bar进行填充
pp+geom_bar(stat = "identity")+coord_flip()#geom_bar进行填充,coord_flip翻转坐标轴
#scale_x_discrete(limits=rev(slimgo$Term))改变过程内的排序顺序
pp+geom_bar(stat = "identity")+coord_flip()+scale_x_discrete(limits=rev(slimgo$Term))
#guides(fill='none')去掉图例
pp+geom_bar(stat = "identity")+coord_flip()+scale_x_discrete(limits=rev(slimgo$Term))+guides(fill='none')
#theme_bw()切换黑白背景为主题
pp+geom_bar(stat = "identity")+coord_flip()+scale_x_discrete(limits=rev(slimgo$Term))+guides(fill='none')+theme_bw()























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








