前言:这是学校多元统计分析课程布置的实验(包括基于python的线性代数运算、线性回归分析实验、聚类分析、因子分析和主成分分析),这里分享出来,注解标注的比较全,供大家参考。
1、为比较10种红葡萄酒的质量,由5名品酒师对每种酒的颜色、香味、甜度、纯度和果味6项指标进行打分,最低分1分,最高分为10分,得到每种酒的每项指标的平均得分,数据见文件“test3-1.csv”。完成以下内容。
① 使用SPSS软件对数据采用系统聚类法进行样本和变量聚类分析,系统聚类法实现最小距离、最大距离、重心距离、类平均距离四种中的2种,同时距离的衡量实现平均欧式距离、欧式距离、绝对距离、切贝谢夫距离、明可夫斯基距离、person相关系数、夹角余弦的2种;
Ⅰ.最小距离+平均欧式距离:


Ⅱ.最大距离+欧式距离:


② 用python语言编程实现系统聚类法分析,并运行上述数据,结果与第1步骤中的结果进行比对。
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import scipy.cluster.hierarchy as sch
from scipy.cluster.hierarchy import dendrogram, set_link_color_palette
# 显示中文标签
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
df = pd.read_csv("F:/成信大/多元统计分析/test3-1.csv")
data = df.columns[1: 7]
# 存放元素
u = df[data]
# 获取第一列数据,便于最后生成图的纵坐标下标从1开始而不是0
x = df.columns[0]
index = np.array(df[x])
'''
具体参考官方文档:
https://docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.linkage.html
'''
# 聚类方法:最远邻元素;测量区间:欧式距离
# 聚类方法:最近邻元素;测量区间:明可夫斯基
result = sch.linkage(u, method='single', metric='minkowski', optimal_ordering=True)
dn = dendrogram(result, orientation='right', labels=index)
plt.show()
运行结果:

2、美国50个州的8个变量的数据.变量包括人口、人均收入、文盲率、预期寿命、每10万人的谋杀和非疏忽杀人数、高中毕业百分比、温度降至零下的平均天数、土地面积。用这组数据来看美国50个州的集群现象,数据见“test3-2.csv”。完成以下内容。
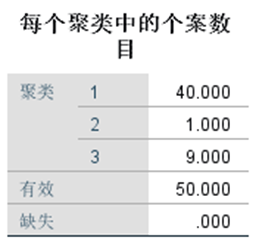
① 使用SPSS软件对数据采用K-mean聚类法进行样本聚类分析;



②用python语言编程实现数据的Z值标准化,然后利用K-mean聚类法分析,运行上述数据,输出聚类结果,并与第1步骤中的结果进行比对。
import numpy as np
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
import matplotlib.pyplot as plt
import pandas as pd
import scipy.cluster.hierarchy as sch
from scipy.cluster.hierarchy import dendrogram, linkage, set_link_color_palette
import seaborn as sns
from sklearn import preprocessing
df = pd.read_csv("test3-2.csv")
data = df.columns[1: 9]
# 获取第一列数据,方便后面分组
place = np.mat(df[df.columns[0:1]])
u = df[data]
# Z-Score数据标准化处理
x_scaled = preprocessing.scale(u)
'''
热力图
sns.set()
cor = df.corr()
sns.heatmap(cor, square=True)
plt.show()
'''
# 聚类结果图
ZT = linkage(np.transpose(x_scaled), 'ward')
dendrogram(ZT, labels=u.columns, orientation='right')
my_palette = plt.cm.get_cmap("Accent", 4)
plt.show()
# K-mean聚类分析
cls = KMeans(3).fit(x_scaled)
# 获取具体数据
cls = cls.labels_
# 分组
groups = [[], [], []]
j = 0
# 通过KMeans获得的数据结合place,将数据分为3组
for i in cls:
if i == 1:
groups[i].append(place[j])
elif i == 2:
groups[i].append(place[j])
else:
groups[i].append(place[j])
j = j + 1
for element in enumerate(groups):
print(len(element[1]), element)

首先通过python将数据进行Z值标准化后的K-mean聚类分析结果来看,3个分组聚类情况个数上与SPSS上的不一致。从SPSS的结果来看显然有一个聚类中只有1个属性。而通过python实现聚类分析结果基本在24,14,12之间,多次运行结果略有浮动。
获取最优K值
import pandas as pd
import numpy as np
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from kam import readfile
df = readfile()
# 获取矩阵行数
line = df.shape[0]
# 获取矩阵列数
col = df.shape[1]
data = []
# 行数
for i in range(df.shape[0]):
if i == 0:
continue
else:
data.append(df.iloc[i])
featureList = df.columns[1: col]
mdl = pd.DataFrame.from_records(data, columns=featureList)
# 存放每次结果的误差平方和
SSE = []
for k in range(1, col):
# 构造聚类器
estimator = KMeans(n_clusters=k)
estimator.fit(
np.array(mdl[featureList]))
SSE.append(estimator.inertia_)
# X取决于元素个数
X = range(1, col)
# x轴
plt.xlabel('k')
# y轴
plt.ylabel('SSE')
# '利用SSE选择k'
plt.plot(X, SSE, 'o-')
plt.show()























 2917
2917











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








