介绍
机器学习工具在生物研究中的应用日益广泛,这得益于预训练的大型语言模型的出现。然而,训练出有效的模型仍是一项复杂的任务,因为众多因素都会影响其性能。自动机器学习(AutoML)方法通过简化整个模型开发流程来应对这些挑战。
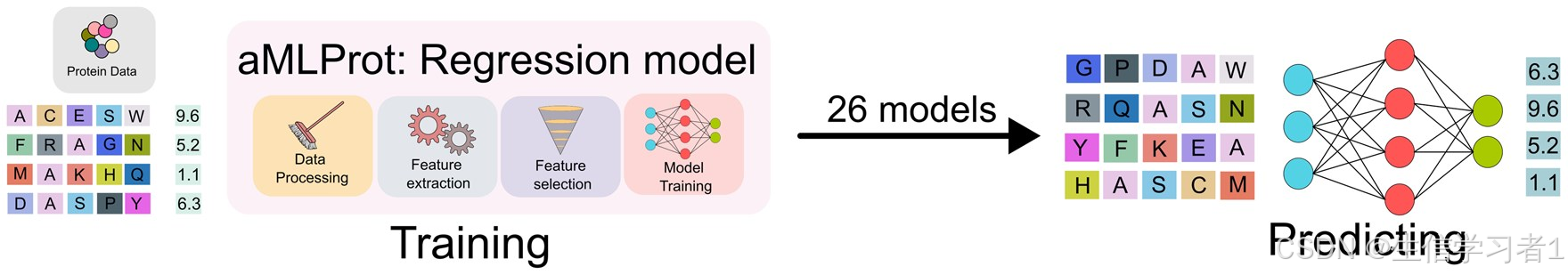
我们开发了 aMLProt,这是一个专为蛋白质应用(如酶工程和生物勘探)而设计的自动机器学习框架。它采用了模块化设计,使得每个组件能够独立使用或组合使用。值得注意的是,aMLProt 集成了 19 种分类器和 26 种回归器,以及预训练的蛋白质语言模型。它还包含经过验证对蛋白质相关工作流程有用的独立应用程序。为了提高易用性,aMLProt 与 Horus 集成,Horus 是一个基于图形用户界面的应用程序,具有直观的界面。
aMLProt 可在以下网址获取:https://github.com/etiur/aMLProt.git 和 https://doi.org/10.5281/zenodo.14971157;aMLProt 插件可通过官方 Horus 插件库 https://horus.bsc.es/repo/plugins/amlprot 获取,而 Horus 本身可从 https://horus.bsc.es 免费下载。此外,无需事先注册或下载,即可在 horus.bsc.es/amlprot 和 horus.bsc.es/amlprot-suggest 网站上找到 aMLProt 的演示版本。pH 最优值回归模型的结果和数据可在以下网址获取:https://zenodo.org/records/15394097。
Machine learning tools have become increasingly common in biological research, driven by the emergence of pre-trained large language models. However, training effective models remains a complex task, since many choices influence their performance. AutoML (automated machine learning) approaches help address these challenges by streamlining the entire model development pipeline.
We developed aMLProt, an AutoML framework tailored specifically for protein applications, such as enzyme engineering and bioprospecting. It features a modular design, allowing each component to be used independently or in combination. Notably, aMLProt integrates 19 classifiers and 26 regressors, along with pre-trained protein language models. It also includes standalone applications proven useful for protein-related workflows. To enhance usability, aMLProt is integrated with Horus, a GUI-based application with a visual interface.
aMLProt is available on https://github.com/etiur/aMLProt.git and https://doi.org/10.5281/zenodo.14971157; The aMLProt plugin is available via the official Horus Plugin Repository https://horus.bsc.es/repo/plugins/amlprot, and Horus itself can be freely downloaded from https://horus.bsc.es. Moreover, a demo of aMLProt can be found, without previous registration or download, at the horus.bsc.es/amlprot and horus.bsc.es/amlprot-suggest. The results and data from the pH optima regression model are available at: https://zenodo.org/records/15394097.

生物数据集的规模和复杂性正在急剧增加。如果能够进行有效分析,这些数据集能够为研究提供有价值的见解。机器学习在这方面表现出色,它能够从训练数据中识别出复杂的关联,并将其应用于对新样本进行预测。
尽管机器学习工具通过开源平台和易于使用的库变得更加用户友好,但开发有效的模型仍然颇具挑战性。在模型架构和训练方面的关键决策会对性能产生重大影响,而这些决策往往需要具备的专业知识,而许多研究人员并不具备这种能力。即使对于经验丰富的从业者来说,选择最佳算法并调整超参数也是一项复杂的工作,而且通常只能带来微小的改进(瓦莱里等人,2023 年)。
此外,将生物数据随机分为训练集和测试集也是存在问题的。蛋白质和其他生物样本往往具有共同的进化相似性或同源性,这可能会导致测试结果过于乐观。模型可能会学会在测试集中预测同源序列的相似性质,从而导致过拟合,并且无法很好地将新数据(与训练集的同源性较弱)的预测能力推广到其他数据上(琼斯 2019 年)。
解决这一问题的一种方法是将预测范围限定在与训练集数据相似的数据范围内,这一概念被称为适用性域(Kaneko,2024)。然而,这样做会极大地限制模型的实用性。
为了克服这些挑战并使机器学习的构建过程更加易于操作,尤其是对于非专业人士而言,一个能够简化整个训练流程且需要用户干预最少的自动化机器学习(AutoML)系统将是理想的选择。尽管存在多种 AutoML 实现方案,但其中许多并不适用于生物数据,例如蛋白质序列(Feurer 等人,2022 年)。有些则专门针对特定任务,如功能预测(de Oliveira 等人,2024 年),或者只专注于深度学习模型。例如,AutoProteinEngine(Liu 等人,2024b)允许用户通过自然语言与 AutoML 平台进行交互,从而降低了没有计算背景的用户应用机器学习工具的门槛。然而,较简单的模型,尤其是基于树的模型,能够在计算成本仅为其数分之一的情况下提供相当的性能表现,这对于处理小型实验数据集尤为重要。BioAutoMATED 试图填补一些这些差距,它将多个 AutoML 框架与对生物序列数据(如蛋白质、DNA 和 RNA)的支持相结合。它还提供了一个用户友好的 Jupyter Notebook 界面。然而,它不支持利用预训练模型,因为每个流程都必须从头开始训练。这是一个限制,因为诸如 ESM 系列这样的模型已经在一系列与蛋白质相关的基准测试中取得了最先进的性能(徐等人,2022 年)。
考虑到这一点,我们开发了 aMLProt,这是一个用于蛋白质应用(如生物勘探和工程)的端到端自动机器学习平台。基于我们之前开发 EP-pred(Xiang 等人,2022 年)的经验,aMLProt 采用了模块化结构,其中每个模块代表机器学习流程中的关键步骤。这些步骤包括数据预处理和划分、特征提取和选择、异常检测以及模型选择和优化(包括预训练的大规模语言模型)(图 1)。这种模块化结构允许用户独立使用每个模块块,或者将它们组合成自定义工作流程,从而提供了高度的灵活性。aMLProt 的另一个关键重点是通过将现有的机器学习工具集成到内置应用程序中来提高其可用性。第一个实现的工具使用了已被证明能够预测突变功能影响的蛋白质语言模型(Meier 等人,2021 年)。例如,ESM 模型提出的突变使一种成熟抗体的结合亲和力增加了七倍(Hie 等人,2024 年),这证明了其在蛋白质优化方面的实际实用性。
代码
https://github.com/etiur/aMLProt.git
参考
- aMLProt: an automated machine learning library for protein applications
- https://github.com/etiur/aMLProt.git























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










