新型冠状病毒传播规律离散微观模型
0 前言
大年三十,全家自我隔离,各路关于新型冠状病毒的消息刷屏,有喜有忧。老裴完全没有心思过年,也无法工作,但自己又无能为力。老裴一直以科学计算工作者自诩,希望能做点什么。跟有相关工作经验的同学交流得知,目前的模型都是基于连续的常微分方程,该种模型理论性强、模型简单,但不够直观。于是老裴设计了一个离散的微观模型,通过实例表明,所有人应果断拥护和执行ZF的相关决定,
1、毫不犹豫实行自我隔离
2、发现不适症状及时就医
由于老裴水平有限,不当之处请多指教。
1、模型
所提出模型包括两个部分,分别为个体病毒感染概率计算模型和个体移动模型。
1.1 个体病毒感染概率计算模型
用平面坐标系上的点P(xi,yi)描述个体,假设任意两个体P(xi,yi)和Q(xj,yj)之间的距离为dij,如果Q是病毒携带者,Q向P传播病毒并使P感染的几率为pij,二者的关系用式(1)描述:
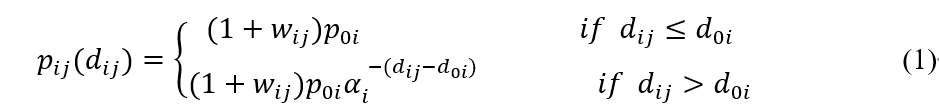
其中:
d0i为个体P(xi,yi)的临界距离;
wij反映个体P和Q的亲密度,其值取0~1;
p0i为wij=0时P在临界距离d0i内被感染的几率,也称基础感染几率;
αi为大于1的系数。
个体的临界距离d0i大小以及其在临界距离内被感染的几率r0i反映了个体自身的抵抗能力,wij反映个体之间的亲密度指数,αi反映影响范围。
需要注意的是,此处所指的距离是广义距离,并不是指现实中两个个体的实际距离,可以认为是两个体多方面的综合距离,包括个体之间的时空关系、亲密度关系、抵抗能力等,同时pij (dij)≠pji (dij)。
以上公式反应了个体距离与被感染病毒几率之间的关系,如图1所示为具体实例(p0i=0.05,d0i=5,αi=2,wij=0和0.5)。

该模型假设个体在临界范围d_0i内的感染几率是一定的,超过该范围后感染几率将逐渐减小,达到一定值后,感染几率迅速减小为0。个体的亲密度越高,感染几率越大。
1.2 个体移动模型
假设任意个体P(xi,yi)在平面内随机游走(Random Walk),用个体的移动步长dstep来描述个体的活跃度,值越大表明越活跃。随机游走相关知识这里不再赘述,请读者自行查阅相关资料。
初始时刻,假设每个个体之间的距离为固定值df,且满足p(df )≈0。如图2所示。

1.3 求解过程
整个求解过程如下:
Step 1:确定模型参数,主要包括:
- 个体数量NI;
- 个体初始化距离df;
- 个体临界距离矩阵
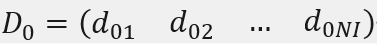
- 个体在临界距离内被携带者感染的0亲密度概率矩阵,也称为基础感染几率矩阵
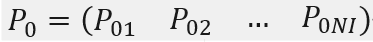
- 每个个体与其他个体的亲密度矩阵
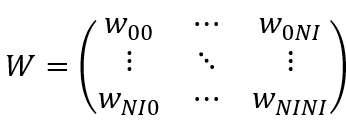
- 个体的概率计算模型底数矩阵
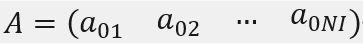
- 随机游走时间步长tstep;
- 和移动距离步长矩阵dstep;
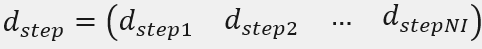
- 个体游走持续时间t1.
Step 2:初始化个体位置;
Step 3:放置传染源个体;
Step 4:在每个时间步长tstep内更新个体位置并根据感染概率模型更新被感染的个体,直到达到时间t1。
2 实例分析
接下来将逐一模拟多种不同的情况。需要注意的是,由于模型中很多参数的随机性,如果要达到较好的结果,可能需要不断的调整参数,做一个快乐的调参侠。
2.1 正常活动传播模拟
该种情形模拟的是病毒发展初期,已有人感染了病毒,但未引起重视,包括感染者在内,所有人正常活动。通过设置不同的参数观察传播过程。
基本参数
N = 20
NI = N*N #个体数量为400
D0 = 2. #临界距离
P0 = 0.3 #个体基础感染几率
W = 0 #亲密度
df = 6. #初始距离
t_step = 0.01 #时间步长
d_step = 0.05 #移动步长
m = Diffusion(N)
m.init_pos(df) #初始化位置
m.init_para(D0,P0,W,t_step = t_step,d_step = d_step) #模型参数设置
m.place_source(153) #放置感染源个体
for t in[1,2,3,4,5,6,7]:
m.walk(t)
m.plot()
接下来的分析是在以上参数的基础上进行修改。
以上模型个体数量为400,每个个体的临界距离d0相同,基础感染几率p0=0.3也相同,所有个体之间亲密度w=0,每个个体的移动步长也相等,默认感染概率模型底数为e。
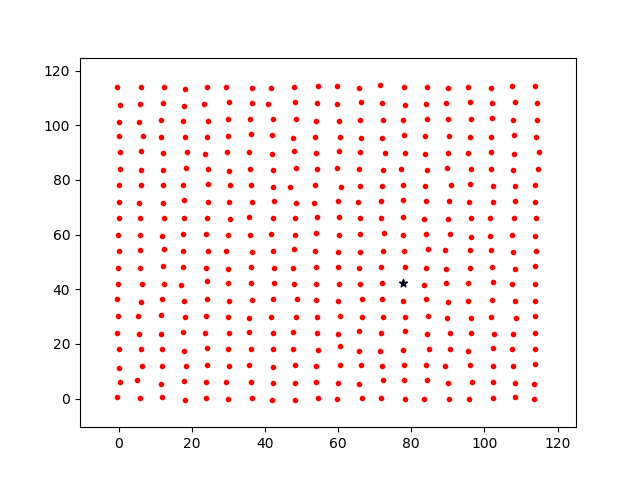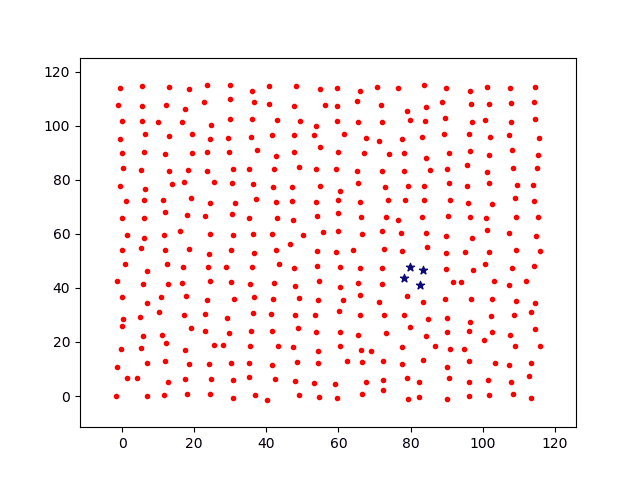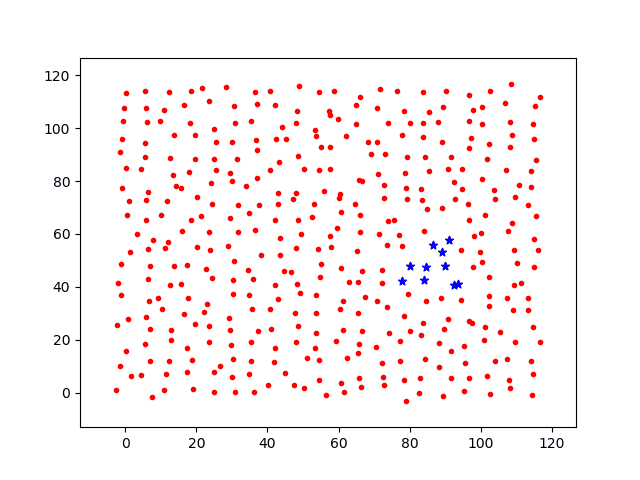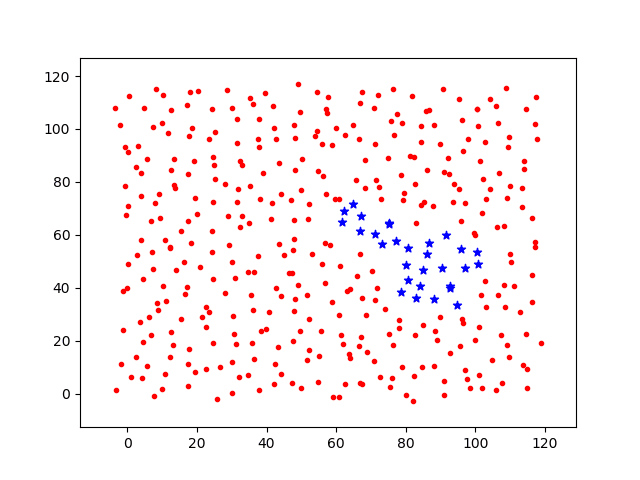
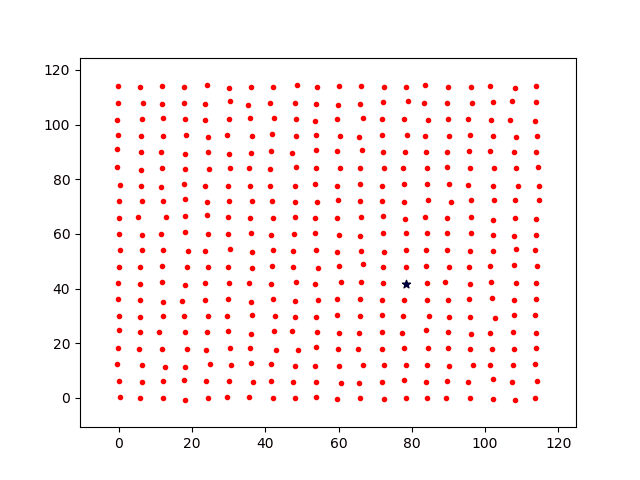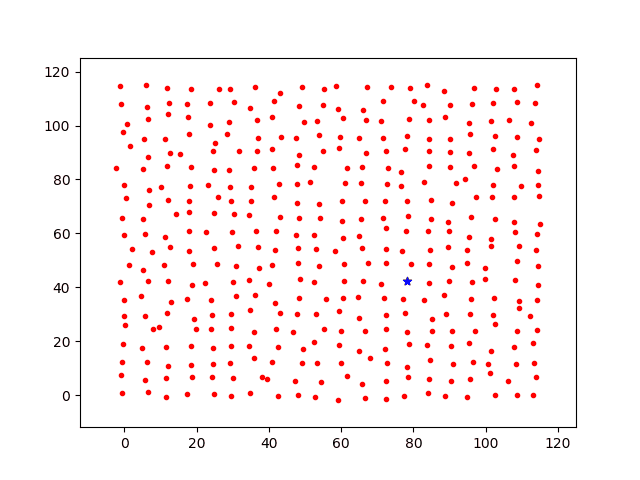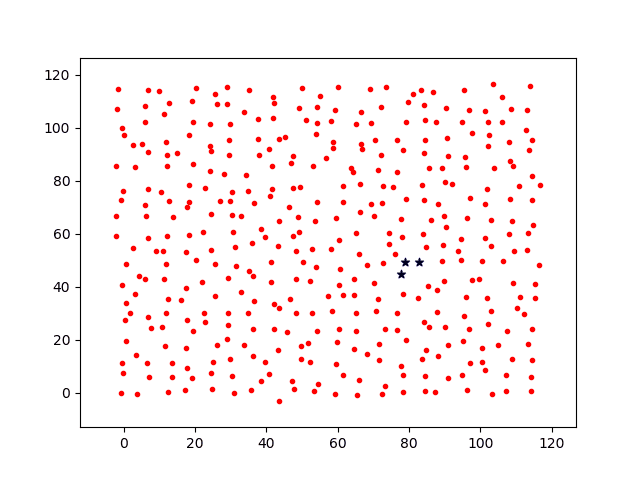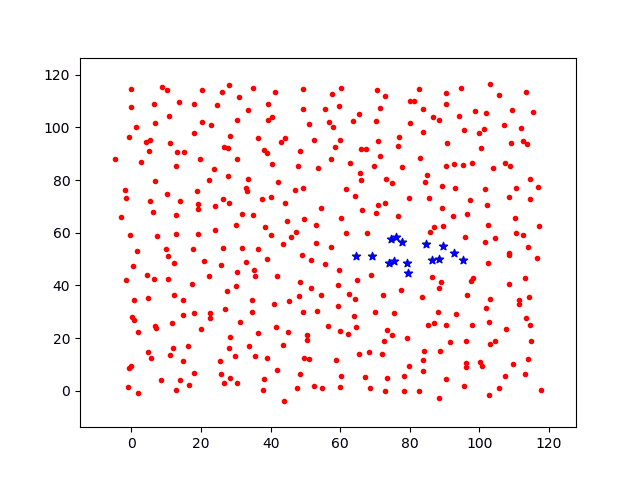
分别计算移动步长dstep = 0.05和dstep = 0.1,基础感染几率p0=0.3,投放1个病源时间增量t=[1,2,3,4,5,6,7]各节点上感染过程图如下。
其中:
红点代表未感染个体;
蓝色星星为被感染个体。
绿点为隔离个体。
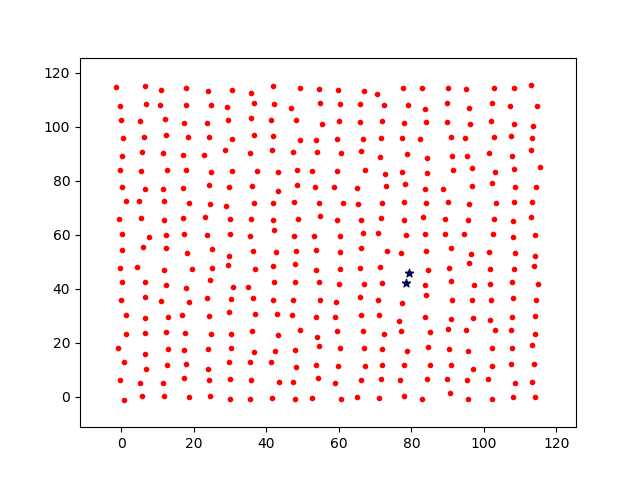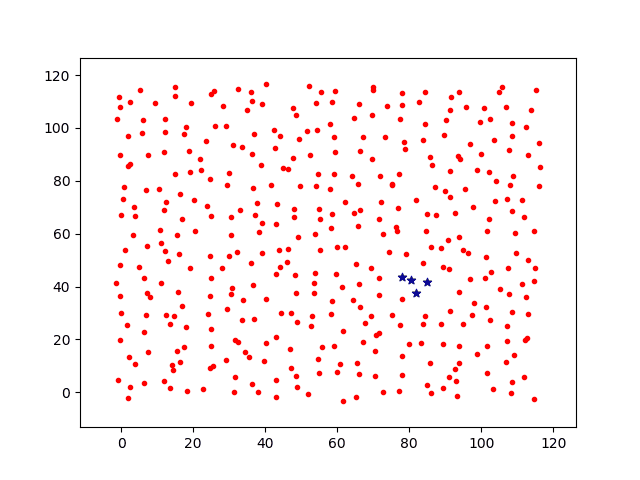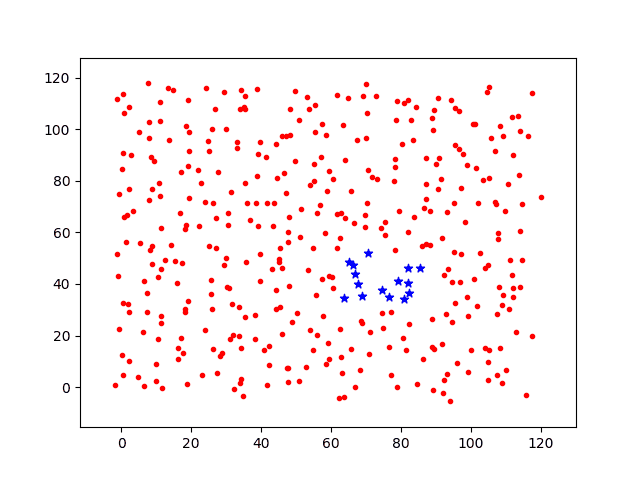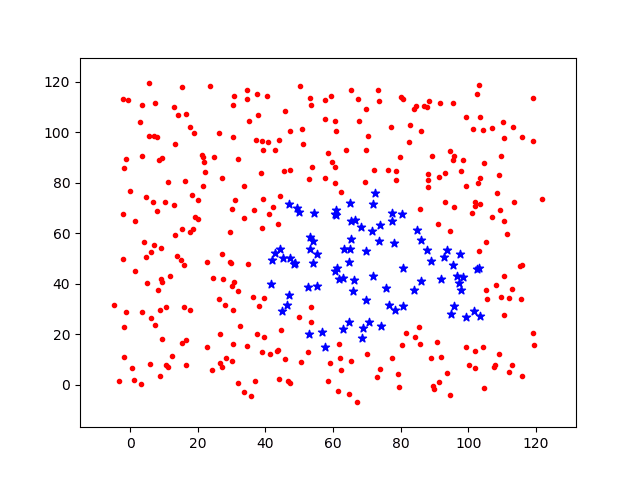
接下来,将基础感染几率降低为p0=0.05,相同时间节点上的感染过程图如下
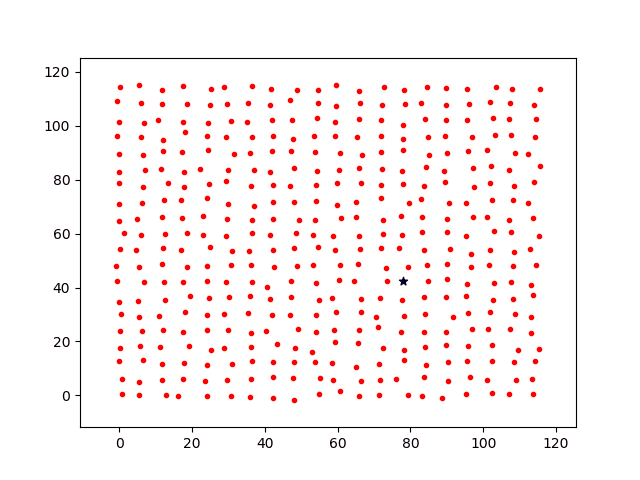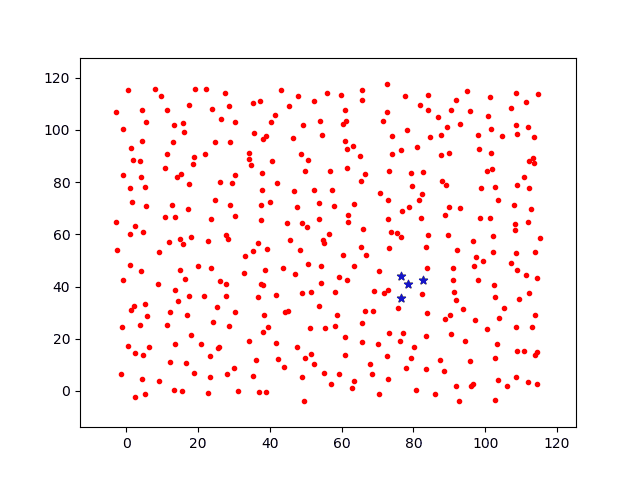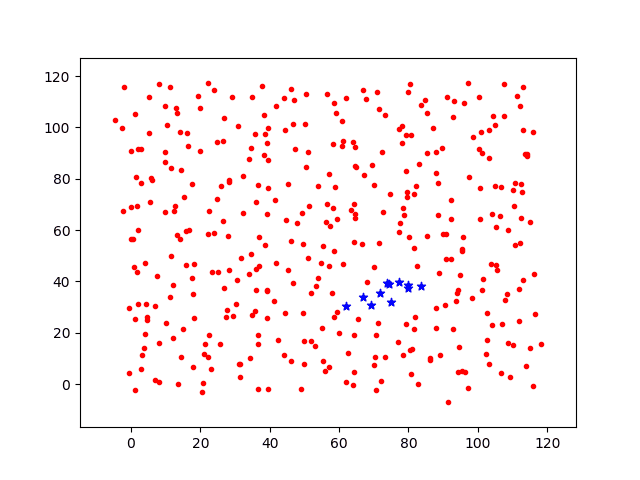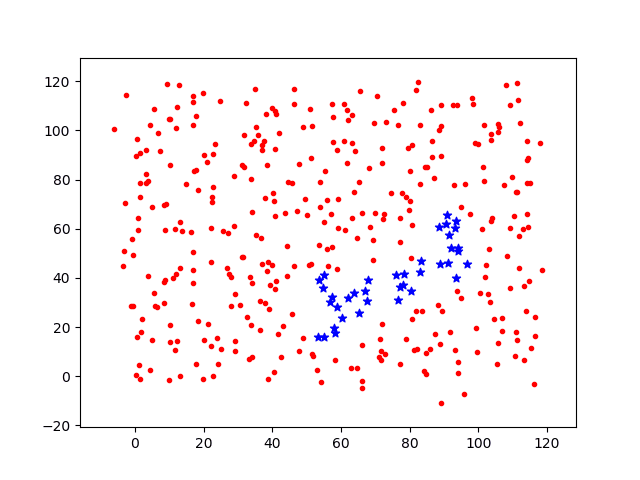
将不同参数下各时间点被感染总数回执在下图中

可以看到,时间t=5内感染总数基本一致,随着时间的增加,高基础感染率p0=0.3和高移动步长dstep = 0.1增长趋势更快。
这也意味着,早期的扩散增长与参数大小无太大的关系,及时发现及时隔离
2.2 隔离模拟
现在将感染源附近的进行自我隔离,及令其活动步长dstep=0(绿点),基础感染率仍设为p0=0.3,其他个体的基础感染率仍为p0=0.3。移动步长dstep = 0.1。感染过程图如下
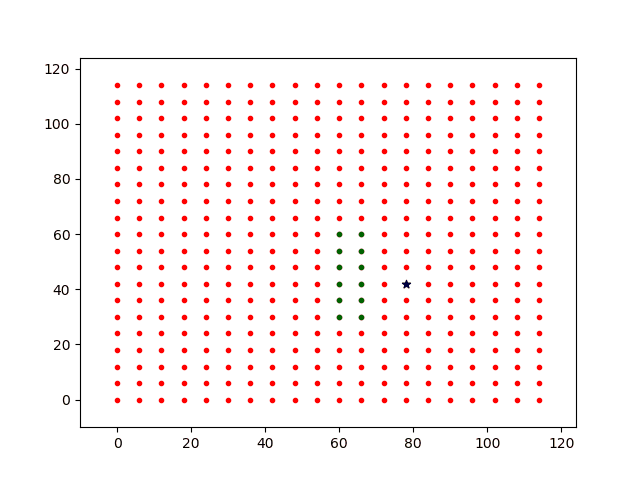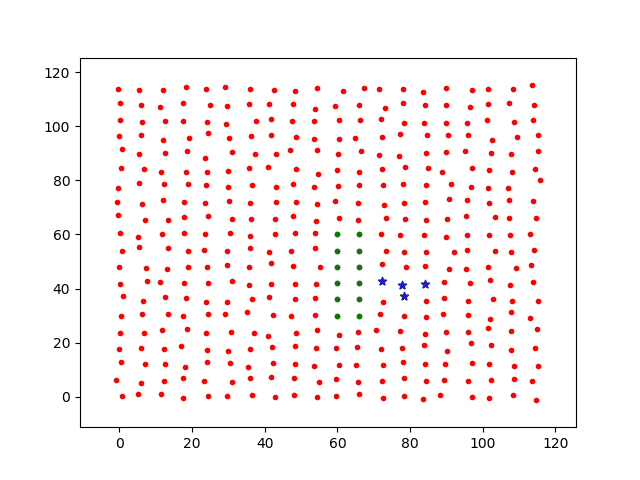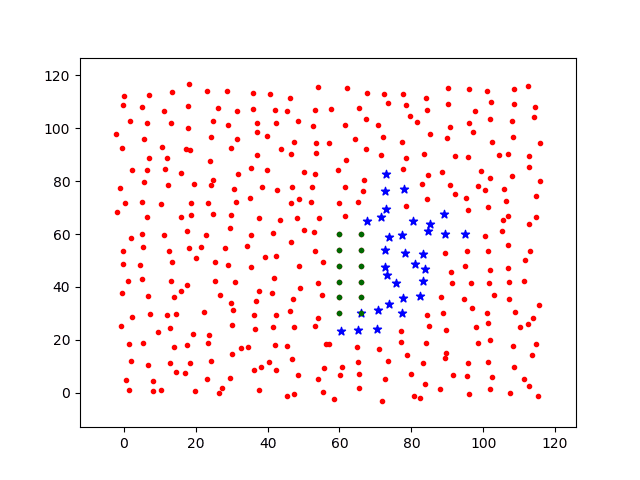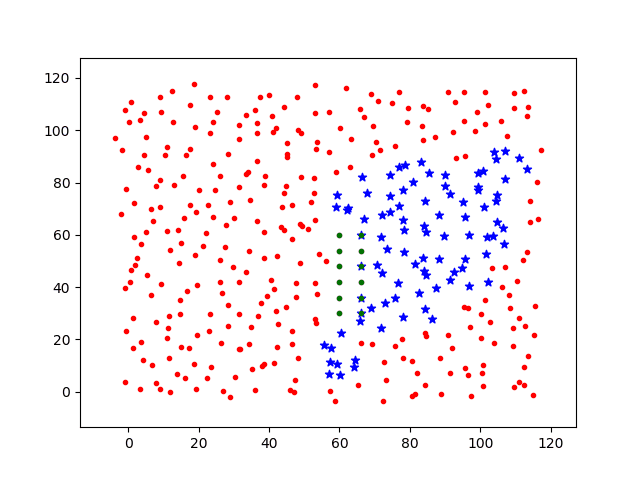
可以看到,自我隔离后的个体被感染的几率明显降低。
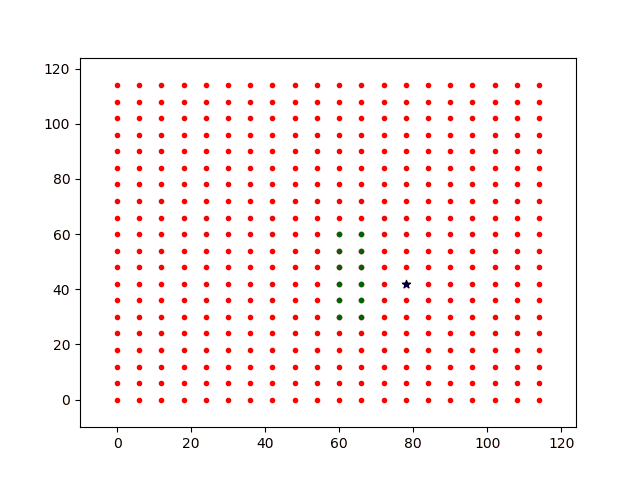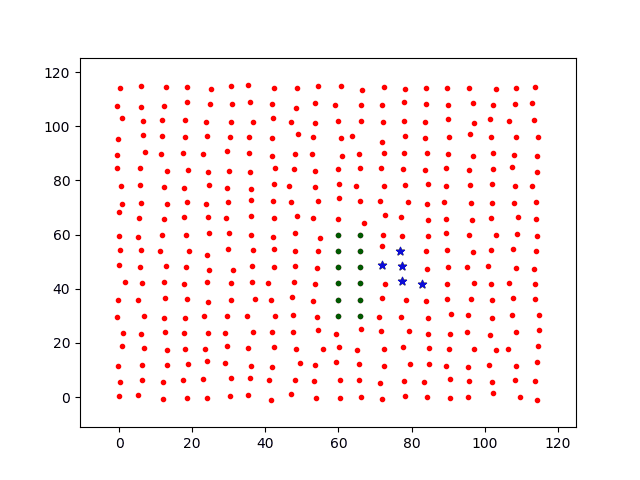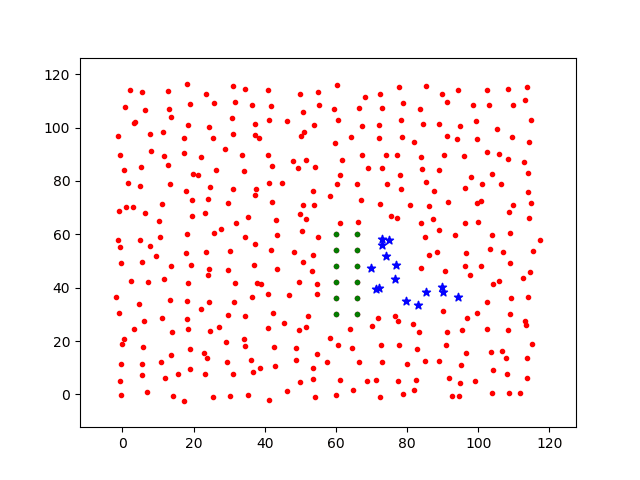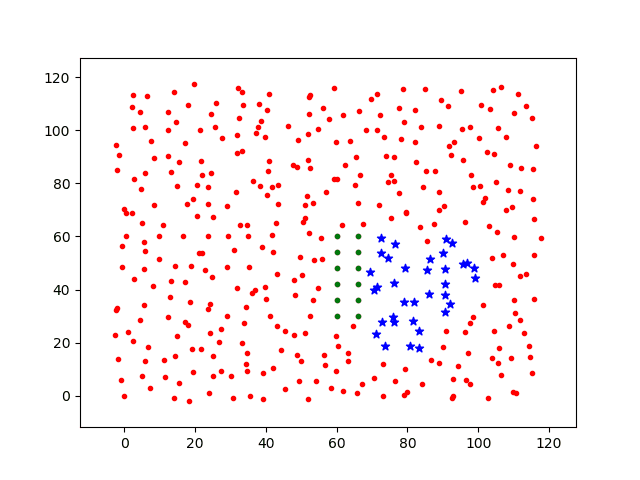
如果将隔离者的基础感染几率设置为p0=0.0,将不会感染,而且将会将低传播速度,因为隔离减少了传播路径。
这也意味着,自我隔离是降低传播的重要途径
2.3 频繁活动模拟
现在提高部分个体的活跃度,将1/3个体的活动步长设置为dstep=0.5,感染过程图如下
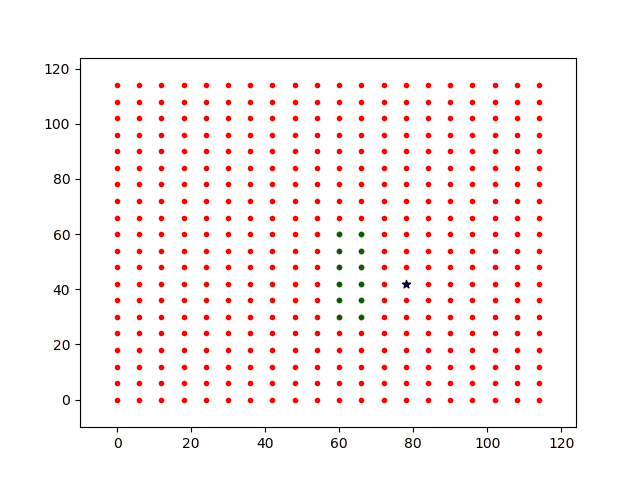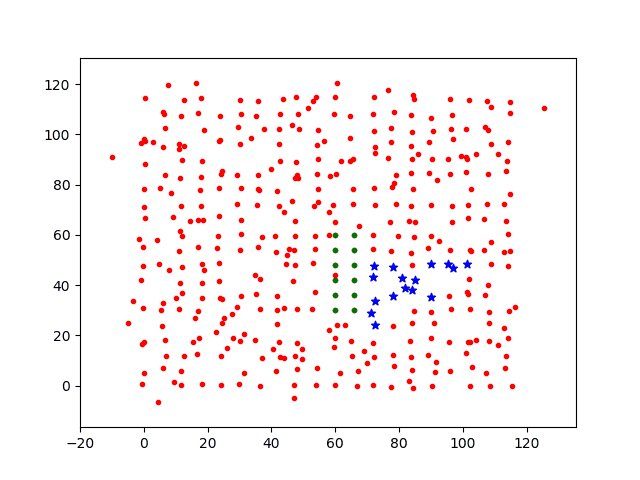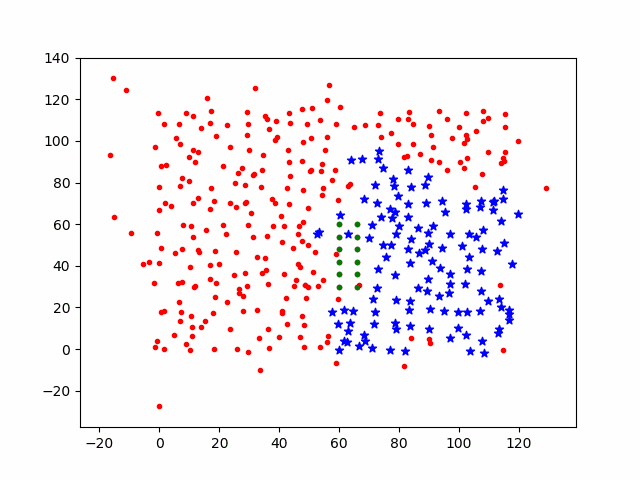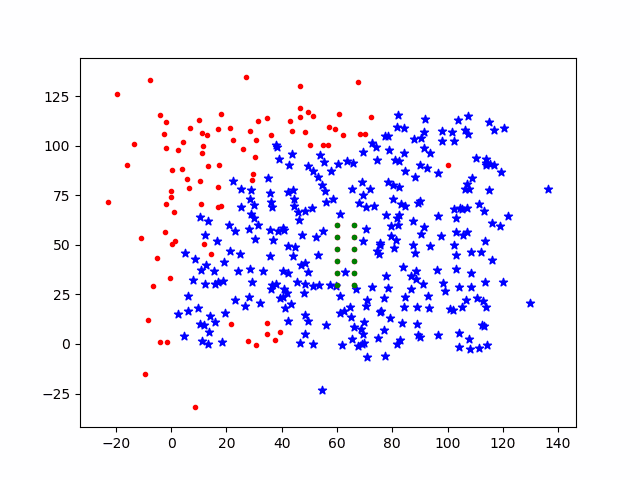
可以看到,随着个体活跃度的增加,感染速度增加。但隔离的个体依然不会受到感染。
3 结论和展望
3.1 结论
结论和现在的实际情况完全一致:
1、自我隔离
2、如若有感染征兆,及时就医
3.2 展望
目前的分析比较简单,调整参数很花时间,但时间有限。有可能希望后期能跟进一些工作:
1、更多感染几率模型的考虑,包括临界距离和模型底数的调整
2、超级感染者的传播路径
3、社区传播模拟(个体间设置亲密度系数)
4 特别说明
1、因为是概率模型,每次计算结果都不尽相同,但差距在可控范围内。时间仓促,老裴没有时间调出最优参数,发现最符合实际的规律。还可以做更多的分析工作,但老裴现在实在没有心思。
2、允许任何人在此模型基础上开展完善和改进,调出最有参数,但必须有引用说明
3、该列将收录至老裴新书《Python科学计算基础》一书
5 计算程序Python代码(参数需要自行调)
#导入所需的库
import numpy as np
import matplotlib.pyplot as plt
#创建扩散模型类
class Diffusion:
#初始化
def __init__(self,N):
self.N = N
self.NI = N**2
self.indivduals = None
self.df = None
self.NoP = 0
#初始化个体位置
def init_pos(self,df):
self.df = df
N = self.N
x,y = np.linspace(0,(N-1)*df,N),\
np.linspace(0,(N-1)*df,N)
vx,vy = np.meshgrid(x,y)
self.ix,self.iy = vx.flatten(),vy.flatten()
self.iix,self.iiy = vx.flatten(),vy.flatten()
#设置模型参数
def init_para(self,D0,P0,W,d_step = 0.05,t_step = 0.01,A=np.e):
self.D0 = np.asarray(D0)
self.P0 = np.asarray(P0)
self.W = np.asarray(W)
self.A = np.asarray(A)
d0 = np.max(self.D0)
p0 = np.min(self.P0)
w = np.min(self.W)
a = np.min(self.A)
self.t_step = t_step
self.d_step = d_step
df = self.df
self.p_min = (1+w)*p0/(a**(df-d0))
self.patients = np.zeros(self.NI,dtype = int)
#放置感染源
def place_source(self,source):
self.patients[source] = 1
#个体游走
def walk(self,t):
t_step = self.t_step
d_step = self.d_step
NI = self.NI
step_d = d_step*self.df
n_step = int(t//t_step)
moves = np.random.random_integers(1,4,n_step*NI).reshape(n_step,NI)
ix,iy = self.ix,self.iy
iix,iiy = self.iix,self.iiy
UP,DOWN,LEFT,RIGHT = 1,2,3,4
for step in range(n_step):
this = moves[step]
ix += np.where(this == RIGHT,d_step,0.)
ix -= np.where(this == LEFT,d_step,0.)
iy += np.where(this == UP,d_step,0.)
iy -= np.where(this == DOWN,d_step,0.)
if np.any(self.patients):
self.update_patients()
self.NoP = np.nonzero(self.patients)[0].shape[0]
#更新感染者
def update_patients(self):
df = self.df
current_patients = np.nonzero(self.patients)[0]
for patient in current_patients:
Dx = self.ix[patient] - self.ix
Dy = self.iy[patient] - self.iy
Dxy = np.hypot(Dx,Dy)
Dxy = np.where(Dxy>df,df,Dxy)
Dij = Dxy-self.D0
WP0 = (1+self.W)*self.P0
WP1 = (1+self.W)*self.P0/(self.A**(Dij))
Pij0 = np.where(Dij<=0,WP0,0.)
Pij1 = np.where(Dij>0,WP1,0.)
Pij = Pij0 + Pij1
Pr = np.random.uniform(self.p_min,1,self.NI)
DP = Pr - Pij
infected = np.argwhere(DP<0)
self.patients[infected] = 1
def plot(self):
ix,iy = self.ix,self.iy
patients = self.patients
x_max,x_min = np.max(ix),np.min(ix)
y_max,y_min = np.max(iy),np.min(iy)
x_max,x_min = x_max+10,x_min-10
y_max,y_min = y_max+10,y_min-10
infected = np.where(patients == 1.)
noninfected = np.where(patients != 1.)
iix = ix[infected]
iiy = iy[infected]
nix = ix[noninfected]
niy = iy[noninfected]
plt.axis([x_min,x_max,y_min,y_max])
plt.scatter(iix,iiy,marker = "*",color = "b")
plt.scatter(nix,niy,marker = ".",color = "r")
plt.show()
if __name__ == "__main__":
N = 20
NI = N*N
D0 = 2.
P0 = 0.3
W = 0
df = 6.
t_step = 0.01
d_step = 0.05
m = Diffusion(N)
m.init_pos(df)
m.init_para(D0,P0,W,t_step = t_step,d_step = d_step)
m.place_source(153)
numbers = []
m.plot()
for t in [1,2,3,4,5,6,7]:
m.walk(t)
m.plot()
numbers.append(m.NoP)
























 1077
1077











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








