糖尿病遗传风险检测挑战赛
赛题介绍
截至2022年,中国糖尿病患者近1.3亿。中国糖尿病患病原因受生活方式、老龄化、城市化、家族遗传等多种因素影响。同时,糖尿病患者趋向年轻化。
糖尿病可导致心血管、肾脏、脑血管并发症的发生。因此,准确诊断出患有糖尿病个体具有非常重要的临床意义。糖尿病早期遗传风险预测将有助于预防糖尿病的发生。
赛事地址:https://challenge.xfyun.cn/topic/info?type=diabetes&ch=ds22-dw-zmt01
赛题任务
在这次比赛中,您需要通过训练数据集构建糖尿病遗传风险预测模型,然后预测出测试数据集中个体是否患有糖尿病,和我们一起帮助糖尿病患者解决这“甜蜜的烦恼”。
赛题数据
赛题数据由训练集和测试集组成,具体情况如下:
- 训练集:共有5070条数据,用于构建您的预测模型
- 测试集:共有1000条数据,用于验证预测模型的性能。
其中训练集数据包含有9个字段:性别、出生年份、体重指数、糖尿病家族史、舒张压、口服耐糖量测试、胰岛素释放实验、肱三头肌皮褶厚度、患有糖尿病标识(数据标签)。
评分标准
采用二分类任务中的F1-score指标进行评价,F1-score越大说明预测模型性能越好,F1-score的定义如下:
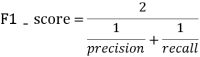
其中:


Tips: 根据题意,糖尿病遗传风险检测挑战赛中会提供2个数据集,分别是训练数据集和测试数据集,其中训练数据集有特征数据和数据标签(患者是否得糖尿病),测试数据集只有特征数据,我们需要根据糖尿病遗传风险预测模型,比赛方通过测试数据集来评估模型的预测准确性,模型预测的准确性越高越好。
Ref:
入门篇
【参考:如何打一个数据挖掘比赛- 入门篇——DATAWHALE - 一个热爱学习的社区】
赛题Baseline
Tips: 在本次比赛中,我们将提供python代码用于比赛数据的分析与模型构建,如果你还不熟悉赛题中的相关代码与原理,可以参考相关学习资料或在Datawhale交流群中来解决你遇到的问题。
安装相关第三方库
#安装相关依赖库
!pip install lightgbm
!pip install pandas
!pip install sklearn
导入第三方库
Tips: 在本baseline中,我们通过pandas对数据进行处理,通过lightgbm算法来构建糖尿病遗传风险预测模型
Ref:
import pandas as pd
import lightgbm
数据预处理
Tips: 在本环节中,我们通常需要检测数据的质量,包括重复值、异常值、缺失值、数据分布和数据特征等,通过训练数据的预处理,我们能得到更高质量的训练数据,这有助于构建更加准确的预测模型。
在本baseline中,我们发现
舒张压特征中存在缺失值,我们采用了填充缺失值的方法进行处理,当然也有其他的处理方法,如果感兴趣可以尝试。Ref:
data1=pd.read_csv('比赛训练集.csv',encoding='gbk')
data2=pd.read_csv('比赛测试集.csv',encoding='gbk')
#测试集的label标记为-1,以便后续挑出测试集
data2['患有糖尿病标识']=-1
#训练集和测试集合并
data=pd.concat([data1,data2],axis=0,ignore_index=True)
#将舒张压特征中的缺失值填充为-1
data['舒张压']=data['舒张压'].fillna(-1)
特征工程
Tips:在本环节中,我们需要对数据进行特征的构造,目的是最大限度地从原始数据中提取特征以供算法和模型使用,这有助于构建更加准确的预测模型。
Ref:
#特征工程
"""
将出生年份换算成年龄
"""
data['出生年份']=2022-data['出生年份'] #换成年龄
"""
人体的成人体重指数正常值是在18.5-24之间
低于18.5是体重指数过轻
在24-27之间是体重超重
27以上考虑是肥胖
高于32了就是非常的肥胖。
"""
def BMI(a):
if a<18.5:
return 0
elif 18.5<=a<=24:
return 1
elif 24<a<=27:
return 2
elif 27<a<=32:
return 3
else:
return 4
data['BMI']=data['体重指数'].apply(BMI)
#糖尿病家族史
"""
无记录
叔叔或者姑姑有一方患有糖尿病/叔叔或姑姑有一方患有糖尿病
父母有一方患有糖尿病
"""
def FHOD(a):
if a=='无记录':
return 0
elif a=='叔叔或者姑姑有一方患有糖尿病' or a=='叔叔或姑姑有一方患有糖尿病':
return 1
else:
return 2
data['糖尿病家族史']=data['糖尿病家族史'].apply(FHOD)
"""
舒张压范围为60-90
"""
def DBP(a):
if a<60:
return 0
elif 60<=a<=90:
return 1
elif a>90:
return 2
else:
return a
data['DBP']=data['舒张压'].apply(DBP)
#------------------------------------
#将处理好的特征工程分为训练集和测试集,其中训练集是用来训练模型,测试集用来评估模型准确度
#其中编号和患者是否得糖尿病没有任何联系,属于无关特征予以删除
train=data[data['患有糖尿病标识'] !=-1]
test=data[data['患有糖尿病标识'] ==-1] # 取出测试集
train_label=train['患有糖尿病标识']
train=train.drop(['编号','患有糖尿病标识'],axis=1)
test=test.drop(['编号','患有糖尿病标识'],axis=1)
构建模型
Tips:在本环节中,我们需要对训练集进行训练从而构建相应的模型,在本baseline中我们使用了Lightgbm算法进行数据训练,当然你也可以使用其他的机器学习算法/深度学习算法,甚至你可以将不同算法预测的结果进行综合,反正最后的目的是获得更高的预测准确度,向着这个目标出发~
在本节中,我们将训练数据使用5折交叉验证训练的方法进行训练,这是一个不错的提升模型预测准确度的方法
Ref:
#使用Lightgbm方法训练数据集,使用5折交叉验证的方法获得5个测试集预测结果
from sklearn.model_selection import KFold
def select_by_lgb(train_data,train_label,test_data,random_state=2022,n_splits=5,metric='auc',num_round=10000,early_stopping_rounds=100):
kfold = KFold(n_splits=n_splits, shuffle=True, random_state=random_state)
fold=0
result=[]
for train_idx, val_idx in kfold.split(train_data):
random_state+=1
train_x = train_data.loc[train_idx]
train_y = train_label.loc[train_idx]
test_x = train_data.loc[val_idx]
test_y = train_label.loc[val_idx]
clf=lightgbm
train_matrix=clf.Dataset(train_x,label=train_y)
test_matrix=clf.Dataset(test_x,label=test_y)
params={
'boosting_type': 'gbdt',
'objective': 'binary',
'learning_rate': 0.1,
'metric': metric,
'seed': 2020,
'nthread':-1 }
model=clf.train(params,train_matrix,num_round,valid_sets=test_matrix,early_stopping_rounds=early_stopping_rounds)
pre_y=model.predict(test_data)
result.append(pre_y)
fold+=1
return result
test_data=select_by_lgb(train,train_label,test)
#test_data就是5折交叉验证中5次预测的结果
pre_y=pd.DataFrame(test_data).T
#将5次预测的结果求取平均值,当然也可以使用其他的方法
pre_y['averge']=pre_y[[i for i in range(5)]].mean(axis=1)
#因为竞赛需要你提交最后的预测判断,而模型给出的预测结果是概率,因此我们认为概率>0.5的即该患者有糖尿病,概率<=0.5的没有糖尿病
pre_y['label']=pre_y['averge'].apply(lambda x:1 if x>0.5 else 0)
pre_y
结果提交
Tips:在本环节中,我们需要将最后的预测结果提交到数据竞赛平台中,需要注意的是我们要严格按照竞赛平台的文件格式提交要求。
- 提交地址: https://challenge.xfyun.cn/topic/info?type=diabetes&ch=ds22-dw-zmt01
其中result.csv就是需要提交到平台的文件,进入到数据竞赛平台,点击
提交结果,选择result.csv文件即可完成结果提交
result=pd.read_csv('提交示例.csv')
result['label']=pre_y['label']
result.to_csv('result.csv',index=False)
后续
经过简单的学习,我们完成了糖尿病遗传风险检测挑战赛的baseline任务,接下来应该怎么做呢?主要是以下几个方面:
- 继续尝试不同的预测模型或特征工程来提升模型预测的准确度
- 加入Datawhale比赛交流群,获取其他更加有效的上分信息
- 查阅糖尿病遗传风险预测相关资料,获取其他模型构建方法
- …
总之,就是在baseline的基础上不断的改造与尝试,通过不断的实践来提升自己的数据挖掘能力,正所谓【纸上得来终觉浅,绝知此事要躬行】,也许你熟练掌握机器学习的相关算法,能熟练推导各种公式,但如何将学习到的方法应用到实践工程中,需要我们不断的尝试与改进,没有一个模型是一步所得,向最后的冠军冲击~
Ref:
进阶篇(强烈建议阅读)
【参考:如何打一个数据挖掘比赛- 入门篇 —— DATAWHALE 】
强烈建议阅读,写得非常不错,可以学习一下思路和代码
- 逻辑回归(分数:0.74):
- 决策树(分数:0.93):
- lightgbm版本5折交叉验证(分数:0.96):
- 模型融合























 585
585











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








