-----------------------------C++----------------------------------------
// Clustering by fast search and find of density peaks
// Science 27 June 2014:
// Vol. 344 no. 6191 pp. 1492-1496
// DOI: 10.1126/science.1242072
// http://www.sciencemag.org/content/344/6191/1492.full//
//
// Code Author: Eric Yuan
// Blog: http://eric-yuan.me
// You are FREE to use the following code for ANY purpose.
//
// Have fun with it
#include "iostream"
#include <stdio.h>
#include <ctime>
#include "vector"
#include "math.h"
using namespace std;
#define DIM 3
#define elif else if
#ifndef bool
#define bool int
#define false ((bool)0)
#define true ((bool)1)
#endif
struct Point3d {
double x;
double y;
double z;
Point3d(double xin, double yin, double zin) : x(xin), y(yin), z(zin) {}
};
void dataPro(vector<vector<double> > &src, vector<Point3d> &dst){
for(int i = 0; i < src.size(); i++){
Point3d pt(src[i][0], src[i][1], src[i][2]);
dst.push_back(pt);
}
}
double getDistance(Point3d &pt1, Point3d &pt2){
double tmp = pow(pt1.x - pt2.x, 2) + pow(pt1.y - pt2.y, 2) + pow(pt1.z - pt2.z, 2);
return pow(tmp, 0.5);
}
double getdc(vector<Point3d> &data, double neighborRateLow, double neighborRateHigh){
int nSamples = data.size();
int nLow = neighborRateLow * nSamples * nSamples;
int nHigh = neighborRateHigh * nSamples * nSamples;
double dc = 0.0;
int neighbors = 0;
cout<<"nLow = "<<nLow<<", nHigh = "<<nHigh<<endl;
while(neighbors < nLow || neighbors > nHigh){
//while(dc <= 1.0){
neighbors = 0;
for(int i = 0; i < nSamples - 1; i++){
for(int j = i + 1; j < nSamples; j++){
if(getDistance(data[i], data[j]) <= dc) ++neighbors;
if(neighbors > nHigh) goto DCPLUS;
}
}
DCPLUS: dc += 0.03;
cout<<"dc = "<<dc<<", neighbors = "<<neighbors<<endl;
}
return dc;
}
vector<int> getLocalDensity(vector<Point3d> &points, double dc){
int nSamples = points.size();
vector<int> rho(nSamples, 0);
for(int i = 0; i < nSamples - 1; i++){
for(int j = i + 1; j < nSamples; j++){
if(getDistance(points[i], points[j]) < dc){
++rho[i];
++rho[j];
}
}
//cout<<"getting rho. Processing point No."<<i<<endl;
}
return rho;
}
vector<double> getDistanceToHigherDensity(vector<Point3d> &points, vector<int> &rho){
int nSamples = points.size();
vector<double> delta(nSamples, 0.0);
for(int i = 0; i < nSamples; i++){
double dist = 0.0;
bool flag = false;
for(int j = 0; j < nSamples; j++){
if(i == j) continue;
if(rho[j] > rho[i]){
double tmp = getDistance(points[i], points[j]);
if(!flag){
dist = tmp;
flag = true;
}else dist = tmp < dist ? tmp : dist;
}
}
if(!flag){
for(int j = 0; j < nSamples; j++){
double tmp = getDistance(points[i], points[j]);
dist = tmp > dist ? tmp : dist;
}
}
delta[i] = dist;
//cout<<"getting delta. Processing point No."<<i<<endl;
}
return delta;
}
int main(int argc, char** argv)
{
long start, end;
FILE *input;
input = fopen("dataset.txt", "r");
vector<vector<double> > data;
double tpdouble;
int counter = 0;
while(1){
if(fscanf(input, "%lf", &tpdouble)==EOF) break;
if(counter / 3 >= data.size()){
vector<double> tpvec;
data.push_back(tpvec);
}
data[counter / 3].push_back(tpdouble);
++ counter;
}
fclose(input);
//random_shuffle(data.begin(), data.end());
start = clock();
cout<<"********"<<endl;
vector<Point3d> points;
dataPro(data, points);
double dc = getdc(points, 0.016, 0.020);
vector<int> rho = getLocalDensity(points, dc);
vector<double> delta = getDistanceToHigherDensity(points, rho);
// saveToTxt(rho, delta);
// now u get the cluster centers
end = clock();
cout<<"used time: "<<((double)(end - start)) / CLOCKS_PER_SEC<<endl;
return 0;
}
/*
void saveToTxt(vector &rho){
FILE *p;
p = fopen("rho.txt", "w");
for(int i = 0; i < rho.size(); i++){
fprintf(p, "%f\n", rho[i]);
}
fclose(p);
}
*/
---------------------------------matlab------------------------------------------------
clear all
close all
disp('The only input needed is a distance matrix file')
disp('The format of this file should be: ')
disp('Column 1: id of element i')
disp('Column 2: id of element j')
disp('Column 3: dist(i,j)')
%mdist=input('example_distances.dat');
disp('Reading input distance matrix')
xx=load('example_distances.dat');
ND=max(xx(:,2));
NL=max(xx(:,1));
if (NL>ND)
ND=NL;
end
N=size(xx,1);
for i=1:ND
for j=1:ND
dist(i,j)=0;
end
end
for i=1:N
ii=xx(i,1);
jj=xx(i,2);
dist(ii,jj)=xx(i,3);
dist(jj,ii)=xx(i,3);
end
percent=2.0;
fprintf('average percentage of neighbours (hard coded): %5.6f\n', percent);
position=round(N*percent/100);
sda=sort(xx(:,3));
dc=sda(position);
fprintf('Computing Rho with gaussian kernel of radius: %12.6f\n', dc);
for i=1:ND
rho(i)=0.;
end
%
% Gaussian kernel
%
for i=1:ND-1
for j=i+1:ND
rho(i)=rho(i)+exp(-(dist(i,j)/dc)*(dist(i,j)/dc));
rho(j)=rho(j)+exp(-(dist(i,j)/dc)*(dist(i,j)/dc));
end
end
%
% "Cut off" kernel
%
%for i=1:ND-1
% for j=i+1:ND
% if (dist(i,j)<dc)
% rho(i)=rho(i)+1.;
% rho(j)=rho(j)+1.;
% end
% end
%end
maxd=max(max(dist));
[rho_sorted,ordrho]=sort(rho,'descend');
delta(ordrho(1))=-1.;
nneigh(ordrho(1))=0;
for ii=2:ND
delta(ordrho(ii))=maxd;
for jj=1:ii-1
if(dist(ordrho(ii),ordrho(jj))<delta(ordrho(ii)))
delta(ordrho(ii))=dist(ordrho(ii),ordrho(jj));
nneigh(ordrho(ii))=ordrho(jj);
end
end
end
delta(ordrho(1))=max(delta(:));
disp('Generated file:DECISION GRAPH')
disp('column 1:Density')
disp('column 2:Delta')
fid = fopen('DECISION_GRAPH', 'w');
for i=1:ND
fprintf(fid, '%6.2f %6.2f\n', rho(i),delta(i));
end
disp('Select a rectangle enclosing cluster centers')
scrsz = get(0,'ScreenSize');
figure('Position',[6 72 scrsz(3)/4. scrsz(4)/1.3]);
for i=1:ND
ind(i)=i;
gamma(i)=rho(i)*delta(i);
end
subplot(2,1,1)
tt=plot(rho(:),delta(:),'o','MarkerSize',5,'MarkerFaceColor','k','MarkerEdgeColor','k');
title ('Decision Graph','FontSize',15.0)
xlabel ('\rho')
ylabel ('\delta')
subplot(2,1,1)
rect = getrect(1);
rhomin=rect(1);
deltamin=rect(4);
NCLUST=0;
for i=1:ND
cl(i)=-1;
end
for i=1:ND
if ( (rho(i)>rhomin) && (delta(i)>deltamin))
NCLUST=NCLUST+1;
cl(i)=NCLUST;
icl(NCLUST)=i;
end
end
fprintf('NUMBER OF CLUSTERS: %i \n', NCLUST);
disp('Performing assignation')
%assignation
for i=1:ND
if (cl(ordrho(i))==-1)
cl(ordrho(i))=cl(nneigh(ordrho(i)));
end
end
%halo
for i=1:ND
halo(i)=cl(i);
end
if (NCLUST>1)
for i=1:NCLUST
bord_rho(i)=0.;
end
for i=1:ND-1
for j=i+1:ND
if ((cl(i)~=cl(j))&& (dist(i,j)<=dc))
rho_aver=(rho(i)+rho(j))/2.;
if (rho_aver>bord_rho(cl(i)))
bord_rho(cl(i))=rho_aver;
end
if (rho_aver>bord_rho(cl(j)))
bord_rho(cl(j))=rho_aver;
end
end
end
end
for i=1:ND
if (rho(i)<bord_rho(cl(i)))
halo(i)=0;
end
end
end
for i=1:NCLUST
nc=0;
nh=0;
for j=1:ND
if (cl(j)==i)
nc=nc+1;
end
if (halo(j)==i)
nh=nh+1;
end
end
fprintf('CLUSTER: %i CENTER: %i ELEMENTS: %i CORE: %i HALO: %i \n', i,icl(i),nc,nh,nc-nh);
end
cmap=colormap;
for i=1:NCLUST
ic=int8((i*64.)/(NCLUST*1.));
subplot(2,1,1)
hold on
plot(rho(icl(i)),delta(icl(i)),'o','MarkerSize',8,'MarkerFaceColor',cmap(ic,:),'MarkerEdgeColor',cmap(ic,:));
end
subplot(2,1,2)
disp('Performing 2D nonclassical multidimensional scaling')
Y1 = mdscale(dist, 2, 'criterion','metricstress');
plot(Y1(:,1),Y1(:,2),'o','MarkerSize',2,'MarkerFaceColor','k','MarkerEdgeColor','k');
title ('2D Nonclassical multidimensional scaling','FontSize',15.0)
xlabel ('X')
ylabel ('Y')
for i=1:ND
A(i,1)=0.;
A(i,2)=0.;
end
for i=1:NCLUST
nn=0;
ic=int8((i*64.)/(NCLUST*1.));
for j=1:ND
if (halo(j)==i)
nn=nn+1;
A(nn,1)=Y1(j,1);
A(nn,2)=Y1(j,2);
end
end
hold on
plot(A(1:nn,1),A(1:nn,2),'o','MarkerSize',2,'MarkerFaceColor',cmap(ic,:),'MarkerEdgeColor',cmap(ic,:));
end
%for i=1:ND
% if (halo(i)>0)
% ic=int8((halo(i)*64.)/(NCLUST*1.));
% hold on
% plot(Y1(i,1),Y1(i,2),'o','MarkerSize',2,'MarkerFaceColor',cmap(ic,:),'MarkerEdgeColor',cmap(ic,:));
% end
%end
faa = fopen('CLUSTER_ASSIGNATION', 'w');
disp('Generated file:CLUSTER_ASSIGNATION')
disp('column 1:element id')
disp('column 2:cluster assignation without halo control')
disp('column 3:cluster assignation with halo control')
for i=1:ND
fprintf(faa, '%i %i %i\n',i,cl(i),halo(i));
end
===========参考=====
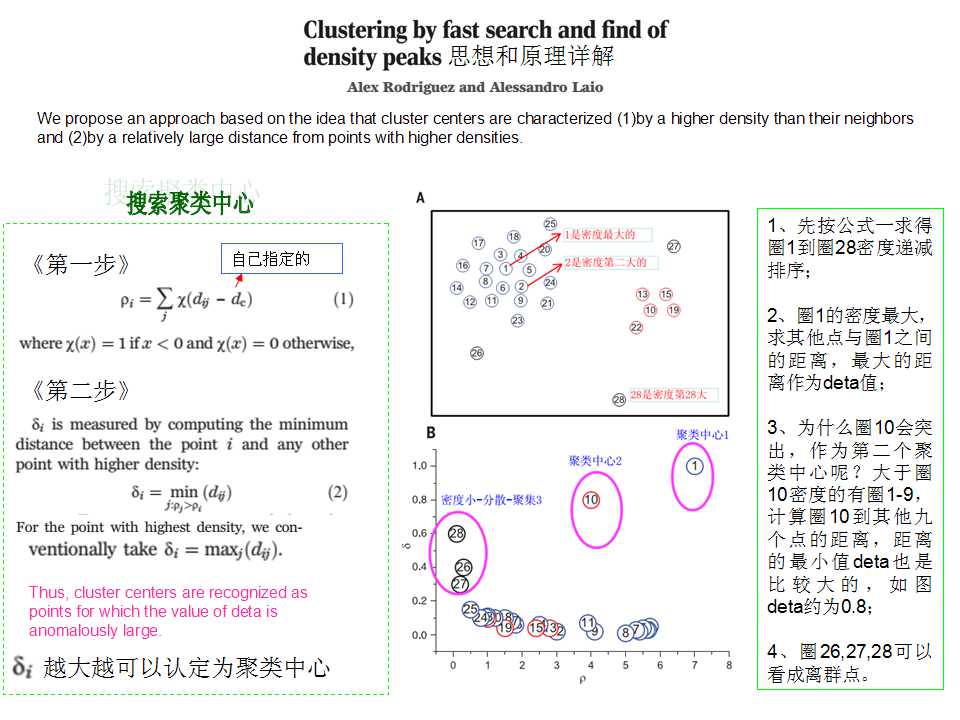
Clustering by fast search and find of density peaks Alex Rodriguez and Alessandro Laio Science 344, 1492 (2014)
http://eric-yuan.me/clustering-fast-search-find-density-peahttp://www.sciencemag.org/content/344/6191/1492.full//






















 890
890

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








