第一章 常用函数及参数介绍
1.1 常用函数
1.1.1 创建族群函数——crtbp
1.调用格式
[chrom,land,basev]=crtbp(Nind,Lind)
[chrom,land,basev]=crtbp(Nind,Base)
[chrom,land,basev]=crtbp(Nind,Lind,Base)
注:Nind:个体数;Lind:个体长度; Base:一个个体每个染色体基因位的进制数。
2.代码举例
%% 遗传算法的创建族群函数,方法1
[Chrom,land,basev]=crtbp(5,10)
%crtbp函数默认为二进制随机种群,这里我设置的是一个族群大小为5,个体长度为10的二进制随机族群。
%% 遗传算法的创建族群函数,方法2
[Chrom,land,basev]=crtbp(8,[2 2 2 3 4 5 6 7 8 9])
%这里我设置的族群中的个体为8个,每个个体的长度通过basev来确定,每个个体每位编码的进制度为2 2 2 3 4 5 6 7 8 9
%% 遗传算法的创建族群函数,方法3
[Chrom,land,basev]=crtbp(5,8,[2 3 4 5 6 7 8 9])
%这里我设置的族群中的个体共有5个,每个个体的长度为8,且个体每位编码的进制度为2到9
结果1:
Chrom =
1 1 1 0 0 0 1 1 1 0
0 1 0 1 1 1 0 1 1 1
0 1 1 1 1 0 0 1 1 0
1 0 0 0 0 0 1 0 1 0
0 1 0 1 1 1 0 0 1 0
land =
10
basev =
2 2 2 2 2 2 2 2 2 2
结果2:
Chrom =
1 1 1 2 2 1 4 5 3 5
0 1 1 0 0 2 1 4 3 1
0 1 0 1 2 3 1 6 6 1
1 1 0 2 2 0 5 0 5 4
0 0 0 1 2 0 5 4 1 8
0 1 1 2 3 1 3 1 2 4
1 0 1 0 3 2 1 5 1 0
0 1 1 0 3 4 0 1 5 0
land =
10
basev =
2 2 2 3 4 5 6 7 8 9
结果3:
Chrom =
0 1 0 3 0 1 2 3
1 0 1 3 5 1 1 0
0 2 2 1 1 2 3 5
1 2 2 2 3 6 2 0
1 2 2 0 5 2 1 5
land =
8
basev =
2 3 4 5 6 7 8 9
1.1.2 适应度计算函数——ranking
1.调用格式
FitnV=ranking(ObjV)
FitnV=ranking(ObjV,RFun)
FitnV=ranking(ObjV,RFun,SUBPOP)
注:
Objv:是个体的目标值(列向量);
RFun:是指定排序方法及选择强度,RFun有三个情况:
(1)RFun是一个具有两个参数的向量
RFun(1):对线性排序,指定压差值为[1,2]的某个值;对非线性排序,RFun(1)在[1,length(ObjV)-2]区间;默认值为2。Rfun(2):指定排序方法,0为线性排列,1为非线性排列。
(2)RFun是一个在[1,2]区间的标量,则采用线性排序
(3)RFun是长度为length(ObjV)的向量,则包含对每一行适应度值的计算
SUBPOP是一个任选参数,指明在ObjV中子群种的数量。默认为1,在ObjV中所有子种群大小必须相同。ranking独立地对每个子种群执行。
2.代码举例
%格式一:
objv=[1;2;3;4;5;10;9;8;7;6]
fitnv=ranking(objv)%将个体的目标值objv(列向量)按从小到达的顺序排列,并返回个体适应度fitnv的列向量
%例如这里[1;2;3;4;5;10;9;8;7;6]为一个列向量,得到的个体适应度的列向量为:[2.0;1.77;1.55;1.33;1.1;0;0.22;0.44;0.66;0.88]
%注意这里返回的列向量的位置还是原先元素的位置。
%这里默认压差为2,所谓压差即是一个[2,0]的区间,将原先的[1,10]映射到[2,0]上,即1为0,10为0其余数字按大小顺序线性插值。
%格式二:
%当线性排列的时候,标量指定的选择压差rfun必须要在[1,2]区间,对于非线性排列[1,length(objv)-2]区间
fitnv=ranking(objv,[1.5,0])
%格式三:
fitnv=ranking(objv,[2,0],1)
%这里subpop是一个任选参数,知名objv中子种群的数量,如果subpop=1则要求,objv中的每一个子种群大小必须相同,如果ranking被调用于多子种群,则ranking独立地对每个子种群执行结果:
objv =
1
2
3
4
5
10
9
8
7
6
fitnv =
2.0000
1.7778
1.5556
1.3333
1.1111
0
0.2222
0.4444
0.6667
0.8889
fitnv =
1.5000
1.3889
1.2778
1.1667
1.0556
0.5000
0.6111
0.7222
0.8333
0.9444
fitnv =
2.0000
1.7778
1.5556
1.3333
1.1111
0
0.2222
0.4444
0.6667
0.8889
1.1.3 选择函数——select
1.调用格式
Selch=select(SEL_F,Chrom,FitnV)
Selch=select(SEL_F,Chrom,FitnV,GGAP)
Selch=select(SEL_F,Chrom,FitnV,GGAP,SUBPOP)
注:
SEL_F是一个字符串,包含两个低级函数即rws(轮盘赌)函数和sus(随机通用采样)函数
rws:轮盘赌函数

sus:随机通用采样函数

Chrom:是种群;
FitnV:是适应度值;
GGAP:是代沟,对于代沟的解释如下
(1) 代沟*父代个体数=子代个体数
(2)代沟表示了父代群体中个体被置换的比率
(3)父代群体中个体被置换的比率要么是设置代沟引起的要么是设置代沟后再进行基于适应度重插引起的。
SUBPOP:是一个可选参数,确定了目标群体中子种群的数量。如果suspop省略或为NAN,则目标种群中所有子种群必须有相同的大小。
2.代码举例
sus函数:
chrom=[1 11 21;2 12 22;3 13 23; 4 14 24; 5 15 25;6 16 26;7 17 27;8 18 28]
fitnv=[1.5;1.35;1.21;1.07;0.92;0.78;0.64;0.5]
Selch=select('sus',chrom,fitnv)结果:
Selch =
7 17 27
1 11 21
3 13 23
5 15 25
6 16 26
2 12 22
4 14 24
1 11 21
rws函数:
%% 选择函数——select
chrom=[1 11 21;2 12 22;3 13 23; 4 14 24; 5 15 25;6 16 26;7 17 27;8 18 28]
fitnv=[1.5;1.35;1.21;1.07;0.92;0.78;0.64;0.5]
Selch=select('rws',chrom,fitnv)结果:
Selch =
3 13 23
6 16 26
7 17 27
2 12 22
2 12 22
4 14 24
4 14 24
3 13 23
1.1.4 交叉算子函数——recombin
1.调用格式
NewChrom=recombin(REC_F,Chrom)
NewChrom=recombin(REC_F,Chrom,RecOpt)
NewChrom=recombin(REC_F,Chrom,RecOpt,SUSBPOP)
注:
REC_F包含低级重组函数名的字符串,包括recidis(离散重组)和xovsp(单点交叉);
RecOpt是一个指明交叉概率的任选函数,默认为缺省值;
2.算法举例
1.2 遗传算法举例
1.2.1 算法代码
clc
clear all
close all
%% 绘制函数图形
hold on
figure(1)
lb=1;ub=2;
ezplot('sin(10*pi*x)/x',[lb,ub]);
xlabel('自变量x')
ylabel('函数值y')
%% 定义遗传算法参数
NIND=40; %种群中个体数为40
MAXGEN=20; %最大遗传代数
PRECI=20; %个体基因位长度
GGAP=0.95; %代沟
px=0.7; %交叉概率
pm=0.01; %变异概率
trace=zeros(2,MAXGEN); %寻优结果的初始值
FieldD=[PRECI;lb;ub;1;0;1;1]; %区域描述器,描述二进制转换为十进制的参数定义
chrom=crtbp(NIND,PRECI); %创建初始随机种群大小为40*20
%% 优化
gen=0; %代数
x=bs2rv(chrom,FieldD); %将二进制转换为十进制,且fieldD前面已经定义好参数,这边直接应用
objv=sin(10*pi*x)./x; %设置目标函数值
while gen<MAXGEN
Fitnv=ranking(objv); %默认压差为2,将objv映射到[2,0]这个区间上
Selch=select('sus',chrom,Fitnv,GGAP); %选择采用sus函数(随机均匀)
Selch=recombin('xovsp',Selch,px); %选择是单点交叉函数,px为交叉概率为0.7
Selch=mut(Selch,pm); %变异概率为0.01
x=bs2rv(Selch,FieldD); %子代课题的进制转换
objvsel=sin(10*pi*x)./x; %子代的目标函数
[chrom,objv]=reins(chrom,Selch,1,1,objv,objvsel); %重新插入子代到父代中
x=bs2rv(chrom,FieldD);
gen=gen+1;
%获取每代的最优解以及其编号,Y为最优解,I为个体的序号
[Y,I]=min(objv)
trace(1,gen)=x(I); %记下每代的最优值
trace(2,gen)=Y; %记下每代的最优解
end
plot(trace(1,:),trace(2,:),'bo'); %画出每代的最优点
grid on;
plot(x,objv,'b*');
hold off %画出最后一代的种群
%% 画出进化图
figure(2);
plot(1:MAXGEN,trace(2,:));
grid on
xlabel('遗传代数');
ylabel('解的过程');
title('进化过程')
bestY=trace(2,end);
bestx=trace(1,end);
fprintf(['最优解:\nx=',num2str(bestx),'\nY=',num2str(bestY),'\n'])结果:

最优解:
x=1.1492
Y=-0.8699

1.3 多元函数的最优解问题
1.3.1 代码部分
clc
clear all
close all
%% 目标函数图(所要求的极值的那个函数)
figure(1)
lbx=-2;ubx=2;%定义出来各个自变量的范围
lby=-2;uby=2;
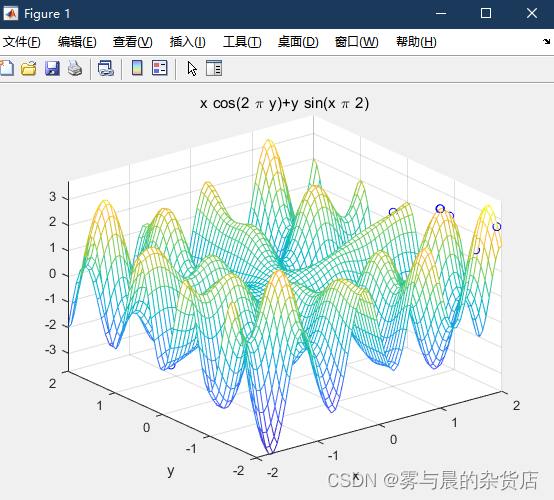
ezmesh('x*cos(2*pi*y)+y*sin(x*pi*2)',[lbx,ubx,lby,uby]);
hold on;
%% 定义遗传算法参数
NIND=40;
MAXGEN=20;
PRICE=20;
GGAP=0.95;
px=0.7;
pm=0.01;
trace=zeros(3,MAXGEN);
FieldD=[PRICE PRICE;lbx lby;ubx uby;1 1 ;0 0;1 1;1 1];
chrom=crtbp(NIND,PRICE*2);
%% 算法优化
gen=0;
XY=bs2rv(chrom,FieldD);
X=XY(:,1);Y=XY(:,2);
ObjV=X.*cos(2*pi*Y)+Y.*sin(X*pi*2);
while gen<MAXGEN
FitnV=ranking(-ObjV);
selch=select('sus',chrom,FitnV,GGAP);
selch=recombin('xovsp',selch,px);
selch=mut(selch,pm);
XY=bs2rv(selch,FieldD);
X=XY(:,1);Y=XY(:,2);
objvsel=Y.*sin(2*pi*X)+X.*cos(2*pi*Y);
[chrom,ObjV]=reins(chrom,selch,1,1,ObjV,objvsel);
XY=bs2rv(chrom,FieldD);
gen=gen+1;
[Y,I]=max(ObjV);
trace(1:2,gen)=XY(I,:);
trace(3,gen)=Y;
end
plot3(trace(1,:),trace(2,:),trace(3,:),'bo');
grid on;
plot3(XY(:,1),XY(:,2),ObjV,'bo');
hold off
%% 画出进化图
figure(2);
plot(1:MAXGEN,trace(3,:));
grid on
xlabel('遗传代数');
ylabel('解的过程');
title('进化过程');
bestZ=trace(3,end);
bestX=trace(1,end);
bestY=trace(2,end);
fprintf(['最优解:\nX=',num2str(bestX),'\nY=',num2str(bestY),'\nZ=',num2str(bestZ),'\n'])1.3.2 运行结果

第三章 基于遗传算法和非线性规划的函数寻优算法
3.1 算法背景描述
标准的遗传算法存在局部搜索能力较为差,容易结果“早熟”的缺点,导致算法过早收敛,而经典的非线性规划算法大多采用梯度下降算法,局部搜索能力强而全局搜索能力较为弱,容易陷入局部最优解。对此我的理解是标准遗传算法由于存在选择、变异以及交换等函数,自变量x的变化范围十分大,因此全局搜索能力较为强。而经典的非线性规划算法主要采用梯度下降算法,对此我的理解是梯度算法类似于求导,求导存在极值和最值的问题,求导容易求得极值,而最值不易求得。
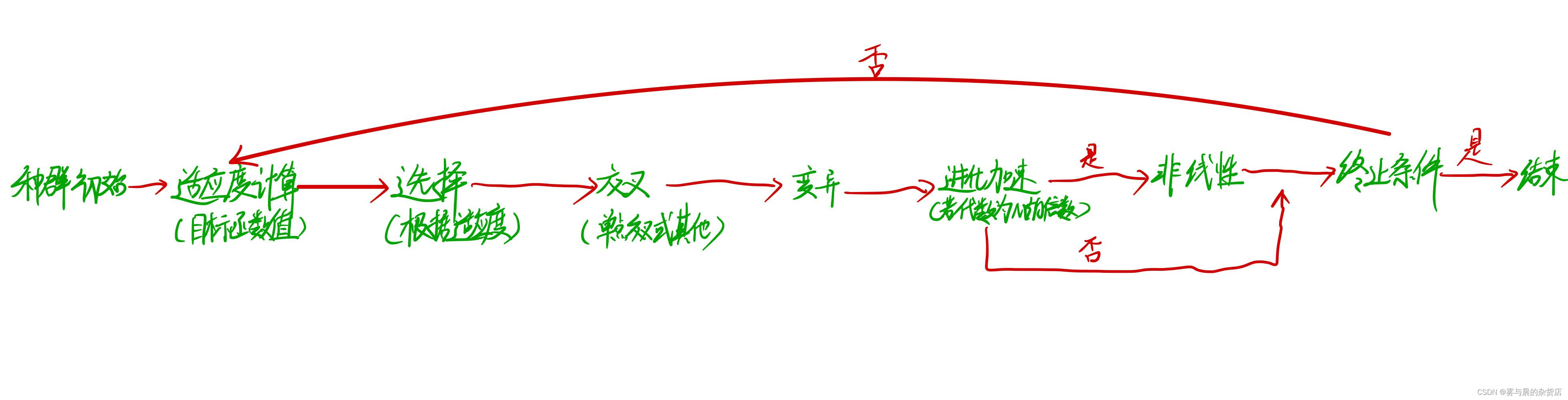
3.2 算法基本思路和流程
3.3 程序的实现
3.3.1 适应度函数
个体的适应度值为目标和函数的倒数(因为求的是最小值),适应度函数如下:
function y = fun(x)
y=-5*sin(x(1))*sin(x(2))*sin(x(3))*sin(x(4))*sin(x(5))-sin(5*x(1))*sin(5*x(2))*sin(5*x(3))*sin(5*x(4))*sin(5*x(5))+8;3.3.2 选择操作
选择操作是从旧群体中以一定的概率选择优良个体组成新的种群,以繁殖得到下一代个体。个体被选中的概率和适应度息息相关,个体的适应度值越高,被选择的概率就越高。
选择操作采用的是轮盘赌法(前文有体现)从种群中选择适应度好的个体组成新的种群,选择操作如下:
function ret=Select(individuals,sizepop)
% 本函数对每一代种群中的染色体进行选择,以进行后面的交叉和变异
% individuals input : 种群信息
% sizepop input : 种群规模
% opts input : 选择方法的选择
% ret output : 经过选择后的种群
individuals.fitness= 1./(individuals.fitness);
sumfitness=sum(individuals.fitness);
sumf=individuals.fitness./sumfitness;
index=[];
for i=1:sizepop %转sizepop次轮盘
pick=rand;
while pick==0
pick=rand;
end
for j=1:sizepop
pick=pick-sumf(j);
if pick<0
index=[index j];%这里记录下了每一行个体的位置,当pick小于sumf(j)(概率)时候会记录。
break; %寻找落入的区间,此次转轮盘选中了染色体i,注意:在转sizepop次轮盘的过程中,有可能会重复选择某些染色体,这里函数的意思是:当pick的值小于对应的sumf(j)的值则落入第j列,记录下j给index,这其中能体现选择意图,及适应度大的个体将会被选择。
end
end
end
individuals.chrom=individuals.chrom(index,:);
individuals.fitness=individuals.fitness(index);
ret=individuals;代码解析:选择函数首先将种群的适应度计算出来,通过适应度的大小选择个体。其中函数的适应度通过先前的fun函数计算出来,并通过求得个体函数值占群体适应度函数值的大小(概率)来进行选择操作,选择操作的部分思考如下:选择操作是通过最多sizepop*sizepop次循环来确定选择记录的个体,sizepop次循环来确定pick的值,在每次的pick值下,进行最多sizepop次概率的比较,倘若pick的值小于sumf(j)(这里为什么是小于,可以想象成pick的数据被概率所包围所以事件生成)则记录下该种群的位置。所以最后index为[1,sizepop]的矩阵。然后最后将该优化后的种群代替原先的种群。并把他们的适应度付给他们每个个体以便于后续的交叉和变异。
3.3.3 交叉操作
在先前外面已经进行过优选,现在进行交叉操作。交叉操作是从种群中选择两个个体,按照一定的概率交叉得到新的个体,两个个体的染色体通过交叉组合,把父串的优秀特征(这里指的是前面的优选)遗传给子串,从而产生新的子串,从而产生新的优秀个体。
本次个体采用的是实数编码,所以交叉操作采用实数交叉法,第k个染色体Ak和第l个染色体Al在j位上的交叉操作方法为:
Akj=Akj(1-b)+Alj*b
Alj=Alj(1-b)+Akj*b
代码如下:
function ret=Cross(pcross,lenchrom,chrom,sizepop,bound)
%本函数完成交叉操作
% pcorss input : 交叉概率
% lenchrom input : 染色体的长度
% chrom input : 染色体群
% sizepop input : 种群规模
% ret output : 交叉后的染色体
for i=1:sizepop
% 随机选择两个染色体进行交叉
pick=rand(1,2);
while prod(pick)==0
pick=rand(1,2);
end
index=ceil(pick.*sizepop);
% 交叉概率决定是否进行交叉
pick=rand;
while pick==0
pick=rand;
end
if pick>pcross
continue;
end
flag=0;
while flag==0
% 随机选择交叉位置
pick=rand;
while pick==0
pick=rand;
end
pos=ceil(pick.*sum(lenchrom)); %随机选择进行交叉的位置,即选择第几个变量进行交叉,注意:两个染色体交叉的位置相同
pick=rand; %交叉开始
v1=chrom(index(1),pos);
v2=chrom(index(2),pos);
chrom(index(1),pos)=pick*v2+(1-pick)*v1;
chrom(index(2),pos)=pick*v1+(1-pick)*v2; %交叉结束
flag1=test(lenchrom,bound,chrom(index(1),:)); %检验染色体1的可行性
flag2=test(lenchrom,bound,chrom(index(2),:)); %检验染色体2的可行性
if flag1*flag2==0
flag=0;
else flag=1;
end %如果两个染色体不是都可行,则重新交叉
end
end
ret=chrom;
代码流程讲解:首先是确定好交叉发生的位置,即包括行和列,第一步是行。通过rand(1,2)生成一行两列的随机数赋予pick,假设无0元素出现则进行下一操作:将pick*sizepop得到个体发生交叉的位置(行),接着判断是否发生交叉,再重新生成一个无0的pick判断是否是否大于pc概率若大于则不进行交叉,当pick落入概率区间的时候,交叉开始,又生成一个无0的pick确定好交叉的位置。接着交叉开始,将交叉其一的染色体的值赋予v1,其二的值赋予v2。通过先前的交叉函数计算出交叉后的函数值。然后这里面还嵌套了一个test函数主要是为了判断交叉后的数值是否超出了自变量的范围,如果超出了。则重新进行交叉。依次循环最后生成结果选择和交叉的种群。
3.3.4 变异操作
变异操作的主要目的是为了维持种群的多样性。变异操作主要是从种群中随机选择一个个体并在个体中的某一点进行变异以产生更优秀的个体。基因的写操作函数如下:
当r大于等于0.5的时候:aij=aij+(aij-amax)*f(g)
当r小于0.5的时候:aij=aij+(amin-aij)*f(g)
其中f(g)=r2*(1-g/gmax)^2
参数解释:aij为群体第i行第j列的自变量值;amax为自变量的上限,同理amin为下线。r2为0~1的随机参数,g为当前代数,gmax为进化的最大代数。
代码操作:
function ret=Mutation(pmutation,lenchrom,chrom,sizepop,pop,bound)
% 本函数完成变异操作
% pcorss input : 变异概率
% lenchrom input : 染色体长度
% chrom input : 染色体群
% sizepop input : 种群规模
% pop input : 当前种群的进化代数和最大的进化代数信息
% ret output : 变异后的染色体
for i=1:sizepop
% 随机选择一个染色体进行变异
pick=rand;
while pick==0
pick=rand;
end
index=ceil(pick*sizepop);
% 变异概率决定该轮循环是否进行变异
pick=rand;
if pick>pmutation
continue;
end
flag=0;
while flag==0
% 变异位置
pick=rand;
while pick==0
pick=rand;
end
pos=ceil(pick*sum(lenchrom)); %随机选择了染色体变异的位置,即选择了第pos个变量进行变异
v=chrom(i,pos); %第i个个体的第pos个位置发生了变异
v1=v-bound(pos,1);
v2=bound(pos,2)-v;%这里表示的是v1是距离自变量下限的距离,v2是指的是距离自变量上线的距离。v1+v2=2.8274
pick=rand; %变异开始
if pick>0.5
delta=v2*(1-pick^((1-pop(1)/pop(2))^2));
chrom(i,pos)=v+delta;
else
delta=v1*(1-pick^((1-pop(1)/pop(2))^2));
chrom(i,pos)=v-delta;
end %变异结束
flag=test(lenchrom,bound,chrom(i,:)); %检验染色体的可行性
end
end
ret=chrom;
代码介绍:其实变异函数前半部分和交叉函数其实是相差不大。都是为了寻找到变异的位置,即群体中某行某列的位置发生了变异,并将此位置记录在index中,最后是在index的指引下找到变异的位置,最后通过rand对变异函数进行选择,当pick大于0.5的时候选择函数1,反之函数2。最后变异结束返回种群。
3.3.5 算法主函数
%% 清空环境
clc
clear
warning off
%% 遗传算法参数
maxgen=30; %进化代数
sizepop=100; %种群规模
pcross=[0.6]; %交叉概率
pmutation=[0.01]; %变异概率
lenchrom=[1 1 1 1 1]; %变量字串长度
bound=[0 0.9*pi;0 0.9*pi;0 0.9*pi;0 0.9*pi;0 0.9*pi]; %变量范围
%% 个体初始化
individuals=struct('fitness',zeros(1,sizepop), 'chrom',[]); %种群结构体
avgfitness=[]; %种群平均适应度
bestfitness=[]; %种群最佳适应度
bestchrom=[]; %适应度最好染色体
% 初始化种群
for i=1:sizepop
individuals.chrom(i,:)=Code(lenchrom,bound); %随机产生个体
x=individuals.chrom(i,:);
individuals.fitness(i)=fun(x); %个体适应度
end
%找最好的染色体
[bestfitness bestindex]=min(individuals.fitness);
bestchrom=individuals.chrom(bestindex,:); %最好的染色体
avgfitness=sum(individuals.fitness)/sizepop; %染色体的平均适应度
% 记录每一代进化中最好的适应度和平均适应度
trace=[];
%% 进化开始
for i=1:maxgen
% 选择操作
individuals=Select(individuals,sizepop);
avgfitness=sum(individuals.fitness)/sizepop;
% 交叉操作
individuals.chrom=Cross(pcross,lenchrom,individuals.chrom,sizepop,bound);
% 变异操作
individuals.chrom=Mutation(pmutation,lenchrom,individuals.chrom,sizepop,[i maxgen],bound);
if mod(i,10)==0
individuals.chrom=nonlinear(individuals.chrom,sizepop);
end
% 计算适应度
for j=1:sizepop
x=individuals.chrom(j,:);
individuals.fitness(j)=fun(x);
end
%找到最小和最大适应度的染色体及它们在种群中的位置
[newbestfitness,newbestindex]=min(individuals.fitness);
[worestfitness,worestindex]=max(individuals.fitness);
% 代替上一次进化中最好的染色体
if bestfitness>newbestfitness
bestfitness=newbestfitness;
bestchrom=individuals.chrom(newbestindex,:);
end
individuals.chrom(worestindex,:)=bestchrom;
individuals.fitness(worestindex)=bestfitness;
avgfitness=sum(individuals.fitness)/sizepop;
trace=[trace;avgfitness bestfitness]; %记录每一代进化中最好的适应度和平均适应度
end
%进化结束
%% 结果显示
figure
[r c]=size(trace);
plot([1:r]',trace(:,1),'r-',[1:r]',trace(:,2),'b--');
title(['函数值曲线 ' '终止代数=' num2str(maxgen)],'fontsize',12);
xlabel('进化代数','fontsize',12);ylabel('函数值','fontsize',12);
legend('各代平均值','各代最佳值','fontsize',12);
ylim([1.5 8])
disp('函数值 变量');
% 窗口显示
disp([bestfitness x]);
grid on
























 4431
4431











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








