创新性应用:
我所从事的是生物信息学方向的研究及应用工作,生物信息学本身是由多学科交叉而形成的,是用以来解决越来越多的生物学数据积累和数据挖掘等方面问题而产生的。因此,对我所从事的整个方向的工作来说,引入数据库技术来解决一些问题,本身就是一个创新。
生物信息学数据的产出在不断加速,数据类型和内容也更加丰富,传统的生物学研究受到了极大的挑战。截止到2004年11月23日,世界上共有1225个在研的基因组相关项目,529个原核生物的基因组数据库、461个真核生物的基因组数据库,这中间已经公开的有232个。到2005年初GenBank数据库中已经有44.6亿碱基数据,4000万条基因序列,包括人类、小鼠、大鼠、等哺乳动物;拟南芥、水稻等植物;鱼类;昆虫类;线虫类;酵母等真菌类;各种原核生物;以及病毒等,包含了生物的各个进化分支。
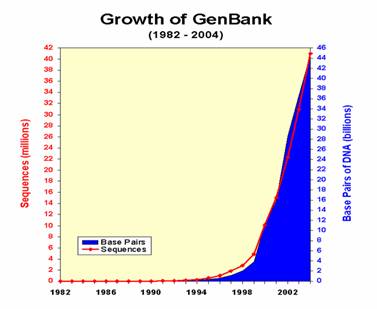
急剧积累的数据给我们的带来解开生命之谜的希望,同时也带来了存储、计算、分析等多方面的挑战,传统的生物学研究已经无法再胜任这其中的任何一项工作。必须要与其它学科交叉,引入其它学科,特别是数据、计算机等计算、信息相关的学科中的技术来帮助解决所遇到的问题。面对现状,首要解决的问题就是数据的存储、信息的提取问题。在很大程度上,这项工作将依赖于数据库技术。目前世界上建立了三大核酸数据库(NCBI、EMBL、DDBJ)用以存放最为原始的核酸信息,以这个三个数据库为基础又相继建立起了二级、三级等多类数据库。相关的管理和研究机构一方面在数据库基础上开发了大量的应用实例,同时又提供了数据库的接口类使得我们能够自主来进行数据分析工作。
在另一方面,一些大的IT公司,例如IBM、Oracle都在为生物信息学的研究工作而提供解决方案。特别是Oracle公司,利用新版的ODM中提供了Blast(生物信息学研究中最常用的序列比对的工具),今后我们可以非常容易的在RDBMS中进行序列比对工作,这一切不需要从数据库中再取出数据。另一方面Oracle还提供了完整的“Oracle 生命科学平台”平台方案,由 Oracle 数据库 10g、Oracle 应用服务器 10g 和协作套件中的一些功能组成,它解决了生命科学中的主要 IT 问题,其中包括分布式数据、集成各种数据类型、管理大量数据、安全协作以及查找方式和数据挖掘等。
还有一点,目前越来越多的生物学数据库被开发出来,不仅仅是由于生物学数据需要存储,更为重要的一点是存储的这些数据对于后面的研究将有非常大的帮助。因此在数据库之上,不同的研究者都在努力的开发着新的分析工具,用以更为高效的来分析这些数据。
对数据库来说,大部分的提到的工作都是前所未有的,它们被一项项的提出并实现,这不仅仅是一种需求与开发的关系,而是在两个学科交叉后,不同学科背景的人员在一起进行的创新性的工作。
行业借鉴经验:
生物学数据库对于生物学的发展是具有重要的意义的。传统的实验室的“湿”科学转向以信息为研究对象的“干”科学,是在上世纪末,随着信息技术以及生命科学的几次突破而必然产生的结果。以往,一个实验室可能只研究一个物种的一个或者几个基因,而且常常是一研究几年,甚至几十年,但信息社会中,这种速度是非常慢的。新的技术手段使得生物学的分析平台,能够在短短的几天的时间内完成几万甚至上亿条序列的分析,这在以往是不可以想象的。而构建这样一个平台,首先的第一步就是构建平台运行相关的数据库。
因为现在的研究是以大规模的测序以及工厂化的实验产出的数据为目标的。就像我们利用数据库来存储各类商品信息一样,我们生物学数据按照不同的意义进行分类,分别建立不同数据之间的关系,再由这么关系出发,通过专门的算法以及软件来分析数据总体的趋势、数据中所蕴含的信息、数据最终所代表的含义。知道这些之后,我们再反过来重新进到实验室里来对我们的分析结果进行验正,再用验正的结果来修正我们的分析过程。这将是一个循序渐进的过程,通过这个过程我们的生物学研究才能够不断进步,同时我们的数据库技术、信息技术才能够不断的改进。
从另外一方面来分析,我们可以看到数据库技术本身的发展也正是由于各方面信息的不断积累,以及人类对信息处理的要求。从最简单的链表,到现在复杂的关系性数据库,数据库技术之所以不断的再改进也正是因为信息处理的要求的断改进。如今面对生物信息学的海量数据,数据库技术同样也需要有所改进,例如Oracle10g所支持的网格技术、所提供的数据库内部的Blast功能等等。
这些改进不仅对生物信息学研究是有价值的,同样对于我们其它许多应用都是有潜在的价值的。生物信息数据的挖掘算法和软件对于环境分析同样的适用,对于疾病的病理病因研究也有巨大的应用前景。同样,在生物信息学的研究过程中,我们建立的各类生物信息学数据库对于我们的生产、生活也有着潜在的经济效益。例如我们建立的“虚拟分子育种数据库”能够在线提供多物种的分子虚拟育种,这种虚拟育种能够使我们有的放矢,通过指定的条件来帮助我们确定准确的育种条件的育种后的作物生长条件。此外还有我们建立的各种蛋白质库,能够在我们未来的药物筛选的过程中为我们提供准备的目标群,从而对于药物研发、药物设计以及疾病治疗都提供帮助。
对于生物信息学本身来说,是一个新兴的交叉学科,它的深入以及发展都是借助于其它学科的技术手段,许多的分析工具、实验仪器、数据库平台都是各个不同学科融合的成果。由此可以看出,在生物信息学这里,各个学科是互为补充、相互促进的关系。生物信息学的遇到问题促使其它的科学不断的提供新的技术方法,新的技术方法的引入使得生物信息学不断向前发展。
应用难点技巧:
构建生物信息学数据库平台与构建普通的数据库应用有一定的区别。普通的数据库应用,我们关注的是数据本身的意义,因此可以直接将全部的数据抽象成几类数据类型,对这些数据类型我们再进行数据库设计。而生物信息学数据库平台最终所关注的不是数据本身,而是数据所蕴含的信息以及不同数据关系所组合成的有意义的内容。所以在进行生物信息学数据库设计时,我们需要关注的除了数据之外,更多的是这些数据的生物学意义。很有可能它们其中的某些意义就是解决生命意义的关键环节。
另外一点,目前的应用也具有一定的共同性,例如我们构建的网格平台。目前有生物信息学应用网格,也有药物研发网格,有气象网格,天文网格等等。技术对于不同的应用领域来说并没有太大的区别,区别的本身在于技术以什么样的形态切入到领域中去。对于我们所关心的数据库技术是同样的道理。在普通的数据库应用中,数据库是以数据存储和管理的职能而被应用的。而在生物信息学领域中呢,数据库不但要起到数据库存储和管理的作用,它还需要具有数据库仓库的特点,同时还需要具有数据库挖掘的部分功能,甚至需要内置一些分析过程、算法等。
在构建我们的生物信息应用网格的过程中,如何让数据库能够在网格上提供服务是困扰我们很久的问题。由于网格服务的形式以及特点,大部分的数据库无法直接提供针对网格的服务。在前期针对我们常用的数据库,我们采用的办法是分解数据库操作动作,对这些动作我们进行分类以及抽象。然后为每一个抽象出来的动作构建一个小的网格服务,这些服务之间是独立的,服务本身通过网格中间件所提供工具来与不同的数据库服务器建立可信连接。相当于在以前的数据库与应用层之间多了一层数据库操作服务。这样全部的数据库可以被隐藏到后台,用户在使用时只需要告诉网格系统你需要什么样的服务,余下的工作交由网格上的数据库操作服务来处理。这个办法对于我们其它几类无法直接运行于网格上的软件也同样适用。
在我们处理生物信息学数据的时候,还遇到另外一种情况。不同的研究中心采用的数据标准不一样,导致了数据处理起来非常麻烦。针对这个问题,我们专门提供了一个数据库标准转换的服务。我们分析了绝大部分的数据标准,为这些标准两两之间建立了数据字典,这样不同的标准之间需要转换时,我们先将数据按我们自己定义的标准生成为XML格式的数据,再将这个数据按照要求的标准进行转换。这样可以降低手工转换操作的出错几率。
后记:对于数据库,我目前关注的重点在于如何让它能够存储更多的数据,如何能够让数据库更有效率,如何能够方便的提供面向网格的服务,以及如何让数据库提供更多实用的功能。我对数据库的接触也只限于应用开发和简单的数据库管理等,因此我需要学的东西还非常多。在论述的过程中有不当之处也请各位老师谅解。也希望能够对我所从事的这些工作提供宝贵的建议。
我所从事的是生物信息学方向的研究及应用工作,生物信息学本身是由多学科交叉而形成的,是用以来解决越来越多的生物学数据积累和数据挖掘等方面问题而产生的。因此,对我所从事的整个方向的工作来说,引入数据库技术来解决一些问题,本身就是一个创新。
生物信息学数据的产出在不断加速,数据类型和内容也更加丰富,传统的生物学研究受到了极大的挑战。截止到2004年11月23日,世界上共有1225个在研的基因组相关项目,529个原核生物的基因组数据库、461个真核生物的基因组数据库,这中间已经公开的有232个。到2005年初GenBank数据库中已经有44.6亿碱基数据,4000万条基因序列,包括人类、小鼠、大鼠、等哺乳动物;拟南芥、水稻等植物;鱼类;昆虫类;线虫类;酵母等真菌类;各种原核生物;以及病毒等,包含了生物的各个进化分支。

急剧积累的数据给我们的带来解开生命之谜的希望,同时也带来了存储、计算、分析等多方面的挑战,传统的生物学研究已经无法再胜任这其中的任何一项工作。必须要与其它学科交叉,引入其它学科,特别是数据、计算机等计算、信息相关的学科中的技术来帮助解决所遇到的问题。面对现状,首要解决的问题就是数据的存储、信息的提取问题。在很大程度上,这项工作将依赖于数据库技术。目前世界上建立了三大核酸数据库(NCBI、EMBL、DDBJ)用以存放最为原始的核酸信息,以这个三个数据库为基础又相继建立起了二级、三级等多类数据库。相关的管理和研究机构一方面在数据库基础上开发了大量的应用实例,同时又提供了数据库的接口类使得我们能够自主来进行数据分析工作。
在另一方面,一些大的IT公司,例如IBM、Oracle都在为生物信息学的研究工作而提供解决方案。特别是Oracle公司,利用新版的ODM中提供了Blast(生物信息学研究中最常用的序列比对的工具),今后我们可以非常容易的在RDBMS中进行序列比对工作,这一切不需要从数据库中再取出数据。另一方面Oracle还提供了完整的“Oracle 生命科学平台”平台方案,由 Oracle 数据库 10g、Oracle 应用服务器 10g 和协作套件中的一些功能组成,它解决了生命科学中的主要 IT 问题,其中包括分布式数据、集成各种数据类型、管理大量数据、安全协作以及查找方式和数据挖掘等。
还有一点,目前越来越多的生物学数据库被开发出来,不仅仅是由于生物学数据需要存储,更为重要的一点是存储的这些数据对于后面的研究将有非常大的帮助。因此在数据库之上,不同的研究者都在努力的开发着新的分析工具,用以更为高效的来分析这些数据。
对数据库来说,大部分的提到的工作都是前所未有的,它们被一项项的提出并实现,这不仅仅是一种需求与开发的关系,而是在两个学科交叉后,不同学科背景的人员在一起进行的创新性的工作。
行业借鉴经验:
生物学数据库对于生物学的发展是具有重要的意义的。传统的实验室的“湿”科学转向以信息为研究对象的“干”科学,是在上世纪末,随着信息技术以及生命科学的几次突破而必然产生的结果。以往,一个实验室可能只研究一个物种的一个或者几个基因,而且常常是一研究几年,甚至几十年,但信息社会中,这种速度是非常慢的。新的技术手段使得生物学的分析平台,能够在短短的几天的时间内完成几万甚至上亿条序列的分析,这在以往是不可以想象的。而构建这样一个平台,首先的第一步就是构建平台运行相关的数据库。
因为现在的研究是以大规模的测序以及工厂化的实验产出的数据为目标的。就像我们利用数据库来存储各类商品信息一样,我们生物学数据按照不同的意义进行分类,分别建立不同数据之间的关系,再由这么关系出发,通过专门的算法以及软件来分析数据总体的趋势、数据中所蕴含的信息、数据最终所代表的含义。知道这些之后,我们再反过来重新进到实验室里来对我们的分析结果进行验正,再用验正的结果来修正我们的分析过程。这将是一个循序渐进的过程,通过这个过程我们的生物学研究才能够不断进步,同时我们的数据库技术、信息技术才能够不断的改进。
从另外一方面来分析,我们可以看到数据库技术本身的发展也正是由于各方面信息的不断积累,以及人类对信息处理的要求。从最简单的链表,到现在复杂的关系性数据库,数据库技术之所以不断的再改进也正是因为信息处理的要求的断改进。如今面对生物信息学的海量数据,数据库技术同样也需要有所改进,例如Oracle10g所支持的网格技术、所提供的数据库内部的Blast功能等等。
这些改进不仅对生物信息学研究是有价值的,同样对于我们其它许多应用都是有潜在的价值的。生物信息数据的挖掘算法和软件对于环境分析同样的适用,对于疾病的病理病因研究也有巨大的应用前景。同样,在生物信息学的研究过程中,我们建立的各类生物信息学数据库对于我们的生产、生活也有着潜在的经济效益。例如我们建立的“虚拟分子育种数据库”能够在线提供多物种的分子虚拟育种,这种虚拟育种能够使我们有的放矢,通过指定的条件来帮助我们确定准确的育种条件的育种后的作物生长条件。此外还有我们建立的各种蛋白质库,能够在我们未来的药物筛选的过程中为我们提供准备的目标群,从而对于药物研发、药物设计以及疾病治疗都提供帮助。
对于生物信息学本身来说,是一个新兴的交叉学科,它的深入以及发展都是借助于其它学科的技术手段,许多的分析工具、实验仪器、数据库平台都是各个不同学科融合的成果。由此可以看出,在生物信息学这里,各个学科是互为补充、相互促进的关系。生物信息学的遇到问题促使其它的科学不断的提供新的技术方法,新的技术方法的引入使得生物信息学不断向前发展。
应用难点技巧:
构建生物信息学数据库平台与构建普通的数据库应用有一定的区别。普通的数据库应用,我们关注的是数据本身的意义,因此可以直接将全部的数据抽象成几类数据类型,对这些数据类型我们再进行数据库设计。而生物信息学数据库平台最终所关注的不是数据本身,而是数据所蕴含的信息以及不同数据关系所组合成的有意义的内容。所以在进行生物信息学数据库设计时,我们需要关注的除了数据之外,更多的是这些数据的生物学意义。很有可能它们其中的某些意义就是解决生命意义的关键环节。
另外一点,目前的应用也具有一定的共同性,例如我们构建的网格平台。目前有生物信息学应用网格,也有药物研发网格,有气象网格,天文网格等等。技术对于不同的应用领域来说并没有太大的区别,区别的本身在于技术以什么样的形态切入到领域中去。对于我们所关心的数据库技术是同样的道理。在普通的数据库应用中,数据库是以数据存储和管理的职能而被应用的。而在生物信息学领域中呢,数据库不但要起到数据库存储和管理的作用,它还需要具有数据库仓库的特点,同时还需要具有数据库挖掘的部分功能,甚至需要内置一些分析过程、算法等。
在构建我们的生物信息应用网格的过程中,如何让数据库能够在网格上提供服务是困扰我们很久的问题。由于网格服务的形式以及特点,大部分的数据库无法直接提供针对网格的服务。在前期针对我们常用的数据库,我们采用的办法是分解数据库操作动作,对这些动作我们进行分类以及抽象。然后为每一个抽象出来的动作构建一个小的网格服务,这些服务之间是独立的,服务本身通过网格中间件所提供工具来与不同的数据库服务器建立可信连接。相当于在以前的数据库与应用层之间多了一层数据库操作服务。这样全部的数据库可以被隐藏到后台,用户在使用时只需要告诉网格系统你需要什么样的服务,余下的工作交由网格上的数据库操作服务来处理。这个办法对于我们其它几类无法直接运行于网格上的软件也同样适用。
在我们处理生物信息学数据的时候,还遇到另外一种情况。不同的研究中心采用的数据标准不一样,导致了数据处理起来非常麻烦。针对这个问题,我们专门提供了一个数据库标准转换的服务。我们分析了绝大部分的数据标准,为这些标准两两之间建立了数据字典,这样不同的标准之间需要转换时,我们先将数据按我们自己定义的标准生成为XML格式的数据,再将这个数据按照要求的标准进行转换。这样可以降低手工转换操作的出错几率。
后记:对于数据库,我目前关注的重点在于如何让它能够存储更多的数据,如何能够让数据库更有效率,如何能够方便的提供面向网格的服务,以及如何让数据库提供更多实用的功能。我对数据库的接触也只限于应用开发和简单的数据库管理等,因此我需要学的东西还非常多。在论述的过程中有不当之处也请各位老师谅解。也希望能够对我所从事的这些工作提供宝贵的建议。






















 5911
5911

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








