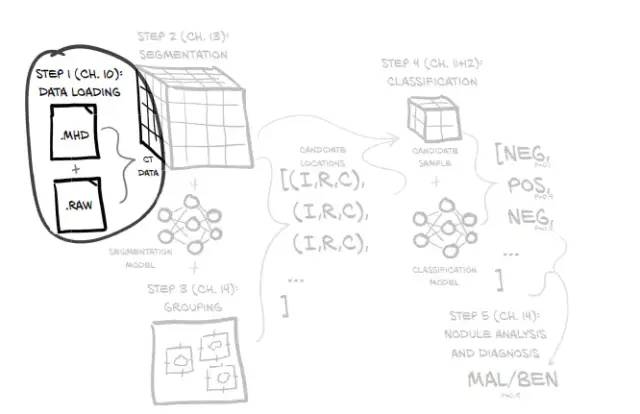
这部分内容主要是数据集准备,加载原始CT扫描数据,并将其转换为可以使用PyTorch处理的数据格式。
目录
5.3、setdefault()方法构造value值为列表/字典的字典
一、主要内容与实际工作
1、主要内容
- 加载和处理原始数据文件
- 实现一个Python类来表示我们的数据
- 将数据转换为PyTorch可用的格式
- 可视化训练和验证数据
2、实际工作
数据集准备需要完成以下几个工作:
- 读取annotations.csv内容;
- 读取candidates.csv内容;
- 构造Ct类,用于根据输入的series_uid,获取该uid的CT数据的信息。
- 构造Dataset类,用于加载数据集。
二、代码解析
按照原文代码顺序解析每一部分代码。有问题欢迎讨论。
1、logging模块
使用log.info( )打印日志
前面要加上下面的代码
import logging
log = logging.getLogger(__name__)
# log.setLevel(logging.WARN)
# log.setLevel(logging.INFO)
log.setLevel(logging.DEBUG)2、diskcache缓存
相关的函数在文档util/disk下
代码中用到了diskcache库,用于将CT数据解析后缓存到磁盘中,使用缓存可以较大的提高训练时数据加载速度。
from util.disk import getCache
raw_cache = getCache('part2ch10_raw')3、namedtuple 具名元组
因为元组的局限性:不能为元组内部的数据进行命名,所以往往我们并不知道一个元组所要表达的意义,所以在这里引入了 collections.namedtuple 这个工厂函数,来构造一个带字段名的元组。
namedtuple(typename, field_names)
typename:元组名称。
field_names:元组中元素的名称;可以是有多个字符串组成的可迭代对象,或者是有空格分隔开的字段名组成的字符串。
from collections import namedtuple
CandidateInfoTuple = namedtuple(
'CandidateInfoTuple',
'isNodule_bool, diameter_mm, series_uid, center_xyz',
)4、functools.lru_cache 缓存机制
这是一项优化技术,把耗时的函数的结果保存起来,避免了传入相同的参数时重复计算。
@functools.lru_cache(maxsize=None, typed=False)
maxsize: 表示存储多少个调用的结果。
typed:如果设置为True,会把不同的类型分开存储,比如说通常认为1.0和1是一样的结果,但是他们类型不同,一个是浮点数,一个数整数。
@functools.lru_cache(1) # 缓存一次调用结果5、getCandidateInfoList函数
处理标注数据:annotations.csv和candidates.csv文件,分别存到diameter_list和candidateInfo_list,最后返回candidateInfo_list. 由candidateInfoTuple构成的list,包含uid,坐标(x,y,z),直径,分类(是否结节)
5.1、glob.glob()函数
glob.glob(pathname, *, recursive=False)
功能:返回一个某一种文件夹下面的某一类型文件路径列表
5.2、os.path.split('PATH')
1.PATH指一个文件的全路径作为参数:
2.如果给出的是一个目录和文件名,则输出路径和文件名
3.如果给出的是一个目录名,则输出路径和为空文件名
[0] 返回的是输出路径, [1]返回的是文件名,经测试[-1]返回的也是文件名
所以代码中os.path.split(p)[-1][:-4] 返回的就是 所有的文件名,即uid
5.3、setdefault()方法构造value值为列表/字典的字典
dic.setdefault(key,[ ]).append(value)
dic = {}
dic.setdefault('a',[]).append(1)
dic.setdefault('a',[]).append(2)
print(dic)
# {'a': [1, 2]}5.4、结节处理
# annotations.csv: 记录实际结节。文件结构: uid, x, y, z, diameter # candidates.csv: 记录候选结节。文件结构: uid, x, y, z, class 将两个csv文件合并,得到candidateInfo_list,包含uid,x,y,z,diameter,class 如果candidate中的xyz坐标和annotation中的xyz坐标偏差大于半径的一半, 则认为它们不是同一个节点,将直接用零代替。
def getCandidateInfoList(requireOnDisk_bool=True):
# We construct a set with all series_uids that are present on disk.
# This will let us use the data, even if we haven't downloaded all of
# the subsets yet.
"""
加载annotations.csv和candidates.csv,分别存到diameter_list和candidateInfo_list
:param requireOnDisk_bool. 如果文件不存在,是否跳过
:return candidateInfo_list. 由candidateInfoTuple构成的list
"""
mhd_list = glob.glob('data-unversioned/part2/luna/subset*/*.mhd')
presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list} # 提取所有文件名,即uid
diameter_dict = {}
with open('data/part2/luna/annotations.csv', "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0]
annotationCenter_xyz = tuple([float(x) for x in row[1:4]])
annotationDiameter_mm = float(row[4])
diameter_dict.setdefault(series_uid, []).append(
(annotationCenter_xyz, annotationDiameter_mm)
)
candidateInfo_list = []
with open('data/part2/luna/candidates.csv', "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0]
# 如果series_uid不存在,那么它属于一个数据未存储在磁盘的子集,所以我们应该跳过它。
if series_uid not in presentOnDisk_set and requireOnDisk_bool:
continue
isNodule_bool = bool(int(row[4]))
candidateCenter_xyz = tuple([float(x) for x in row[1:4]])
# 如果candidate中的xyz坐标和annotation中的xyz坐标偏差大于半径的一半,
# 则认为它们不是同一个节点,将直接用零代替,即认为这不是结节
candidateDiameter_mm = 0.0
for annotation_tup in diameter_dict.get(series_uid, []):
annotationCenter_xyz, annotationDiameter_mm = annotation_tup
for i in range(3):
delta_mm = abs(candidateCenter_xyz[i] - annotationCenter_xyz[i])
if delta_mm > annotationDiameter_mm / 4:
break
else:
candidateDiameter_mm = annotationDiameter_mm
break
candidateInfo_list.append(CandidateInfoTuple(
isNodule_bool,
candidateDiameter_mm,
series_uid,
candidateCenter_xyz,
))
candidateInfo_list.sort(reverse=True)
return candidateInfo_list
6、CT类
获取CT影像数据,从CT扫描中取出一个结节。其中涉及了坐标系的转换。
6.1、SimpleITK
用SampleSTK包可直接读取CT扫描数据,可通过【conda install simpleitk】命令安装。
6.2、clip
np.clip(a, a_min, a_max, out=None)
CT文件中数据单位为HU(HounsField Units,亨氏单位)。其中:
空气:-1000HU,水:0HU,骨骼:1000HU
因此超出-1000HU到1000HU外的数据并不是我们需要关心的数据,可强制转换为限值。
6.3、CT.getRawCandidate函数
输入:center_xyz: 结节的xyz坐标
width_irc: 体素宽度,也是数据集输入到模型的输入尺寸
输出:ct_chunk: 结节包含的体素块的HU值,array
center_irc: 结节的病人坐标信息(i,r,c)
坐标转换 (x,y,z) 到 (i,r,c):以毫米为单位的坐标称为(X,Y,Z)坐标,以体素为单位的坐标称为(I,R,C)坐标,这部分util中给出了代码,感兴趣的可以研究一下。
IrcTuple = collections.namedtuple('IrcTuple', ['index', 'row', 'col'])
XyzTuple = collections.namedtuple('XyzTuple', ['x', 'y', 'z'])
def irc2xyz(coord_irc, origin_xyz, vxSize_xyz, direction_a):
cri_a = np.array(coord_irc)[::-1]
origin_a = np.array(origin_xyz)
vxSize_a = np.array(vxSize_xyz)
coords_xyz = (direction_a @ (cri_a * vxSize_a)) + origin_a
# coords_xyz = (direction_a @ (idx * vxSize_a)) + origin_a
return XyzTuple(*coords_xyz)
def xyz2irc(coord_xyz, origin_xyz, vxSize_xyz, direction_a):
origin_a = np.array(origin_xyz)
vxSize_a = np.array(vxSize_xyz)
coord_a = np.array(coord_xyz)
cri_a = ((coord_a - origin_a) @ np.linalg.inv(direction_a)) / vxSize_a
cri_a = np.round(cri_a)
return IrcTuple(int(cri_a[2]), int(cri_a[1]), int(cri_a[0]))CT类代码:
import SimpleITK as sitk
class Ct:
def __init__(self, series_uid):
mhd_path = glob.glob(
'data-unversioned/part2/luna/subset*/{}.mhd'.format(series_uid)
)[0]
# 用SampleSTK包可直接读取CT扫描数据
ct_mhd = sitk.ReadImage(mhd_path)
# HU: 亨氏单位,Hounsfield Unit.
# 空气为-1000 HU,约等于0 g/cm3. 水为0 HU,约等于1 g/cm3, 骨骼至少时1000HU,约等于2~3g/cm3
ct_a = np.array(sitk.GetArrayFromImage(ct_mhd), dtype=np.float32) # 读取到的数据单位为HU
# 将数据限定再-1000~1000 HU
# CTs are natively expressed in https://en.wikipedia.org/wiki/Hounsfield_scale
# HU are scaled oddly, with 0 g/cc (air, approximately) being -1000 and 1 g/cc (water) being 0.
# The lower bound gets rid of negative density stuff used to indicate out-of-FOV
# The upper bound nukes any weird hotspots and clamps bone down
ct_a.clip(-1000, 1000, ct_a)
self.series_uid = series_uid
self.hu_a = ct_a
self.origin_xyz = XyzTuple(*ct_mhd.GetOrigin()) # xyz坐标和irc坐标的原点偏移量
self.vxSize_xyz = XyzTuple(*ct_mhd.GetSpacing()) # 体素在xyz坐标轴的大小
self.direction_a = np.array(ct_mhd.GetDirection()).reshape(3, 3) # 体素方向矩阵,等于eye(3)
def getRawCandidate(self, center_xyz, width_irc):
"""
根据xyz坐标算出病人坐标irc。然后根据每个结节的irc和体素宽度,算出结节包含的体素块数据
:param center_xyz: 结节的xyz坐标
:param width_irc: 体素宽度,也是数据集输入到模型的输入尺寸
:return ct_chunk: 结节包含的体素块的HU值,array
:return center_irc: 结节的病人坐标信息
"""
center_irc = xyz2irc(
center_xyz,
self.origin_xyz,
self.vxSize_xyz,
self.direction_a,
)
slice_list = []
for axis, center_val in enumerate(center_irc):
start_ndx = int(round(center_val - width_irc[axis]/2))
end_ndx = int(start_ndx + width_irc[axis])
assert center_val >= 0 and center_val < self.hu_a.shape[axis], repr([self.series_uid, center_xyz, self.origin_xyz, self.vxSize_xyz, center_irc, axis])
if start_ndx < 0:
# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(
# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))
start_ndx = 0
end_ndx = int(width_irc[axis])
if end_ndx > self.hu_a.shape[axis]:
# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(
# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))
end_ndx = self.hu_a.shape[axis]
start_ndx = int(self.hu_a.shape[axis] - width_irc[axis])
slice_list.append(slice(start_ndx, end_ndx))
ct_chunk = self.hu_a[tuple(slice_list)]
return ct_chunk, center_irc7、性能优化方法
首先对getCt的结果进行了缓存,它会存在内存中,但是需要注意的是,在内存中只会缓存一个CT文件,如果频繁访问不同的CT文件就会导致大量的miss,这种缓存就没有太大意义了,所以我们处理的时候需要注意顺序。然后是getCtRawCandidate,获取的CT的数值会在磁盘中缓存,同时我们减少了数据的数量级,从而降低了读取压力。
@functools.lru_cache(1, typed=True) # 保留一次缓存结果
def getCt(series_uid):
return Ct(series_uid)
@raw_cache.memoize(typed=True) # 数据缓存到同路径的cache文件夹下
def getCtRawCandidate(series_uid, center_xyz, width_irc):
ct = getCt(series_uid)
ct_chunk, center_irc = ct.getRawCandidate(center_xyz, width_irc)
return ct_chunk, center_irc8、LunaDataset类
像做CIFAR数据一样,把它转换成Dataset数据集,方便我们使用同样的API。
符合Dataset数据集的要求,需要实现两个方法 _ _ len _ _ 和 _ _ getitem _ _。
getitem返回
返回指定索引对应的结节信息 :param ndx: 某个ct数据中的第ndx个结节索引 :return: candidate_t. 结节所包含的所有体素的三位数组。t代表数组时个tensor :return: post_t. 结节是否为肿瘤。0代表不是,1代表肿瘤。 :return: series_uid. ndx所对应的结节uid :return: center_irc. 结节的重心坐标。类型为tensor
init初始化分割训练集和验证集
val_stride:作为验证集时,从数据集中抽取样本作为验证集样本的步长。即每隔val_stride抽取一个样本作为验证集样本。 isValSet_bool:是否作为验证集。 series_uid:获取某个uid对应的所有样本。
class LunaDataset(Dataset):
def __init__(self,
val_stride=0,
isValSet_bool=None,
series_uid=None,
):
"""
val_stride:作为验证集时,从数据集中抽取样本作为验证集样本的步长。即每隔val_stride抽取一个样本作为验证集样本。
isValSet_bool:是否作为验证集。
series_uid:获取某个uid对应的所有样本。
"""
self.candidateInfo_list = copy.copy(getCandidateInfoList())
if series_uid:
self.candidateInfo_list = [
x for x in self.candidateInfo_list if x.series_uid == series_uid
]
if isValSet_bool:
assert val_stride > 0, val_stride
self.candidateInfo_list = self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
elif val_stride > 0:
del self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
log.info("{!r}: {} {} samples".format(
self,
len(self.candidateInfo_list),
"validation" if isValSet_bool else "training",
))
def __len__(self):
return len(self.candidateInfo_list)
def __getitem__(self, ndx):
"""
返回指定索引对应的结节信息
:param ndx: 某个ct数据中的第ndx个结节索引
:return: candidate_t. 结节所包含的所有体素的三位数组。t代表数组时个tensor
:return: post_t. 结节是否为肿瘤。0代表不是,1代表肿瘤。
:return: series_uid. ndx所对应的结节uid
:return: center_irc. 结节的重心坐标。类型为tensor
"""
candidateInfo_tup = self.candidateInfo_list[ndx]
width_irc = (32, 48, 48)
candidate_a, center_irc = getCtRawCandidate(
candidateInfo_tup.series_uid,
candidateInfo_tup.center_xyz,
width_irc,
)
candidate_t = torch.from_numpy(candidate_a)
candidate_t = candidate_t.to(torch.float32)
candidate_t = candidate_t.unsqueeze(0)
pos_t = torch.tensor([
not candidateInfo_tup.isNodule_bool,
candidateInfo_tup.isNodule_bool
],
dtype=torch.long,
)
return (
candidate_t,
pos_t,
candidateInfo_tup.series_uid,
torch.tensor(center_irc),
)三、总结
如果解析和加载例程的开销很大,那么缓存是十分有用的。
PyTorch数据集子类用于将数据从原生形式转换为适合传递给模型的张量。我们可以使用此功能将实际数据与PyTorch API集成在一起
Dataset的子类需要提供2个函数的实现:__len__()和__getitem__()。允许使用其他辅助方法,但不是必需的。
将我们的数据分成一个合理的训练集和验证集,要求确保没有样本同时存在于2个集合中。作者在这里通过先对样本排序,然后每隔val_stride个样本取一个样本来构建验证集。
写在后面
本文参考文章:
18 | 使用PyTorch完成医疗图像识别大项目:理解数据
这次按照代码顺序进行书写,然后给一些函数进行了解释,这么写下来看起来很乱。从以后开始打算介绍每个类和函数是在做什么,然后把其中涉及的函数单拿出来去解释,谢谢,欢迎大家讨论。























 1064
1064











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








