本文章是我在towards data science上发表文章《Neo4j for Diseases》的自译中译本。
注意:本文章并不提供医学建议。它只是提供信息。它并不能取代专业的医学建议,诊断和治疗。
目录
新冠疫情笼罩着全世界。疫情极大地影响着我们的生活。家庭破碎,经济损失,而且我们的日常行为可能也因此而永久改变。同时,疫情也让我们重新关注起公共健康这个问题。有一点是很清楚的,我们人类需要在医学研究上投资更多来避免下一次瘟疫灾情。

图1:使用Neo4j显示KEGG数据库中COVID-19条目,作者作图。
我在写完之前的文章《Analyzing Genomes in a Graph Database》后,发现其实京都基因和基因组百科全书(KEGG)还有很多的宝藏等着我们去发掘。其中之一就是其疾病数据库。该子数据库含有很多关于人类疾病方面的细节。结合其他的KEGG子数据库,例如基因组,药物和基因子数据库,我们可以构建一个关于疾病,致病生物和治疗药物的非常完备的知识网络。一如我之前的文章里所说的,Neo4j这样的图数据库非常适用于在生物医疗领域进行知识挖掘。而KEGG的疾病数据正好适用于这样一个项目。我从KEGG那里通过API下载了疾病等子数据库,导入到Neo4j里面,做了几项分析,并且发现了一些有趣的信息。
在这篇文章里,我集中分析疾病,致病生物和药物之间的关系。本项目的源代码放在了我Github的库上:https://github.com/dgg32/kegg_disease
1. 预处理并导入数据到Neo4j
你可以在data文件夹里找到本项目所需要的所有CSV文件。不过如果你想更新数据到最新的状态,请按照README.md里的指引行事。
在Neo4j官网那里下载Neo4j Desktop。下载之后,添加一个新的本地DMBS(“Local DMSBS”),称之为kegg_disease。通过点击...然后Open folder,打开其Import文件夹。将所有的CSV文件放到里面,并执行以下命令,导入数据:
LOAD CSV WITH HEADERS FROM 'file:///disease.csv' AS row MERGE (n:disease {name: row.name, ko: row.ko, description: row.description, disease_category:row.disease_category});
LOAD CSV WITH HEADERS FROM 'file:///drug.csv' AS row MERGE (n:drug {name: row.name, ko: row.ko});
LOAD CSV WITH HEADERS FROM 'file:///pathogen.csv' AS row MERGE (n:pathogen {name: row.name, ko: row.ko, taxonomy: row.taxonomy});
CREATE CONSTRAINT ON (n:disease) ASSERT n.ko IS UNIQUE;
CREATE CONSTRAINT ON (n:drug) ASSERT n.ko IS UNIQUE;
CREATE CONSTRAINT ON (n:pathogen) ASSERT n.ko IS UNIQUE;
LOAD CSV WITH HEADERS FROM 'file:///drug_disease.csv' AS row MERGE (n1:drug {ko: row.from}) MERGE (n2:disease {ko: row.to}) MERGE (n1)-[r:treats]->(n2);
LOAD CSV WITH HEADERS FROM 'file:///pathogen_disease.csv' AS row MERGE (n1:pathogen {ko: row.from}) MERGE (n2:disease {ko: row.to}) MERGE (n1)-[r:causes]->(n2);2. 快速地获得数据全貌
数据导入之后,我们可以很快地计算出一些KEGG疾病数据库的基本信息。以下三个命令可以算出三种类型节点的总数:
MATCH (p:pathogen) RETURN COUNT(DISTINCT p)
#returns 333
MATCH (dr:drug) RETURN COUNT(DISTINCT dr)
#returns 1339
MATCH (di:disease) RETURN COUNT(DISTINCT di)
#returns 2498查询结果显示,图数据库里一共有333个“致病生物”,1339个“药物”和2498个“疾病”节点。这个数据库并不大,因此我们可以使用Neo4j Bloom来制作一个数据库的拓扑全貌,在Bloom里输入以下命令并运行(我之前的文章有说明更详细步骤):
MATCH p=(n:disease) <-[]-() RETURN p;
图2: 本项目的拓扑概览。绿色的点代表“疾病”,红色的是“药物”而蓝色的是“致病生物”。作者作图。
以下查询命令可以显示Top 10疾病类型。要注意的是,一些疾病例如Schwartz-Jampel综合征和中央核肌肉病变被归纳到两个以上的疾病类型,但以下的查询命令不会对它们进行重复计数:
MATCH (d:disease) RETURN d.disease_category, COUNT(d.disease_category) as count ORDER BY count DESC LIMIT 10;结果如下:
| d.disease_category | count |
| Congenital malformation | 647 |
| Infectious disease | 345 |
| Nervous system disease | 234 |
| Inherited metabolic disease | 124 |
| Cancer | 97 |
| Congenital disorder of metabolism | 94 |
| Hematologic disease | 63 |
| Immune system disease | 56 |
| Cardiovascular disease | 54 |
| Nervous system disease; Musculoskeletal disease | 52 |
让我惊奇的是,感染疾病以及两大杀手-心血管疾病和癌症都没有登上榜首。最多的疾病类型竟然是先天性畸形(Congenital malformation),例如指甲-髌骨综合征(nail-patella syndrome)和Meckel综合征。
病毒,细菌和真核生物都可引起感染。以下的命令可以显示每种感染源的数量:
MATCH (v:pathogen) RETURN split(v.taxonomy, "; ")[0] as domain, COUNT(split(v.taxonomy, "; ")[0]) as count ORDER BY count DESC;结果如下:
| domain | count |
| Bacteria | 147 |
| Viruses | 132 |
| Eukaryota | 54 |
又一次的惊喜,细菌,而不是病毒,是最频繁的致病生物。我们还可以进一步看看病毒的情况:
MATCH (v:pathogen) WHERE v.taxonomy contains "virus" RETURN split(v.taxonomy, "; ")[0] + "; " + split(v.taxonomy, "; ")[1] as domain, COUNT(split(v.taxonomy, "; ")[0] + "; " + split(v.taxonomy, "; ")[1]) as count ORDER BY count DESC;该查询命令取回第一和第二层生物分类名字以及它们引起的疾病的总数:
| domain | count |
| Viruses; Riboviria | 104 |
| Viruses; Duplodnaviria | 9 |
| Viruses; Varidnaviria | 9 |
| Viruses; Monodnaviria | 9 |
| Viruses; Deltavirus | 1 |
如结果显示,核糖病毒域是最频繁的域。但核糖病毒是一个很大的类别。引发2002-2004 SARS大爆发的SARS冠状病毒和引发本次疫情的SARS-CoV-2病毒都属于核糖病毒。另外,引发艾滋病的人类免疫缺陷病毒(HIV)和肝炎病毒(hepatitis viruses)也属于该域。
最后,我们可以看看治疗感染的药物的数量:
MATCH (dr:drug) -[]->(di:disease) WHERE di.disease_category = "Infectious disease" RETURN COUNT(DISTINCT(dr));该查询返回293件药物。注意该查询一定要加“DISTINCT”,因为一些药物能够用于治疗几种疾病,“DISTINCT”保证它们不会被重复计算。
3. 寻找多用途药物
多用途药物就是能够治疗多种疾病的药物。一药多用。因为药物研发的花费巨大,测试时长,所以如果“旧药能医新病”的话(在这里观看NIH的Francis Collins关于此话题的TED talk),对病人和药商无疑都是件大好事。实际上,药物是受到例如美国的FDA的严格管理的。药商只能以FDA批准的用途营销药物。那究竟哪些药物是用途最多的呢,它们又有什么用途呢?
以下的查询命令可以返回Top 10最多用途的药物:
MATCH (dr:drug) -[r:treats]->(di:disease) RETURN dr.name as drug, COUNT(r) as count ORDER BY count DESC LIMIT 10;结果如下:
| drug | count |
| Prednisolone sodium phosphate | 37 |
| Prednisone | 32 |
| Dexamethasone | 26 |
| Triamcinolone acetonide | 26 |
| Doxycycline hyclate | 26 |
| Doxycycline | 25 |
| Doxycycline calcium | 25 |
| Hydrocortisone | 24 |
| Dexamethasone sodium phosphate | 24 |
| Methylprednisolone | 24 |
这前十名主要就是两类药物:甾体激素和四环素里的多西环素。甾体激素用于压制免疫系统和治疗一系列的炎症。列表里的第三地塞米松(Dexamethasone),曾用于治疗新冠病人。初步结果显示,它能够减少那些有创机械通气和输氧病人的死亡率,但对于那些无需辅助呼吸的病人则没差别(The RECOVERY Collaborative Group, 2020)。多西环素是一种广谱抗生素,被用于治疗疟疾,莱姆病,霍乱和梅毒。实际上,以下的查询命令显示,列表里的这些甾体激素的用途重叠很多;而多西环素及其衍生药物也是用于多种同样疾病。
MATCH (a:drug)-[r:treats]->(b:disease)
WITH a, COUNT(r) AS indicationCount
ORDER BY indicationCount DESC LIMIT 10
MATCH (a:drug)-[r:treats]->(b:disease)
RETURN (a) -[r] -> (b)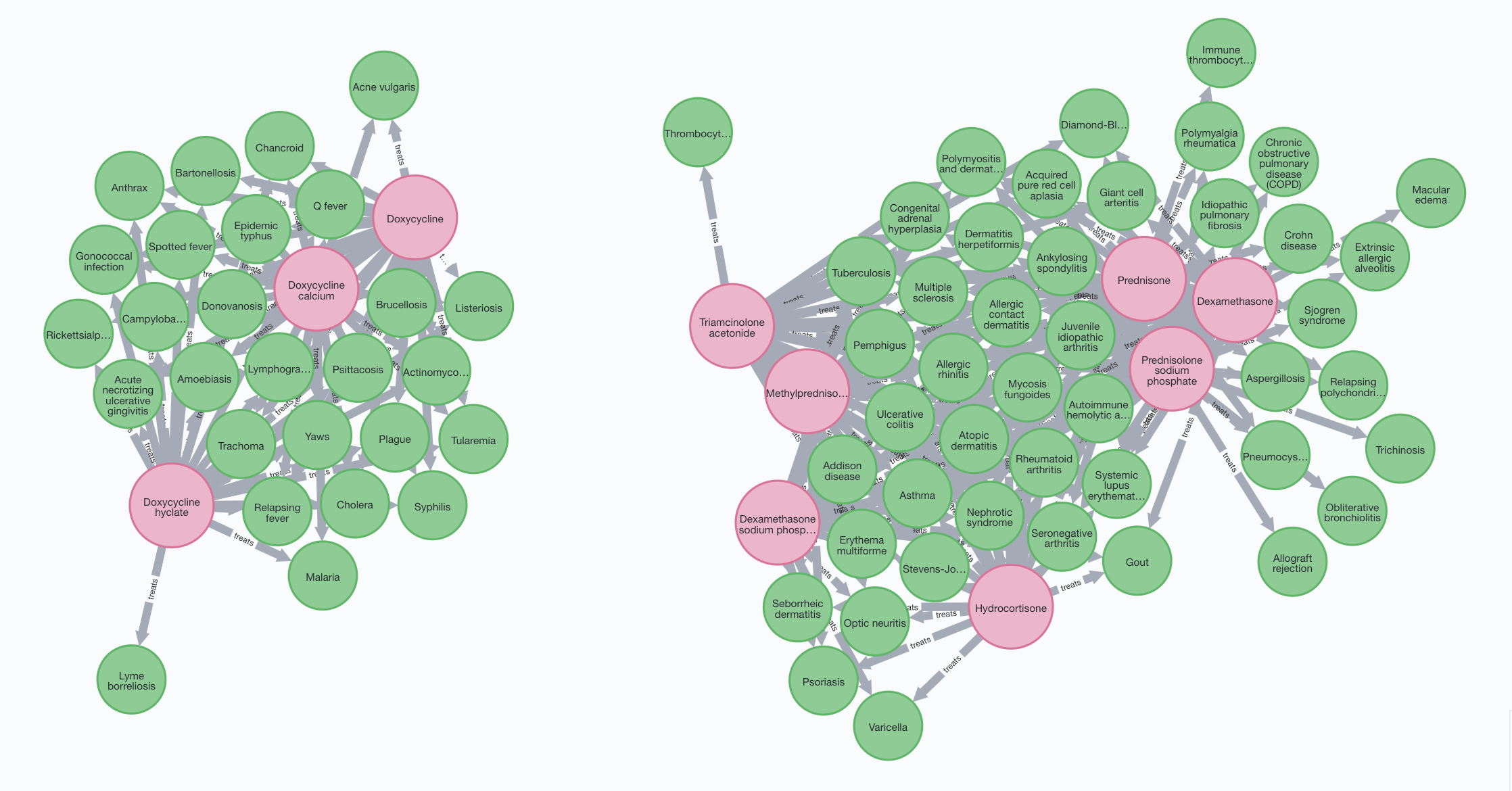
图3,前十多用途药物及其用途。作者作图。
例如,前两名的药物泼尼松(prednisone)和其活性形式泼尼松龙(prednisolone)自1955年起已经在美国被批准药用。它们也非常常见:在美国,泼尼松是第21,泼尼松龙是第134被开得最多的药物(DrugStats Database)。以下的两条命令可以显示泼尼松龙治疗的疾病类型:
MATCH (dr:drug) -[]->(di:disease) WHERE dr.name="Prednisolone sodium phosphate" RETURN di.disease_category, count(di.disease_category) as count ORDER BY count DESC LIMIT 10;
MATCH (dr:drug) -[]->(di:disease) WHERE dr.name="Prednisolone sodium phosphate" AND (di.disease_category = "Lung disease" or di.disease_category="Infectious disease") RETURN di.name, di.disease_category ORDER BY di.disease_category;结果是:
| di.disease_category | count |
| Immune system disease | 10 |
| Infectious disease | 4 |
| Musculoskeletal disease | 3 |
| Skin and connective tissue disease | 2 |
| Hematologic disease; Immune system disease | 2 |
| Lung disease | 2 |
| Skin and connective tissue disease; Immune system disease | 1 |
| Urinary system disease | 1 |
| Endocrine and metabolic diseases | 1 |
| Respiratory disease | 1 |
还有:
| di.name | di.disease_category |
| Trichinosis | Infectious disease |
| Pneumocystis pneumonia | Infectious disease |
| Tuberculosis | Infectious disease |
| Aspergillosis | Infectious disease |
| Chronic obstructive pulmonary disease (COPD) | Lung disease |
| Idiopathic pulmonary fibrosis | Lung disease |
结果显示,泼尼松龙不但用于免疫系统疾病,也用于治疗感染,例如旋毛虫病和结核病。因为它能消炎,它还用于治疗慢性阻塞性肺疾病(COPD)。但对特发性肺纤维化(IPF),其效果尚不明朗。
至于2021年到目前为止在美国开得最多的药物,阿托伐他汀和它的衍生物,只是用于四种代谢和心血管疾病:高脂血症,家族性高胆固醇血症,心绞痛和心肌梗死。

图4,阿托伐他汀和它的衍生物的药用,作者作图。
4. 一些致病生物的细节
现在看看致病生物的情况。首先看看一些Top致病生物的致病的数目。
MATCH (p:pathogen) -[r:causes]->(di:disease) RETURN p.name, COUNT(r) as count ORDER BY count DESC LIMIT 10; | p.name | count |
| Human papillomavirus type 16 | 7 |
| Human papillomavirus type 18 | 5 |
| Human herpesvirus 4 (Epstein-Barr virus) | 4 |
| Human immunodeficiency virus 1 (HIV-1) | 4 |
| Machupo mammarenavirus | 3 |
| Hepatitis B virus (HBV) | 3 |
| Sabia mammarenavirus | 3 |
| Guanarito mammarenavirus | 3 |
| Oropouche virus | 3 |
| Hepatitis C virus (HCV) | 3 |
能致最多疾病的前十全是病毒。前两名人类乳头瘤病毒(HPV)16和18型可以引发多种癌症。根据维基百科全书,大多HPV的感染都是无症状的,而且90%的病例会在两年内褪去。但在其他情况,它们则会残留在体内并引起疣和病变,并增加例如宫颈,外阴,阴道,阴茎,肛门,口部,扁桃体和喉咙等人体腔道表面各种癌变的几率(图5)。几乎所有的宫颈癌都是由HPV引起的;而这前两个病毒型则是占据了70%的病例。而90%的HPV阳性口咽癌就是由16型引起的。

图5,HPV 18和16型和癌症的关系。作者作图。
相比之下,SARS和SARS-CoV-2则各只与一种疾病相关联。SARS引发了2002-2004年中国的SARS疫情,至今仍没有相对的抗病毒治疗。KEGG把新冠肺炎与瑞德西韦(remdesivir)联系起来。根据Siemieniuk等人的持续监察,瑞德西韦可能可以缩短新冠的病情时长。

图6,SARS和SARS-CoV-2及其引起的疾病。作者作图。
而根据KEGG,用于治疗疟疾的硫酸羟氯喹不是新冠治疗药物。

图7,硫酸羟氯喹及治疗的疾病,作者作图。
这也对应了Siemieniuk的一个研究结论:
羟氯喹可能并不能减少死亡率和机械通气,对新冠肺炎也很可能不会带来其他的好处。
5. 使用图算法去发现群和连接性高的节点
图2的拓扑图有些奇特之处。很多小的网络围绕着一个大的网络。如果能够分离出那些群,并且得出那些枢纽,就是连接度高的节点,就好了。幸好,Neo4j提供了很强大的插件:图数据科学(Graph Data Science (gds))来处理这些任务。
首先去Neo4j Desktop里的该项目Plugins项,安装该插件:

图8,安装图数据科学插件,作者作图。
在Neo4j里,图算法要运行在图投射(graph projections)上。所以,首先要用Cypher建一个包括所有节点和联系的图投射“disease-graph”:
CALL gds.graph.create.cypher(
'disease-graph',
'MATCH (n) RETURN id(n) AS id',
'MATCH (n)--(m) RETURN id(n) AS source, id(m) AS target'
)5.1 用Louvain算法构建疾病群
现在我们可以用Louvain算法来构建群。Louvain算法浓缩紧密关联的节点成大的节点,不断重复,直至再没有浓缩可做。首先,看看我们能构建多少群。
CALL gds.louvain.stats('disease-graph')
YIELD communityCount
#return 2120从4170个节点,Louvain算法浓缩出了2120个群。我试着用不同的“maxIterations”和“tolerance”去运行该命令,而结果都是大同小异。
我们可以看看最大的群和里面的成员:
CALL gds.louvain.stream('disease-graph')
YIELD nodeId, communityId, intermediateCommunityIds
RETURN communityId, COUNT(communityId) as count
ORDER BY count DESC LIMIT 10;结果是:
| communityId | count |
| 3421 | 190 |
| 2767 | 189 |
| 2615 | 172 |
| 3549 | 160 |
| 3402 | 120 |
| 3561 | 116 |
| 3518 | 108 |
| 3190 | 90 |
| 2848 | 74 |
| 3239 | 73 |
群3421是最大的,让我们看看里面的成员:
CALL gds.louvain.stream('disease-graph')
YIELD nodeId, communityId, intermediateCommunityIds
WHERE communityId = 3421
RETURN gds.util.asNode(nodeId).name AS name, communityId其前十的成员是:
| name | communityId |
| Osteosarcoma | 3421 |
| Breast cancer | 3421 |
| Melanoma | 3421 |
| Hodgkin lymphoma | 3421 |
| Congenital nongoitrous hypothyroidism (CHNG) | 3421 |
| Basal cell carcinoma | 3421 |
| Cervical cancer | 3421 |
| Viral hepatitis | 3421 |
| Subependymal giant cell astrocytoma | 3421 |
| Head and neck cancer | 3421 |
细细地看过群的成员之后,可以知道该群主要是肿瘤。肝炎病毒如乙肝和丁肝也在该群里面。
我们可以进一步看看其他群。我发现大的群都是被研究得很多的疾病,它们则是进一步通过共同的致病生物或多用药物联系起来形成群。相比之下,很多单独的小岛则是罕见疾病,例如Christianson综合征和Zimmermann-Laband综合征。值得注意的是,虽然SARS和新冠引发了大新闻,它们在KEGG的疾病数据里也只是两个独立的小岛(图2)。说明了:我们相对地还是对新冠知道得很少。
5.2 用PageRank去发现连接度高的节点
最后,我们用PageRank去看看图里哪些节点是连接度高的:
CALL gds.pageRank.stream('disease-graph')
YIELD nodeId, score
RETURN gds.util.asNode(nodeId).name AS name, score
ORDER BY score DESC, name ASC LIMIT 12;结果是:
| name | score |
| High blood pressure | 39.84163054917008 |
| Type 2 diabetes mellitus | 25.417368324007835 |
| HIV infection | 23.399820479750638 |
| Breast cancer | 13.46088549327105 |
| Major depressive disorder | 12.292233879491686 |
| Primary open angle glaucoma | 10.7658223032151 |
| Schizophrenia | 10.442885867986478 |
| Rheumatoid arthritis | 9.220130287948999 |
| Chronic obstructive pulmonary disease (COPD) | 9.169124054256827 |
| Non-small cell lung cancer | 8.981185495341197 |
| Asthma | 8.857213483797388 |
| Psoriasis | 8.154248254094272 |
没有一个“药物”和“致病生物”节点能打进前12,表里全是常见病。原因一方面是,每一个“药物”和“致病生物”都会连接在至少一个“疾病”节点上。所以“疾病”本来就是处在该图项目的中心位置。另一方面,列表里全是现代人类社会里最常见的疾患,而我们也开发了很多药物去治疗它们。我们还可以用另一个查询来数数治疗这些疾患的药物:
CALL gds.pageRank.stream('disease-graph')
YIELD nodeId, score
WITH COLLECT(DISTINCT(gds.util.asNode(nodeId))) AS di_list, score
ORDER BY score DESC
MATCH (dr:drug) -[r:treats]-> (di:disease)
WHERE di in di_list
RETURN di.name as disease_name, score AS pagerank_score, COUNT(DISTINCT(r)) AS drug LIMIT 12;| disease_name | pagerank_score | drug |
| High blood pressure | 39.84163054917008 | 103 |
| Type 2 diabetes mellitus | 25.417368324007835 | 63 |
| HIV infection | 23.399820479750638 | 53 |
| Breast cancer | 13.46088549327105 | 45 |
| Major depressive disorder | 12.292233879491686 | 36 |
| Primary open angle glaucoma | 10.7658223032151 | 26 |
| Schizophrenia | 10.442885867986478 | 30 |
| Rheumatoid arthritis | 9.220130287948999 | 48 |
| Chronic obstructive pulmonary disease (COPD) | 9.169124054256827 | 31 |
| Non-small cell lung cancer | 8.981185495341197 | 32 |
| Asthma | 8.857213483797388 | 40 |
| Psoriasis | 8.154248254094272 | 34 |
结果显示,103样药物可用于治疗第一项的高血压。西方社会2-4%的人口患有牛皮癣,表里的最后一项,也获得了34样药物的治疗。但是细看最后两列可以发现,药物数量的排名和PageRank的排名并不一致。比如,有48样药物可用于类风湿关节炎,多于排在前面的乳腺癌,重性抑郁疾患和精神分裂症。
结论
该小项目又一次显示了Neo4j在医药生物研究方面的优势。导入数据之后,我们可以在弹指之间获得大量关于疾病的数字和细节。如果是用关系数据库,我们至少得用三个表格做数据模型。而在Neo4j,我们只要一个就可以了。另外,其查询语言Cypher非常直观,没有SQL里频繁的“JOIN”,编写的人可以很直白地写出查询语句,读者也能很快地读懂作者的意图。最后,图数据库还允许我们运行图算法,让我们很快地获得诸如群和高连接度的节点
当然这并不是说我们要舍弃其他数据库。如果我们的数据里具有很多的关联,图数据库是个很好的选择。我们之前的基因组项目和眼下这个疾病数据项目就是好例子。但如果我们有的是简单的表格,例如价目表,我们则应该用关系数据库了。对于文档,我们可以考虑MongoDB。时间系列的数据则有它们自己的数据库,例如Amazon的Timestream和InfluxDB。
这个项目也带给了我们不少关于疾病和健康的新知识。其中让我印象最深的是关于我们的免疫系统。现在疫情,我们很容易会误入那些铺天盖地的“加强你的免疫力”广告。但其实我们的免疫系统是很容易被矫枉过正的,我们也绝不能让它超速过热。相反,我们上面的分析表明,最多用和开得最多的药物,其实是抑制免疫力的药物。而世界上也有很多人在受着免疫力过强之苦。约翰斯·霍普金斯大学估计3%的美国人患有自身免疫疾病,大概一千万人。而患者更多为女性(自身免疫疾病的男女比率为1:2)。这让我想起了Matt Richtel的书《An Elegant Defense》里提到,美国新冠疫情的理性代表人物--免疫学家安东尼·弗契(Anthony Fauci),当他听到提高免疫力的广告的时候,说道:
那让我想笑。首先,它们认定你需要提升免疫力,其实那应该是不要的。就算你成功地提升了你的免疫力,你可能提升了它去做有害的事。
该项目只涉及了KEGG很少的一部分数据:疾病,药物和致病生物。我好奇,如果我们能动用所有KEGG的数据的话,我们会发现一些什么样的新知识呢。所以,我鼓励大家去用图数据库来进一步发掘这个数据宝藏,并告诉我你发现的东西。加油!







 本文介绍了如何利用Neo4j分析KEGG疾病数据库,探讨疾病、药物和致病生物之间的关系。通过预处理数据、构建图数据库,使用Louvain算法识别疾病群和PageRank发现高连接度节点。研究发现,免疫抑制药物用途广泛,而病毒是最常见的致病生物。项目源代码已公开在GitHub上。
本文介绍了如何利用Neo4j分析KEGG疾病数据库,探讨疾病、药物和致病生物之间的关系。通过预处理数据、构建图数据库,使用Louvain算法识别疾病群和PageRank发现高连接度节点。研究发现,免疫抑制药物用途广泛,而病毒是最常见的致病生物。项目源代码已公开在GitHub上。
















 763
763

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








