六、搭建基于知识图谱的农医对话系统
3.命名实体识别数据标注
任务描述
在本任务中,使用doccano工具标注非结构化水稻疾病文本,仅将”水稻病症状“实体词标注出来即可。
知识点:doccano工具的安装、标注数据
重 点:标注数据
难 点:标注数据
内 容:1. 安装doccano工具
2. 标注数据
1. 安装doccano工具
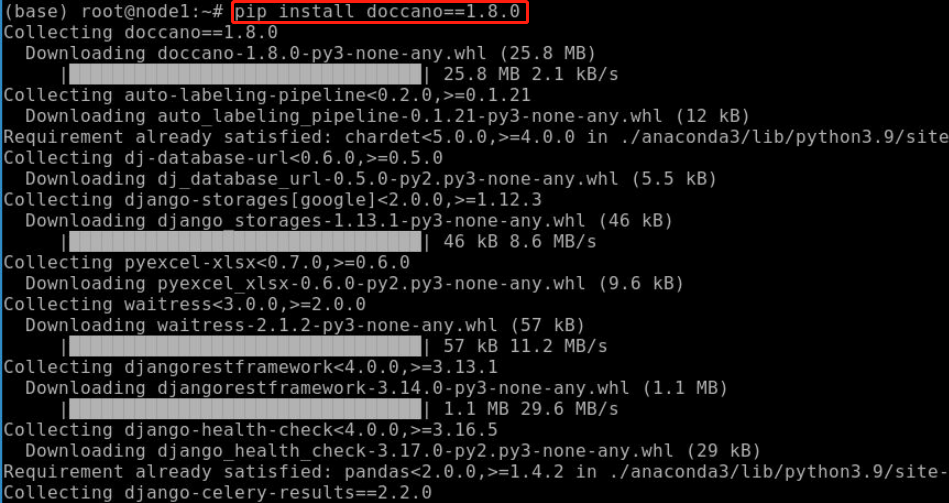
在终端输入以下命令安装doccano:
pip install doccano==1.8.0注:如果安装过程较慢,可以采用清华源进行安装,在上述命令的基础上加“-i https://pypi.tuna.tsinghua.edu.cn/simple”。
安装过程如下图所示:
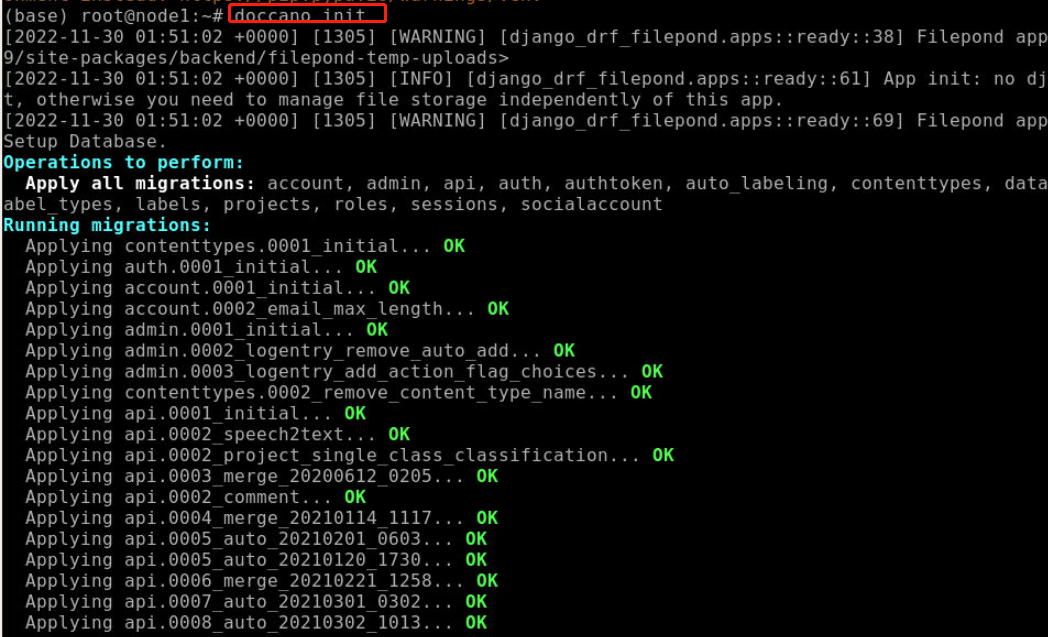
输入以下命令初始化doccano:
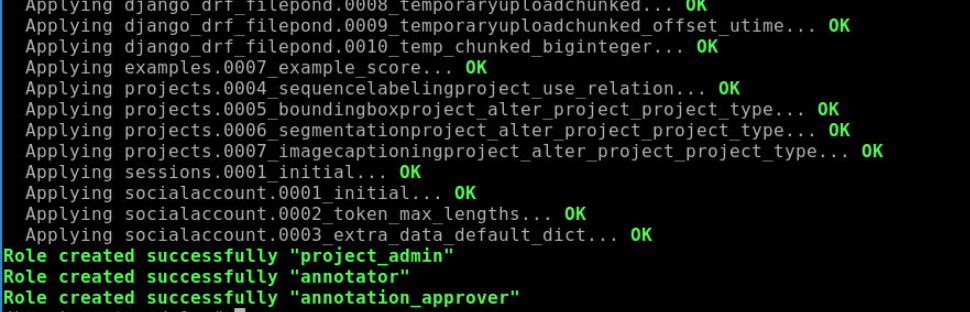
doccano init初始化过程如下图所示:
……
在终端输入以下命令设置用户名和密码:
doccano createuser --username admin --password 1234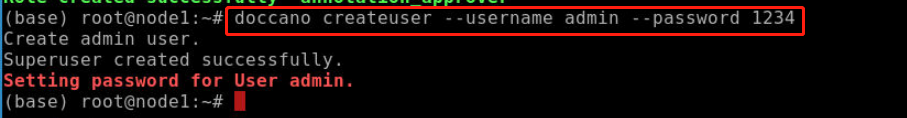
输入以下命令,启动Web服务,端口号不必必须使用8000,可使用其他端口号:
doccano webserver --port 8000启动过程如下图所示:
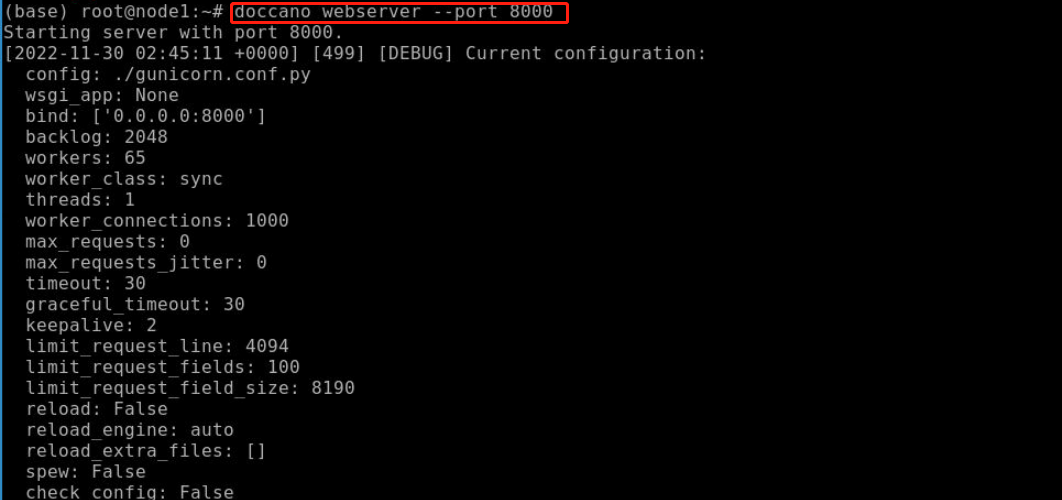
保持上一个终端,再打开一个新的终端,并输入以下命令启动doccano的任务队列:
doccano task命令执行过程如下图所示:

打开浏览器,访问http://127.0.0.1:8000/,进入如下图所示的界面:

点击右上角的“登录”,进入登陆界面:
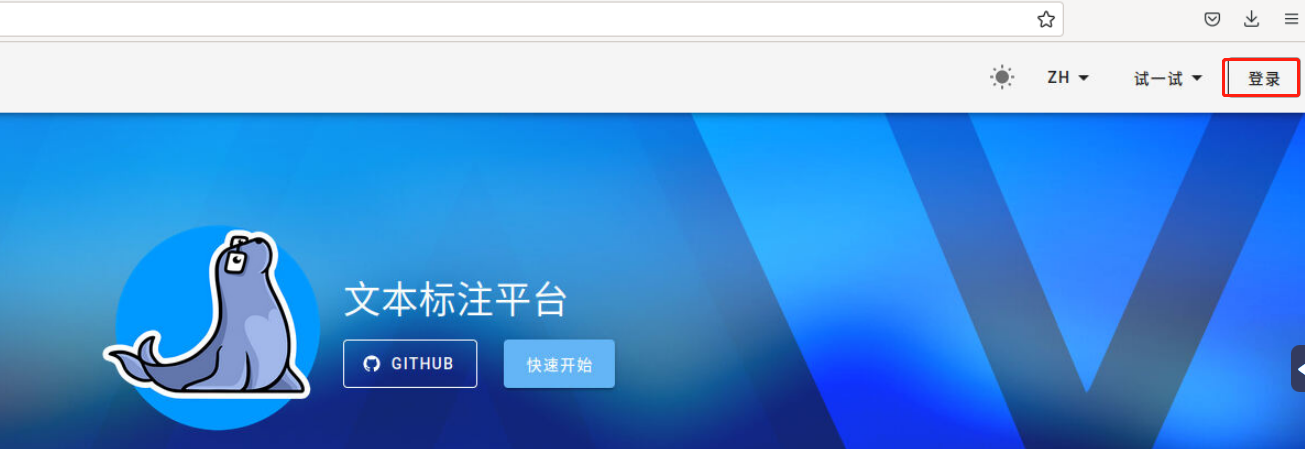
如下图所示,填写用户名和密码,并点击“登录”:
点击“创建”创建标注项目:

进入如下图所示的界面后,选择“序列标注”标注:

选择“序列标注”后,下拉界面,填写“项目名称”和“描述”,并勾选“Allow overlapping entity”及“Use relation Labeling”。完成后,点击“创建”按钮,如下图所示:
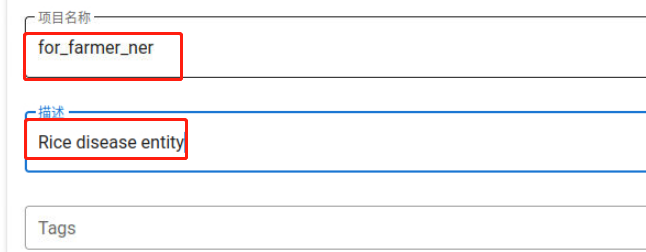
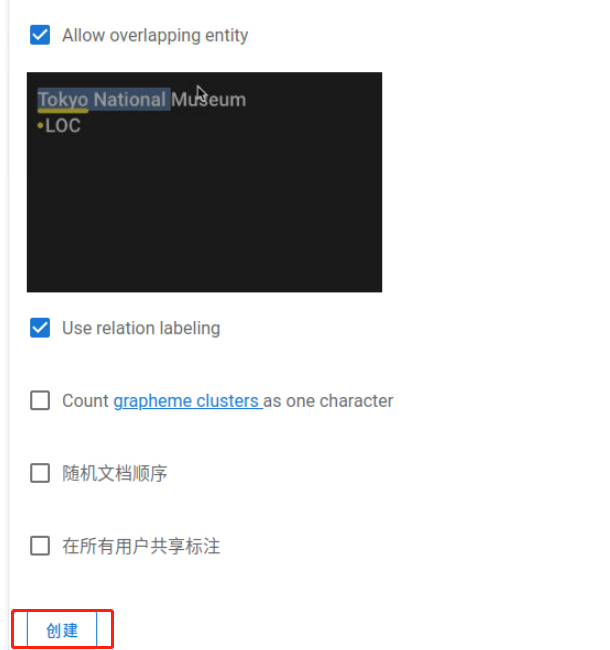
创建完成后,进入如下图所示的页面:
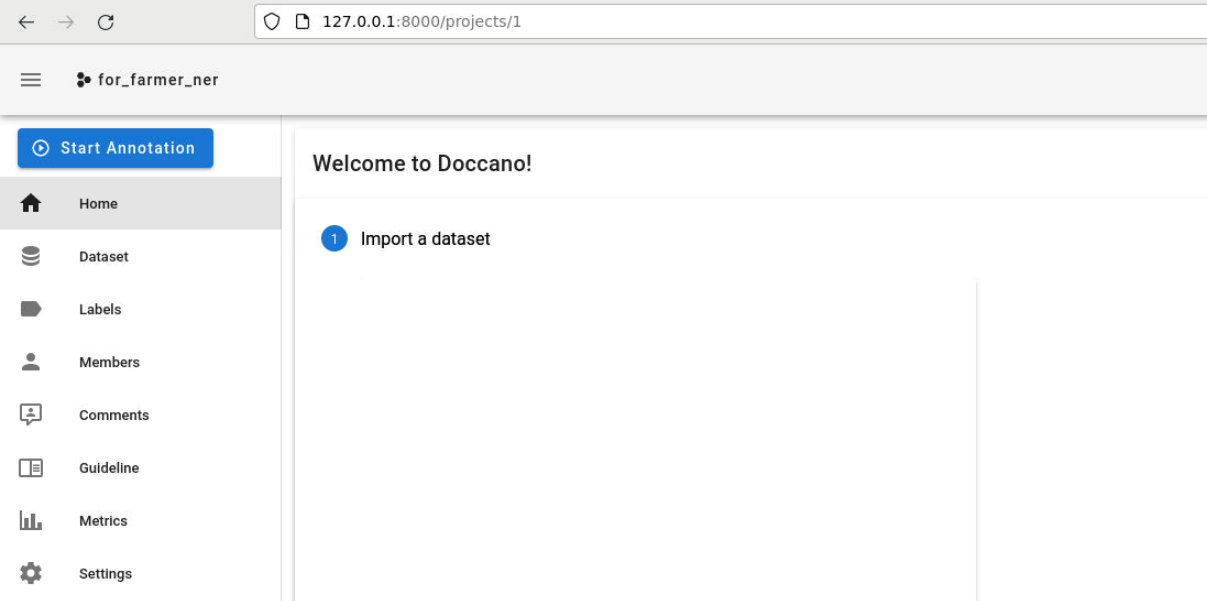
2. 标注数据
按照上述步骤安装好doccano工具,并创建好项目后,便可以开始标注数据。
首先,在"Ai_Farmer_Doctor"目录下新建”local“子目录,在”local“目录下创建子目录”dataset“;接着在”dataset“目录下创建”unstructured_data“子目录。创建好的目录结构如下图所示:
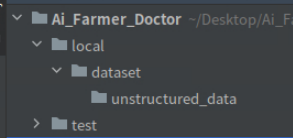
然后,把收集到的水稻疾病数据放到”unstructured_data“目录下,如下图所示:
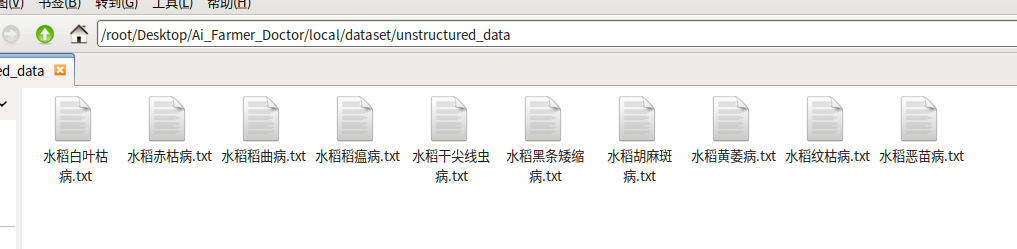
接下来,便可以使用doccano工具开始标注数据。
依次点击“Dataset”->"Actions"->"Import Dataset":
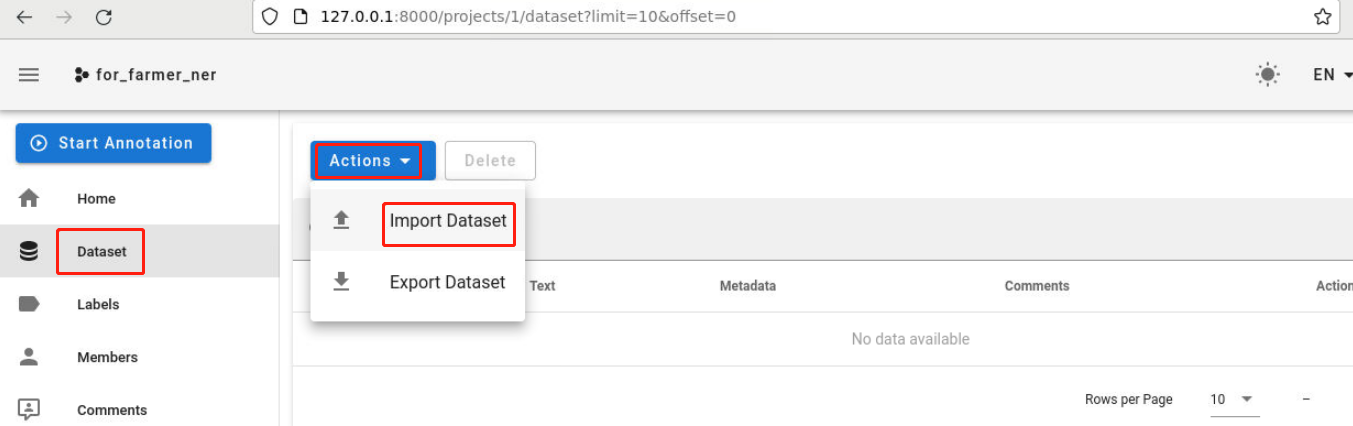
点击“File format”框中的三角符号,选择文本格式:
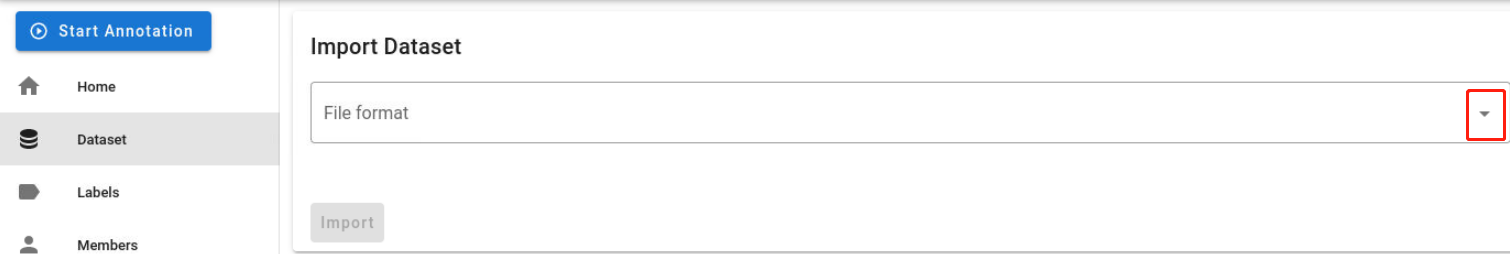
选择“TextFile”格式:
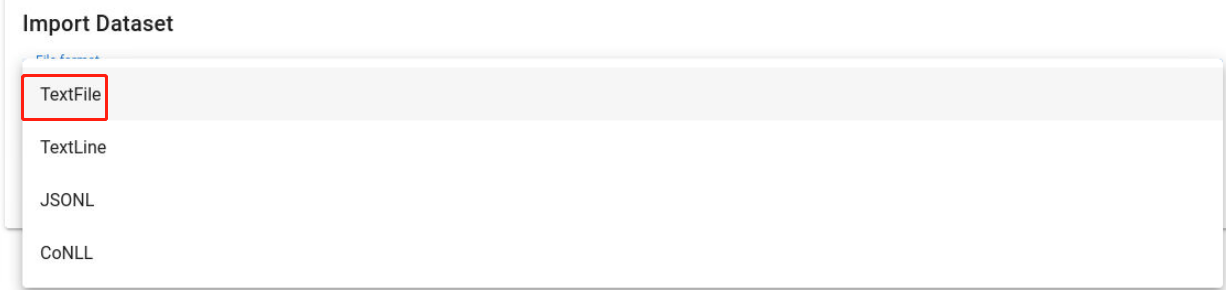
点击“Drop files here…”,导入要进行标注的非结构化水稻疾病数据:
找到水稻疾病数据的存放目录,点击”打开“:
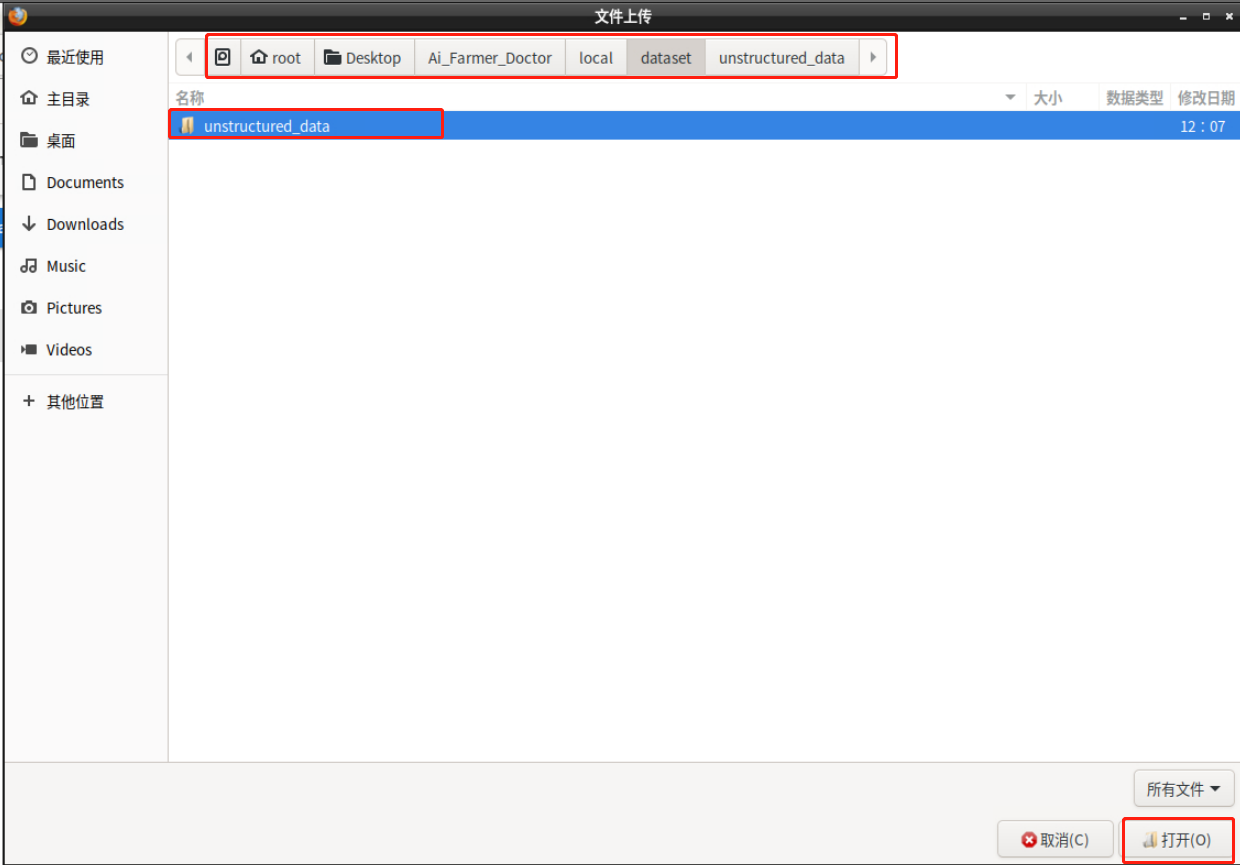
按住”shift“键,选中所有需要的标注的”.txt“文件,并点击”打开“按钮。注意,不需要把所有的数据都进行标注,选取5~8种水稻疾病进行标注即可。
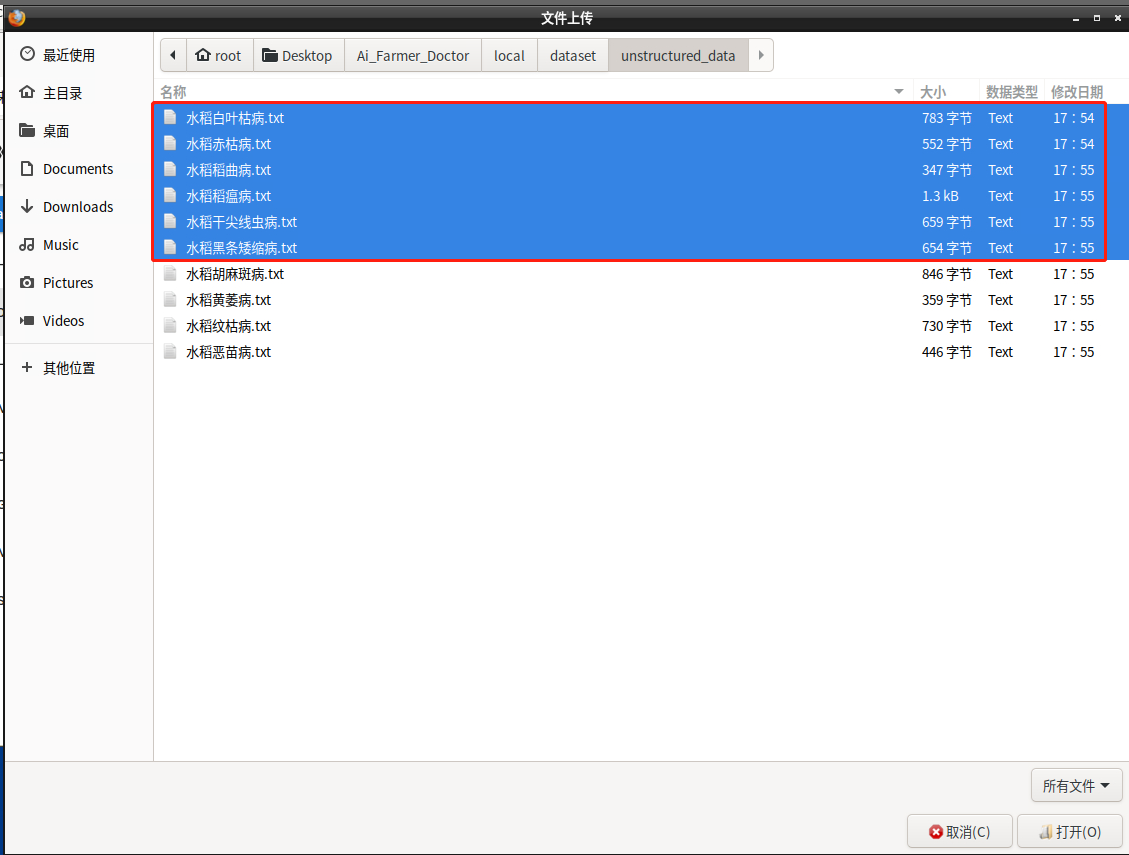
当所有数据的颜色变成绿色时,表明数据已加载成功。点击”Import“按钮导入:
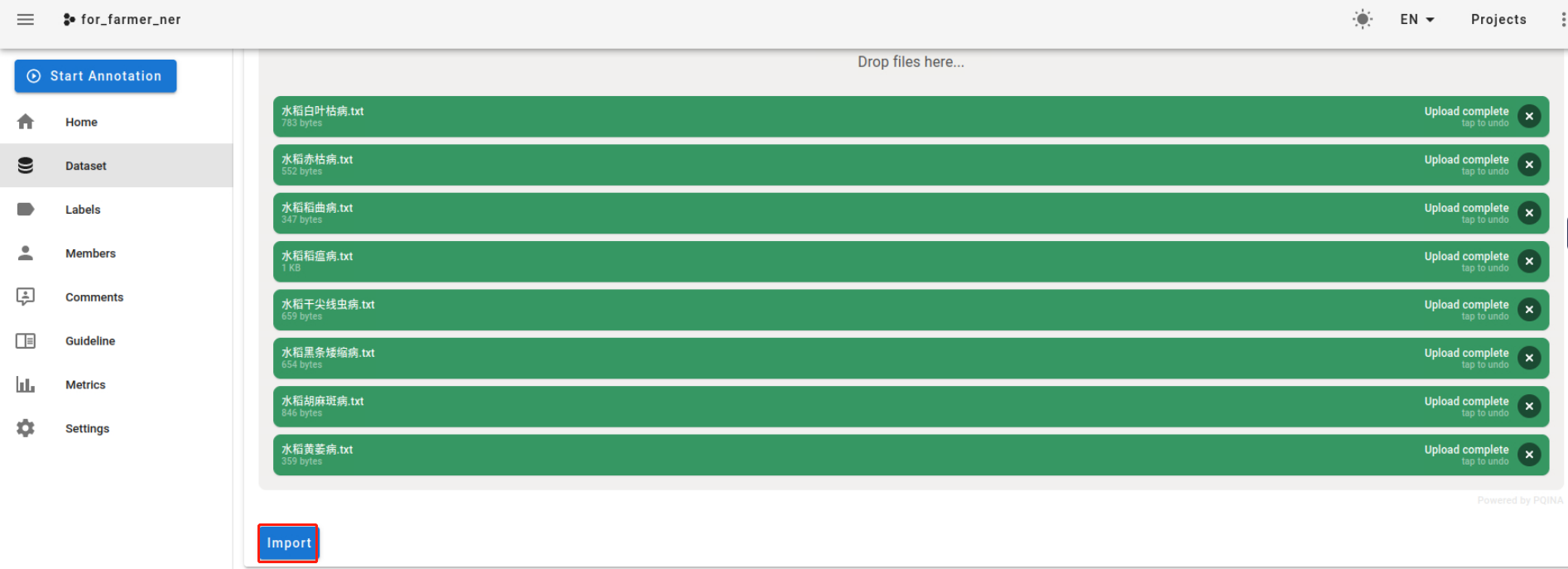
点击”Dataset“,可以看到导入后的数据:

接下来,创建标签。依次点击”Labels“->”Actions“->”Create Label“:
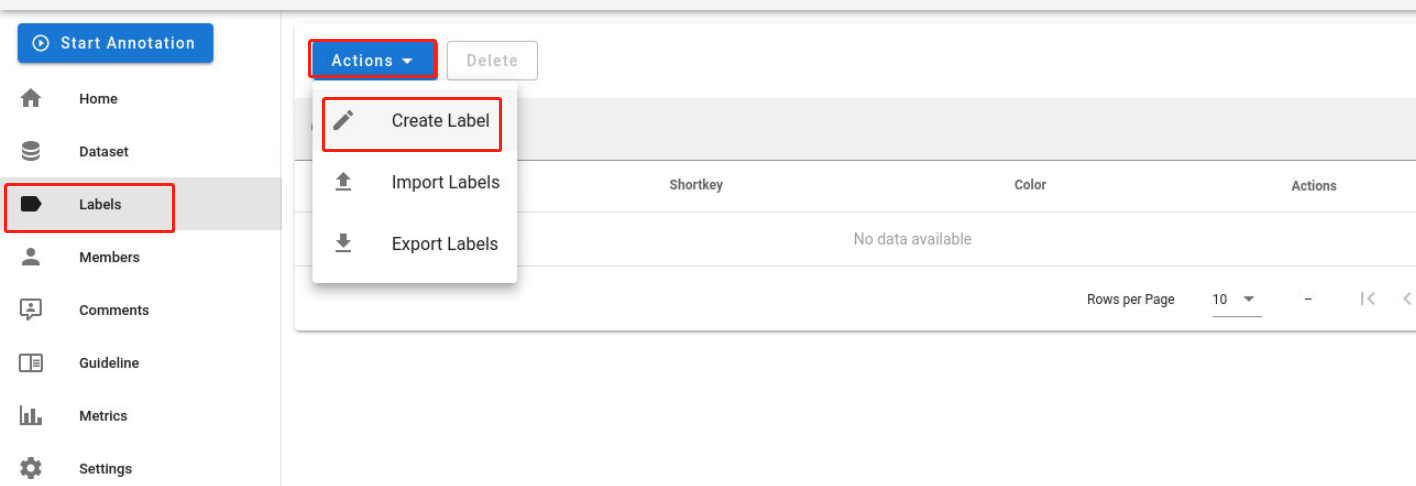
进入以下页面后,填写”Label name“、”Key“、”Color“,并点击”Save“保存.
填写”Label name“时,请切换按住Ctrl+空格键切换谷歌输入法,按Shift键可切换谷歌输入法的中英文输入。
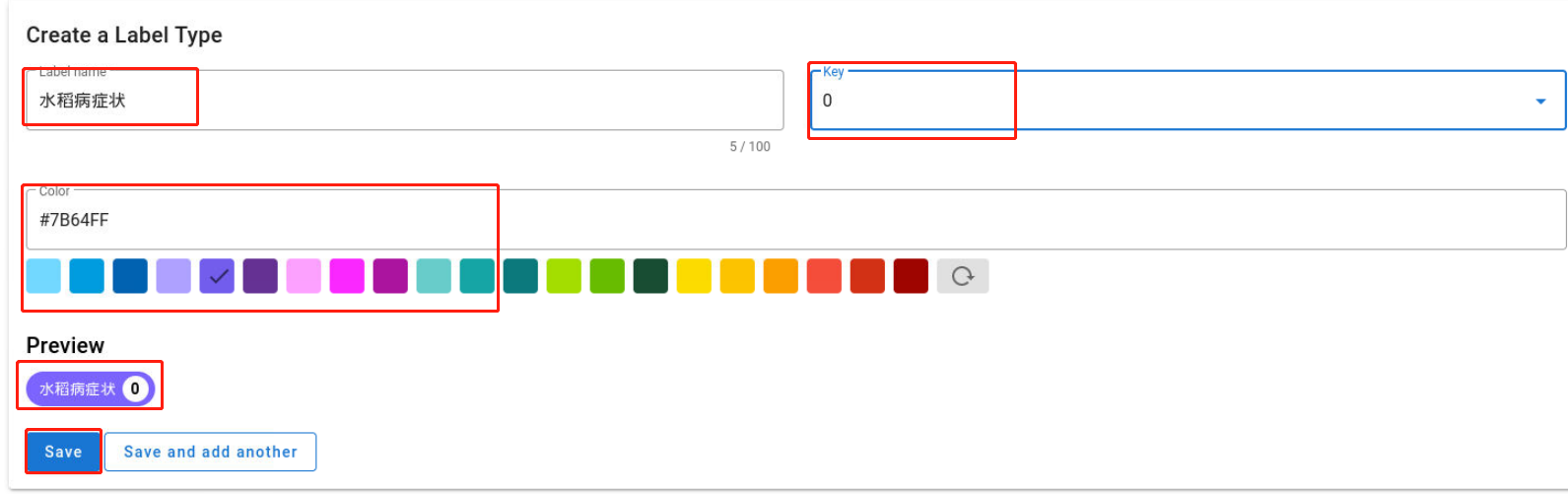
标签信息填写完后,跳转到如下图所示的页面:
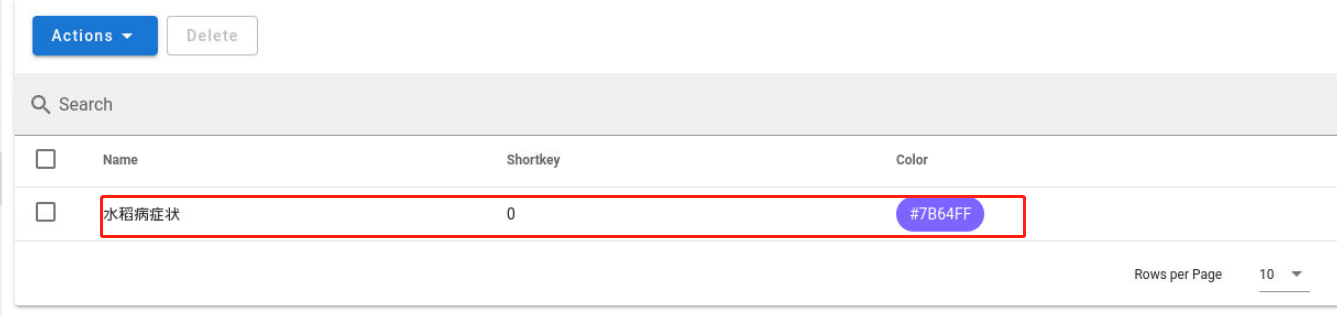
至此,便可以开始标注数据。点击”Dataset“进入数据页面,点击第一条数据对应的”Annotate“:
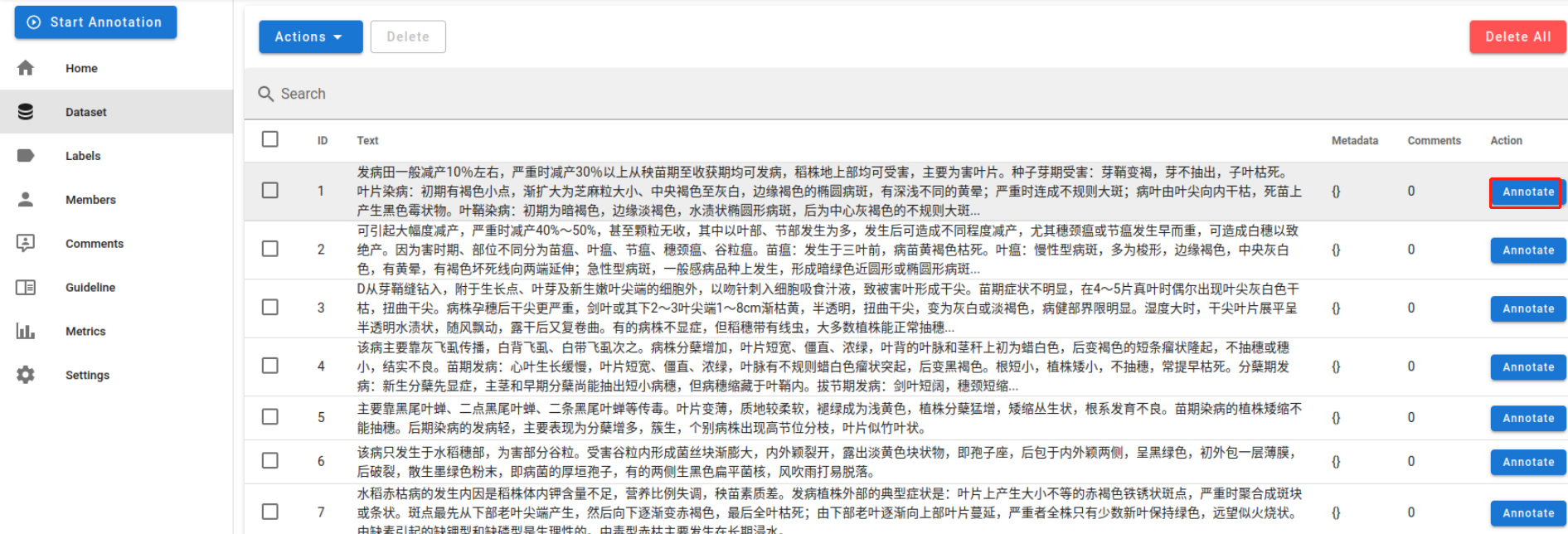
进行命名实体标注时,直接按住鼠标左键选中需要标注的词,并在弹出的”Select a label“对话框中,点击”水稻病症状“标签,即可完成一个实体词的标注。示例:选中”稻苗细长“四个字—>点击”水稻病症状“标签,即可得到如下图所示的结果。
标注完成该条语句的所有”水稻病症状“实体词后,点击左上角的”X“号:

点击左上角的”X“号,变成”√“后,表示一条文本数据的标注完成,同时,右侧的进度条会统计当前数据完成标注的进度:

点击右侧的”>“符号,进行下一条数据的标注。

当完成所有的数据标注后,依次点击”Dataset“—>”Actions“—>”Export Dataset“,以导出标注好的数据文件:
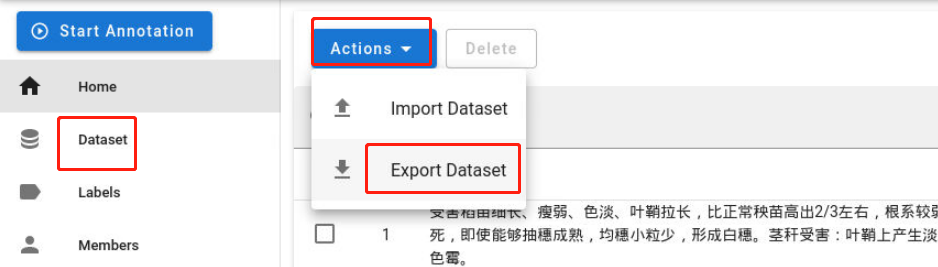
选择数据标注文件的格式为”JSONL“,并点击”Export“按钮:
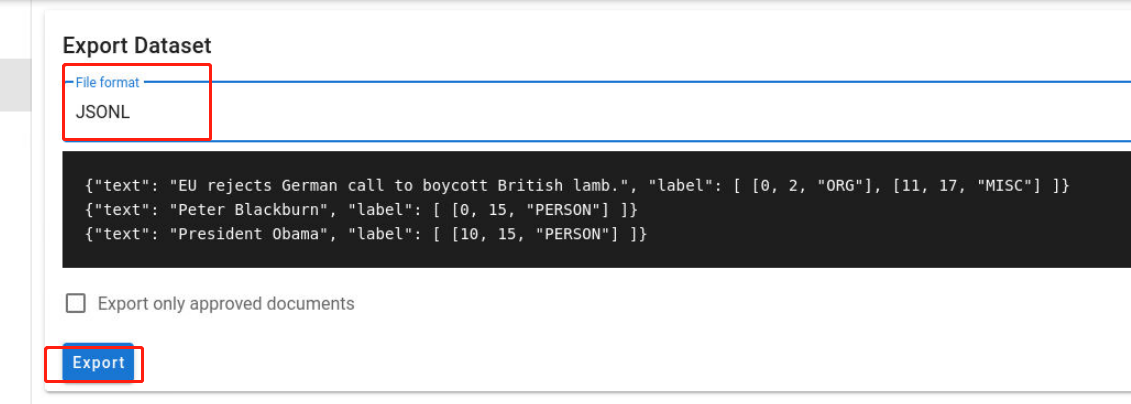
数据会保存在“/root/Downloads/”目录下:
将压缩包解压后,便可得到数据标注文件“admin.jsonl”:
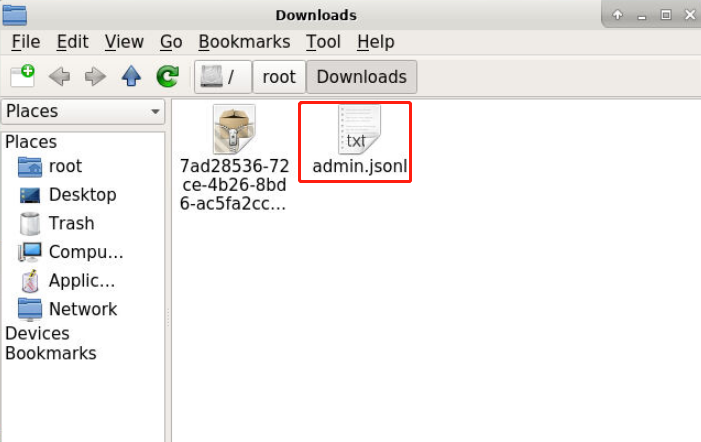
打开“admin.jsonl”文件,内容如下图所示:

- text:对水稻疾病症状描述的非结构化文本数据;
- entities:标注的“水稻病症状”实体词,其中,start_offset为实体词在非结构化文本中的起始位置,end_offset为结束位置。
- relations:为实体词之间的关系,因本任务中并未对实体词之间的关系进行标注,所以该列表中为空。
至此,便完成了数据标注工作,在接下来的任务中,便可使用标注好的数据训练命名实体识别模型。
4.训练命名实体识别模型
任务描述
在本任务中,使用标注好的水稻疾病数据定制训练UIE模型,UIE模型微调较为简单,不需要进行太多的代码改动,只需要更改少量参数即可实现。
知识点:转换并划分数据、微调UIE模型、评估UIE模型、使用UIE模型抽取实体词
重 点:转换并划分数据、微调UIE模型、评估UIE模型、使用UIE模型抽取实体词
难 点:转换并划分数据、微调UIE模型、评估UIE模型、使用UIE模型抽取实体词
内 容:1. 准备工作
2. 转换并划分数据
3. UIE模型微调
4. 模型评估
5. 一键抽取水稻病症状实体词
1. 准备工作
(1)复制代码文件
将uie/目录下的所有代码文件:doccano.py、evaluate.py、finetune.py、model.py、utils.py复制到local/目录下。
(2)新建代码文件
在local/目录下新建一个python文件,并命名为“for_ner_predict”。
(3)新建目录
在local/dataset/目录下新建两个子目录,分别命名为”complete_doccano_data“、“ner_train_data”;
在local/目录下,新建两个子目录,分别命名为“uie_model”、“predict_result”。
(4)复制数据标注文件
将/root/Downlodas/目录下的admin.jsonl文件复制到complete_doccano_data/目录下。
至此,项目目录结构如下图所示:

2. 转换并划分数据
在本模块中,主要对使用doccano工具标注好的数据进行以下两点处理:
(1)将使用doccano工具标注好的数据进行格式转换,转换示例如下:
- 转换前:
{"id":1217,"text":"该病主要靠灰飞虱传播,……","entities":[{"id":864,"label":"水稻病症状","start_offset":5,"end_offset":10},……],"relations":[]}- 转换后:
{"content": "该病主要靠灰飞虱传播,……", "result_list": [{"text": "灰飞虱传播", "start": 5, "end": 10}, ……], "prompt": "水稻病症状"}(2)将转换后的数据按照一定的比例划分成训练集和测试集。
修改doccano.py文件中的代码:
step1:修改配置参数
可修改的配置参数说明如下:
- doccano_file: 从doccano导出的数据标注文件,示例中将admin.jsonl文件放在了dataset/complete_doccanon_data/目录下。
- save_dir: 训练数据的保存目录,默认存储在data目录下。示例中将转换及划分后的结果文件保存在dataset/ner_train_data/目录下。
- negative_ratio: 最大负例比例,该参数只对抽取类型任务有效,适当构造负例可提升模型效果。负例数量和实际的标签数量有关,最大负例数量 = negative_ratio * 正例数量。该参数只对训练集有效,默认为5。为了保证评估指标的准确性,验证集和测试集默认构造全负例。
- splits: 划分数据集时训练集、验证集所占的比例。本项目中,由于数据量较少,因此仅将数据划分为训练集和测试集,比例为[0.8,0,0.2],即训练集与测试集的比例为8:2。
- task_type: 选择任务类型,可选有抽取和分类两种类型的任务。
- options: 指定分类任务的类别标签,该参数只对分类类型任务有效。默认为["正向", "负向"]。
- prompt_prefix: 声明分类任务的prompt前缀信息,该参数只对分类类型任务有效。默认为"情感倾向"。
- is_shuffle: 是否对数据集进行随机打散,默认为True。
- seed: 随机种子,默认为1000.
- separator: 实体类别/评价维度与分类标签的分隔符,该参数只对实体/评价维度级分类任务有效。默认为"##"。
修改完成后,如下图所示:
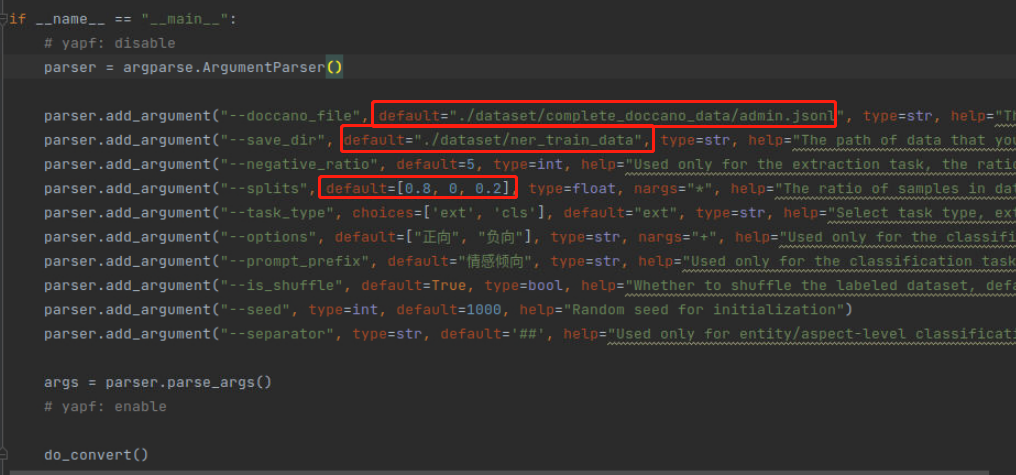
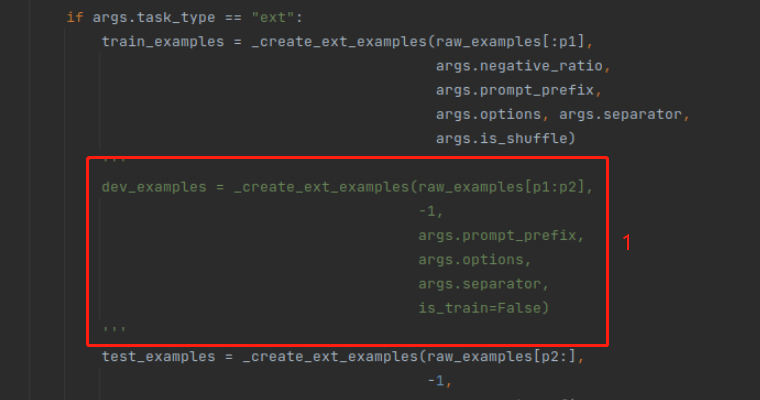
此外,因上述只将数据集划分为了训练集和测试集,因此可将保存验证集的代码注释掉,如下图所示:

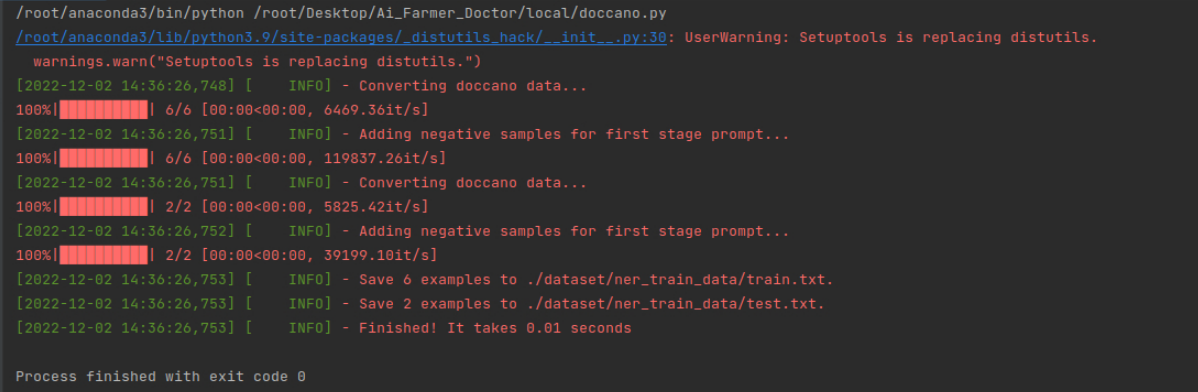
修改完成后,执行doccano.py文件,控制台输出信息如下:
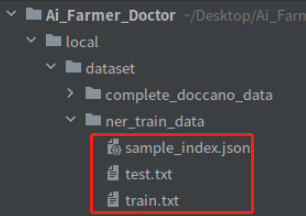
代码执行结束后,会在ner_train_data/目录下,保存数据转换及划分后的结果,如下图所示:
- sample_index.json:保存数据的id,如下图所示:
![]()
- train.txt:训练数据,共6条数据,id为”2,6,5,1,4,7“
- test.txt:测试数据,共2条数据,id为”0,3“。
3. UIE模型微调
本模块,使用标注的少量水稻病症状实体数据微调模型,进一步提升模型效果。
修改finetune.py文件中的代码。可修改配置参数说明如下:
- train_path: 训练集文件路径。
- dev_path: 验证集文件路径。
- save_dir: 模型存储路径,默认为./checkpoint。
- learning_rate: 学习率,默认为1e-5。
- batch_size: 批处理大小,请结合机器情况进行调整,默认为16,本项目的的实验环境需设置为2。
- max_seq_len: 文本最大切分长度,输入超过最大长度时会对输入文本进行自动切分,默认为512。
- num_epochs: 训练轮数,默认为100。
- model: 选择模型,程序会基于选择的模型进行模型微调,可选有uie-base, uie-medium, uie-mini, uie-micro和uie-nano,默认为uie-base。
- seed: 随机种子,默认为1000.
- logging_steps: 日志打印的间隔steps数,默认10。
- valid_steps: evaluate的间隔steps数,默认100。
- device: 选用什么设备进行训练,可选cpu或gpu。
本项目需修改batch_size、train_path、dev_path、save_dir、num_epochs、device六个参数,如下图所示:
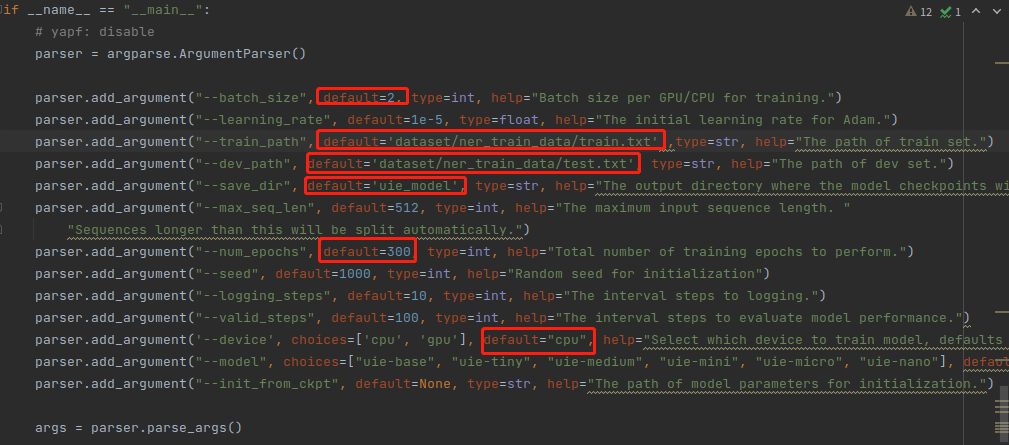
修改完成后,运行finetune.py文件。
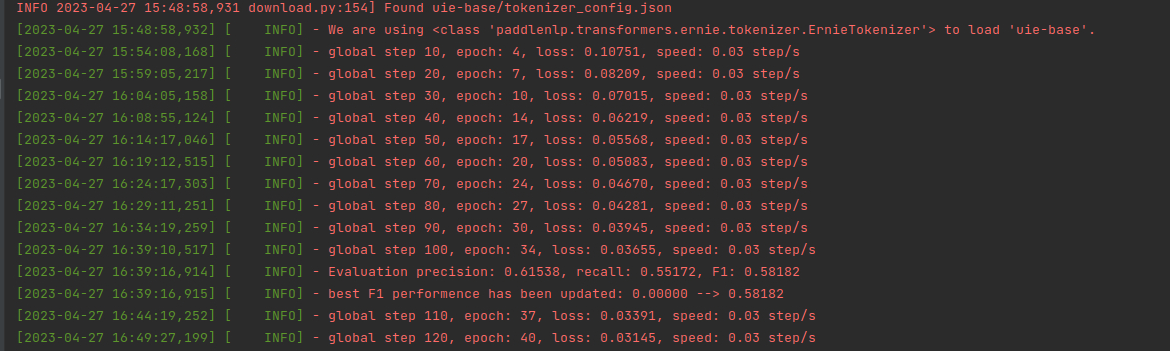
运行过程如下图所示,第一次运行会自动下载“uie-base”预训练模型,保存在uie-base/目录下。
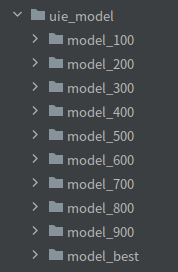
训练过程中,会在uie_model/目录下每隔100个step保存一次模型,且最优的模型会保存在uie_model/model_best目录下,如下图所示:
同学们,可根据训练实际情况调整训练轮次。
4. 模型评估
在本模块中,使用测试集评估训练好的UIE模型的性能。
修改evaluate.py文件中的代码。可配置参数说明如下:
- model_path: 进行评估的模型文件夹路径,路径下需包含模型权重文件model_state.pdparams及配置文件model_config.json。
- test_path: 进行评估的测试集文件。
- batch_size: 批处理大小,请结合机器情况进行调整,默认为16。
- max_seq_len: 文本最大切分长度,输入超过最大长度时会对输入文本进行自动切分,默认为512。
- model: 选择所使用的模型,可选有uie-base, uie-medium, uie-mini, uie-micro和uie-nano,默认为uie-base。
- debug: 是否开启debug模式对每个正例类别分别进行评估,该模式仅用于模型调试,默认关闭。
本项目需修改model_path、test_path两个参数,如下图所示:
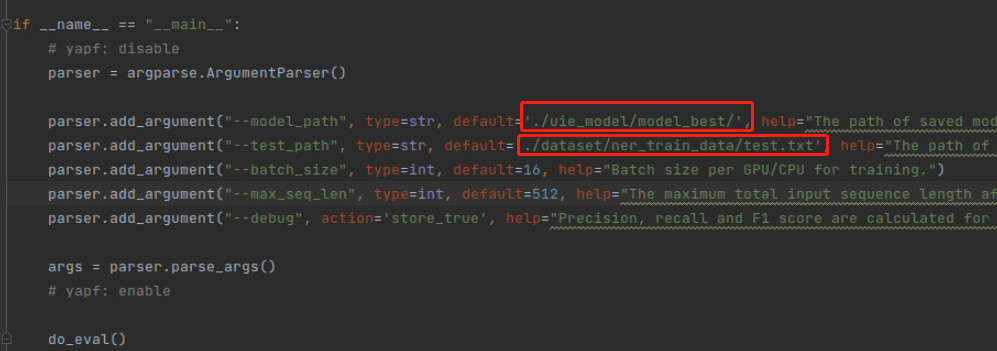
修改完成后,运行evaluate.py文件。运行过程如下图所示:
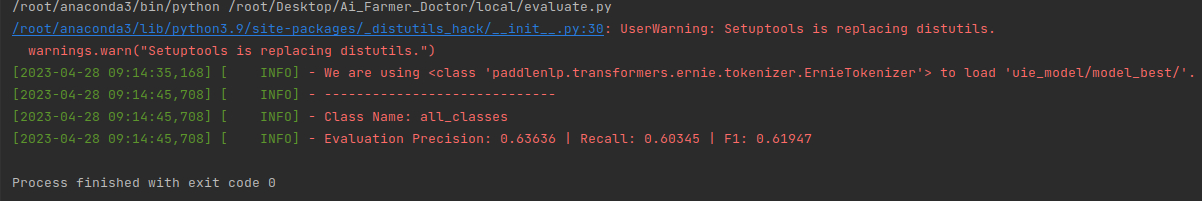
在测试集上的各评价指标得分为“Precision: 0.63636 | Recall: 0.60345 | F1: 0.61947”,各指标的得分并不是很理想,主要有两个原因:(1)用于微调模型以及用于测试的数据量较少;(2)在计算指标时,当抽取的实体词和标注的实体词完全匹配时,认为抽取正确。但实际使用过程中,实体词多一个字和少一个字,并不影响实际的使用。因此,下面需要看一下实际的抽取结果是否满足使用要求。
5. 一键抽取水稻病症状实体词
在本模块中,调用训练好的UIE模型,一键抽取所有水稻疾病的症状实体词。因为后续需要将所有的水稻疾病知识存储到Neo4j图数据库中,以构建知识图谱,因此,需要对所有的数据进行抽取。
在for_ner_predict.py文件中编写本模块的代码。
(1)导入包
from pprint import pprint
from paddlenlp import Taskflow
import os(2)设置全局变量
paddlenlp.Taskflow装载定制模型,通过task_path指定模型权重文件的路径,路径下需要包含训练好的模型权重文件model_state.pdparams。
schema = ['水稻病症状']
anl_model = Taskflow("information_extraction", schema=schema, task_path='./uie_model/model_best') # 训练好的模型
files = os.listdir("./dataset/unstructured_data") # 要进行命名实体识别的数据目录(3)抽取并保存命名实体识别结果
遍历所有待抽取实体词的非结构数据,调用训练好的模型进行命名实体识别,并将抽取的结果按水稻疾病名称进行保存。
for file in files: # 遍历所有的待识别数据文件
with open(os.path.join("./dataset/unstructured_data", file), "r", encoding="UTF-8") as f, open(
os.path.join("./predict_result", file), "w", encoding="UTF-8") as g:
data_1 = f.read() # 读取每一个文件的非结构化数据
data_1 = data_1.replace("\t", "", -1).replace("\n", "", -1) # 去掉空行和空格
#利用模型进行命名实体识别
result = anl_model(data_1) # 进行命名实体识别,抽取水稻病症状实体词
# 保存抽取的实体词
for i in range(len(result[0]['水稻病症状'])):
final_result = result[0]['水稻病症状'][i]['text']
g.write(str(final_result))
g.write("\n")
print("end!!!!")运行for_ner_predict.py文件,运行过程如下图所示:
命名实体识别结果,保存在predict_result/目录下,如下图所示:
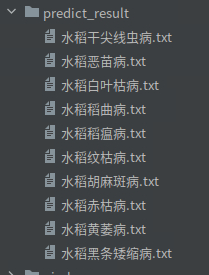
逐个文件打开,对于每一种水稻疾病,人工审核抽取的症状实体词是否正确或者重复,若不正确或存在重复实体词,则删除或手动进行调整。
以“水稻白叶枯病”为例,抽取到的实体词如下图所示:

可以看到,命名实体识别结果是满足使用需求的。







 本文讲述了如何使用doccano进行水稻疾病文本的命名实体识别,包括数据标注、模型微调和评估,以支持农医对话系统的建设。
本文讲述了如何使用doccano进行水稻疾病文本的命名实体识别,包括数据标注、模型微调和评估,以支持农医对话系统的建设。















 722
722

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








