目录
3.实施3种以上的网络模块识别算法, 并使用这些算法来识别重要的蛋白质网络模块
实验目的:
深入了解识别蛋白质-蛋白质相互作用网络关键模块的算法,并从网络中查找重要模块。
实验内容:
1.利用现有的蛋白质相互作用网络,使用三种算法确定重要模块,并计算拓扑特征(网络拓扑属性)。
2.使用软件工具显示蛋白质网络的模块。
3.比较不同算法识别的模块。
实验要求:
1.至少实现三种网络模块识别方法。
2.显示PPI网络的模块。
3.计算PPI网络的拓扑特征。
实验步骤:
1.从多个站点下载小型蛋白质互通网络
HumanNet.txt
2.计算PPI网络的拓扑特征
节点数:1275
边数:16288
平均连通度:25.5498
聚类系数:0.7051792
网络密度:0.02005479
平均路径长度:4.110686
网络直径:9
节点连通度分布:
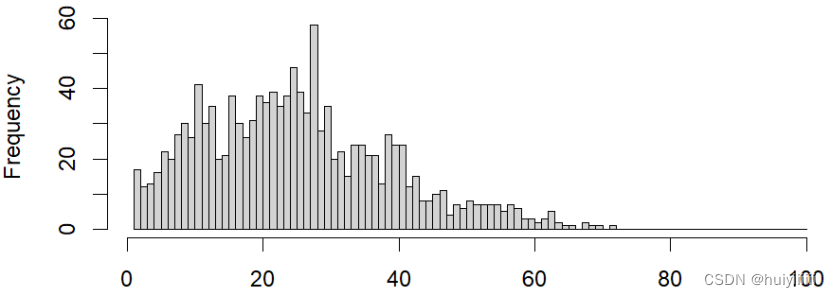
3.实施3种以上的网络模块识别算法, 并使用这些算法来识别重要的蛋白质网络模块
随机步入算法:cluster_walktrap:

贪婪算法cluster_fast_greedy:

GN算法 cluster_edge_betweenness:

4.编写标记为重要的网络可视化程序网络中的功能模块
5.比较基于不同算法的模块,并分析实验结果不同的原因
聚类运行时间分析:
在运行相应的聚类函数时对时间进行记录,结果如下:
cluster_walktrap= 0.4019232 s
cluster_fast_greedy= 0.0344429 s
cluster_edge_betweenness= 288.43704 s
设图的顶点数为n,边数为m:
随机游走算法:时间复杂度O(n^2logn)。随机游走算法计算时间为O(mnH),其中H为相应树状图的高度。最坏的情况是O(mn^2)。但是,大多数现实世界的复杂网络是稀疏的(m=O(n)),并且,H通常很小,并且趋向于树状图平衡的最有利情况(H=O(logn))。在这种情况下,复杂度是O(n^2logn)。
Fast greedy算法:时间复杂度O(n(logn)^2)。在具有n个顶点和m个边的网络上的运行时间为O(mdlogn),其中d是描述群落结构的树状图的深度。许多现实世界的网络都是稀疏和分层的,m=O(n),d=O(logn),在这种情况下,fast greedy算法基本上以线性时间O(n(logn)^2)运行。
GN算法:时间复杂度O(m^2n)。GN算法计算边界数的时间复杂度为O(mn),总时间复杂度在m条边和n个节点的网络下为O(m^2n)。
由于O(m^2n)> O(n^2logn)> O(n(logn)^2),故三种算法最快的是Fast greedy算法,其次是随机游走算法,最慢的是GN算法。
聚类模块数量分析:
随机游走算法:根据参数step的不同,聚类模块数量最多时为80,最小时为17。部分对应关系如下:
| Step | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 |
| Num | 80 | 47 | 33 | 25 | 17 | 20 | 19 | 20 | 19 |
可以看出基本上随机游走算法的step越小,聚类模块的数量越多,但这种关系不是绝对的,例如当step=5时,聚类模块的数量比步数大的还少。
Fast greedy算法:R语言中cluster_fast_greedy算法没有控制聚类模块数量的相关参数,该算法将网络分为11类。
GN算法: R语言中cluster_edge_betweenness算法没有控制聚类模块数量的相关参数,该算法将网络分为23类。
上述三种算法中Fast greedy算法与GN算法只有一种结果,无法控制聚类数量;而随机游走算法可以通过调节参数step的大小进而控制聚类数量,可以通过控制step的大小粗略控制聚类模块数量,但无法准确的控制。
模块度分析:
随机游走:0.8675378 Fast greedy:0.8226374 GN:0.8698903
可以分析出三种聚类算法的模块度均高于0.85,聚类效果很好。其中GN算法的模块度最大,随机游走算法其次,Fast greedy算法最低。
各社区分析:
随机游走算法:
| Id | 顶点数 | 边数 | 聚类系数 | 网络密度 | 平均路径长度 | 网络直径 |
| 1 | 75 | 1088 | 0.7867498 | 0.3920721 | 2.01982 | 6 |
| 2 | 324 | 2507 | 0.5635691 | 0.04791117 | 3.243282 | 8 |
| 3 | 144 | 1887 | 0.6232384 | 0.1832751 | 2.228827 | 5 |
| 4 | 80 | 676 | 0.6131248 | 0.2139241 | 2.317405 | 6 |
| 5 | 58 | 662 | 0.8161668 | 0.400484 | 1.751966 | 4 |
| 6 | 44 | 572 | 0.7999752 | 0.6046512 | 1.491543 | 4 |
| 7 | 129 | 2180 | 0.7638469 | 0.2640504 | 2.074612 | 5 |
| 8 | 27 | 243 | 0.9158919 | 0.6923077 | 1.410256 | 3 |
| 9 | 35 | 318 | 0.8390617 | 0.5344538 | 1.598319 | 4 |
| 10 | 52 | 689 | 0.826532 | 0.5196078 | 1.69457 | 5 |
| 11 | 71 | 1062 | 0.685721 | 0.4273642 | 1.697384 | 5 |
| 12 | 48 | 701 | 0.7892283 | 0.6214539 | 1.425532 | 4 |
| 13 | 28 | 299 | 0.8678269 | 0.7910053 | 1.208995 | 2 |
| 14 | 58 | 1222 | 0.8491207 | 0.7392619 | 1.261948 | 3 |
| 15 | 26 | 325 | 1 | 1 | 1 | 1 |
| 16 | 47 | 757 | 0.7645267 | 0.7002775 | 1.299722 | 2 |
| 17 | 29 | 406 | 1 | 1 | 1 | 1 |


Fast greedy算法:
| 顶点数 | 边数 | 聚类系数 | 网络密度 | 平均路径长度 | 网络直径 | |
| 1 | 232 | 3087 | 0.6668818 | 0.1152038 | 2.8814 | 8 |
| 2 | 337 | 2769 | 0.5928447 | 0.04890844 | 3.284672 | 7 |
| 3 | 107 | 987 | 0.6631464 | 0.1740434 | 2.436255 | 5 |
| 4 | 47 | 730 | 0.7673873 | 0.6753006 | 1.340426 | 3 |
| 5 | 48 | 669 | 0.8033879 | 0.5930851 | 1.484929 | 4 |
| 6 | 31 | 408 | 0.9973615 | 0.8774194 | 1.182796 | 3 |
| 7 | 44 | 544 | 0.7931336 | 0.5750529 | 1.540169 | 4 |
| 8 | 73 | 969 | 0.6801248 | 0.3687215 | 1.92618 | 6 |
| 9 | 87 | 1484 | 0.8457853 | 0.3966854 | 2.046779 | 5 |
| 10 | 84 | 1014 | 0.8426601 | 0.2908778 | 2.244119 | 4 |
| 11 | 185 | 2911 | 0.7632944 | 0.1710341 | 2.526792 | 7 |


GN算法:
| Id | 顶点数 | 边数 | 聚类系数 | 网络密度 | 平均路径长度 | 网络直径 |
| 1 | 66 | 373 | 0.6679178 | 0.1738928 | 2.531935 | 6 |
| 2 | 79 | 1101 | 0.7804871 | 0.3573515 | 2.165531 | 7 |
| 3 | 168 | 1537 | 0.6090893 | 0.1095666 | 2.629099 | 6 |
| 4 | 71 | 1066 | 0.6841199 | 0.4289738 | 1.651911 | 4 |
| 5 | 84 | 1014 | 0.8426601 | 0.2908778 | 2.244119 | 4 |
| 6 | 59 | 713 | 0.8100615 | 0.4167154 | 1.922268 | 6 |
| 7 | 24 | 108 | 0.7319885 | 0.3913043 | 1.826087 | 5 |
| 8 | 76 | 670 | 0.6134454 | 0.2350877 | 2.143158 | 5 |
| 9 | 14 | 53 | 0.6987342 | 0.5824176 | 1.450549 | 3 |
| 10 | 29 | 301 | 0.8619455 | 0.7413793 | 1.268473 | 3 |
| 11 | 90 | 933 | 0.5167477 | 0.2329588 | 2.050936 | 5 |
| 12 | 67 | 937 | 0.7617906 | 0.4237901 | 1.735414 | 5 |
| 13 | 26 | 240 | 0.9169197 | 0.7384615 | 1.316923 | 3 |
| 14 | 9 | 26 | 0.7769784 | 0.7222222 | 1.277778 | 2 |
| 15 | 119 | 2133 | 0.7668013 | 0.3038029 | 1.912833 | 4 |
| 16 | 54 | 730 | 0.7681573 | 0.5101328 | 1.577918 | 4 |
| 17 | 11 | 32 | 0.6705882 | 0.5818182 | 1.418182 | 2 |
| 18 | 59 | 1224 | 0.8485692 | 0.7153711 | 1.300994 | 3 |
| 19 | 35 | 318 | 0.8390617 | 0.5344538 | 1.598319 | 4 |
| 20 | 15 | 51 | 0.7522124 | 0.4857143 | 1.752381 | 5 |
| 21 | 47 | 757 | 0.7645267 | 0.7002775 | 1.299722 | 2 |
| 22 | 44 | 572 | 0.7999752 | 0.6046512 | 1.491543 | 4 |
| 23 | 29 | 406 | 1 | 1 | 1 | 1 |



交叉分析:
随机游走算法与Fast greedy算法:

随机游走算法与GN算法:

Fast greedy算法与GN算法:

在本网络中,三种聚类方法的模块重叠度很高。随机游走算法,Fast greedy算法与GN算法在计算时均以模块度作为聚类算法结束的标准,即三种算法均追求模块度的最大化划分。其中GN算法开始以整个图为一类,计算各个边介数的大小,再不断将最大边介数的边去除来进行聚类。而随机游走算法与Fast greedy算法开始以每个节点为一类,利用层次聚类进行聚类;随机游走算法通过概率转移矩阵定义各类之间的距离,不断将距离最小的两个类别进行合并;Fast greedy算法利用模块度的增量,不断将模块度增量最大的模块进行合并。
参考代码:
rm(list=ls())
library(igraph)
data<- read.csv("HumanNet.txt",header = TRUE,sep ="\t")
g<-graph.data.frame(data,directed = FALSE)
start<-Sys.time()
c1<-cluster_walktrap(g,step=5)
end1<-Sys.time()
runningtime<-end1-start
print(runningtime)
c2<-cluster_fast_greedy(g)
end2<-Sys.time()
runningtime<-end2-end1
print(runningtime)
c3<-cluster_edge_betweenness(g)
end3<-Sys.time()
runningtime<-end3-end2
print(runningtime)
##节点数
V(g)
##边数
E(g)
##计算平均连通度
sum(degree(g))/length(V(g))
##计算聚类系数
transitivity(g)
##计算网络密度
edge_density(g)
##计算平均路径长度
average.path.length(g)
##计算直径
diameter(g)
##度分布
hist(degree(g,mode="all"),breaks=1:100)
##画图
c<-c1
modularity(c)
#plot(c,g)
V(g)$label=NA
V(g)[degree(g)>=600]$label=V(g)[degree(g)>=600]$name
V(g)$size=4
V(g)[degree(g)>=600]$size=15
mem.col<-rainbow(length(unique(c$membership)),alpha=0.5)
V(g)$color<-mem.col[c$membership]
pdf(paste("C:/Users/86136/Documents/R/lab","/1.pdf",sep=""))
plot(g,layout=layout_with_fr,vertex.color=V(g)$color,
vertex.laber=V(g)$label,vertex.size=V(g)$size)
dev.off()
##聚类模块数量
c<-c1
length(table(c$membership))
c<-c3
modularity(c)
##各社区节点与边数量
c<-c3
for (i in 1:length(table(c$membership))){
nodes <- V(g)[c$membership == i]
g1 <- induced_subgraph(g, nodes)
cat(i,'\t',length(V(g1)),'\t',length(E(g1)),'\t',
transitivity(g1),'\t',edge_density(g1),'\t',
average.path.length(g1),'\t',diameter(g1),'\n')
plot(g1, layout = layout_with_fr,main=i)
}
library(pheatmap)
a<-table(c1$membership,c2$membership)
pheatmap(a,display_numbers = TRUE)
b<-table(c1$membership,c3$membership)
pheatmap(b,display_numbers = TRUE)
c<-table(c2$membership,c3$membership)
pheatmap(c,display_numbers = TRUE)
d<-table(c1$membership,c2$membership,c3$membership)
pheatmap(d,display_numbers = TRUE)






 本文详细描述了一项实验,旨在研究如何使用多种算法识别蛋白质-蛋白质相互作用网络的关键模块,包括随机游走、Fastgreedy和GN算法。实验涉及网络拓扑特征分析、模块数量和质量评估,以及不同算法间的性能比较。结果显示Fastgreedy算法运行速度最快,而GN算法具有最高的模块度。
本文详细描述了一项实验,旨在研究如何使用多种算法识别蛋白质-蛋白质相互作用网络的关键模块,包括随机游走、Fastgreedy和GN算法。实验涉及网络拓扑特征分析、模块数量和质量评估,以及不同算法间的性能比较。结果显示Fastgreedy算法运行速度最快,而GN算法具有最高的模块度。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








