一、前言
k-means算法对离群点敏感,因为这种对象远离大多数数据,隐藏分配到一个簇时,它们可能严重地扭曲簇的均值。这不经意间影响了其他对象到簇的分配。
于是一种基于K-means的改进算法k-mediod应运而生。围绕中心点划分(Partitioning Around Medoids, PAM)算法是k-medoids聚类的一种流行的实现。
k-mediod和Kmeans算法核心思想大同小异,但是最大的不同是在中心点的修正,K-means中选取的中心点为当前类中所有点的重心,而K-medoids法选取的中心点为当前cluster中存在的一点k-medoids修正聚类中心的时候,是计算类簇中除开聚类中心的每点到其他所有点的聚类的最小值来优化新的聚类中心.正是这一差别使得k-mediod弥补了k-means算法的缺点.k-mediod对噪声和孤立点不敏感.
但是事情都具有两面性.这种聚类准确性的提高是牺牲聚类时间来实现的.不难看出.k-mediod需要不断的找出每个点到其他所有点的距离的最小值来修正聚类中心,这大大加大了聚类收敛的时间.所以K-mediod对于大规模数据聚类就显得力不从心,只能适应较小规模的数值聚类.
二、算法描述
具体的算法流程如下:
1.在总体n个样本点中任意选取k个点作为medoids
2.按照与medoids最近的原则,将剩余的n-k个点分配到当前最佳的medoids代表的类中(实现了初始的聚类)
3.对于第 i 个类中除对应medoids点外的所有其他点,按顺序计算当其为新的medoids时,准则函数的值,遍历所有可能,选取准则函数最小时对应的点作为新的medoids
4.重复2-3的过程,直到所有的medoids点不再发生变化或已达到设定的最大迭代次数
5.产出最终确定的k个类
准则函数: 最小化绝对误差
E
=
∑
i
=
1
k
∑
p
∈
C
j
d
i
s
t
(
p
,
o
i
)
E=\sum _{i=1}^{k} \sum _{p \in C_{j}}dist(p, o_{i})
E=i=1∑kp∈Cj∑dist(p,oi)
三、实例描述算法
对下列表中(图1)的10个数据聚类, k=2.可以看到这里每个数据的维度都为2。

-
随机挑选k=2个中心点:c1=(3,4) , c2=(7,4).那么将所有点到这两点的距离计算出来(图2),可以看到黑体为到两个中心点距离较小的距离值。那么根据图2,我们可以对所有数据点进行归类:
Cluster1 = {(3,4)(2,6)(3,8)(4,7)}
Cluster2 = {(7,4)(6,2)(6,4)(7,3)(8,5)(7,6)}
很容易算出此时的损失值cost为:20
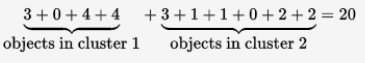
2. 挑选一个非中心点O’,让我们假定挑选的为
X
7
X_{7}
X7 ,即O‘=(7,3)。那么此时这两个中心点暂时变成了c1(3,4) and O′(7,3),那么我们要计算一下这一替换措施所带来的损失cost:
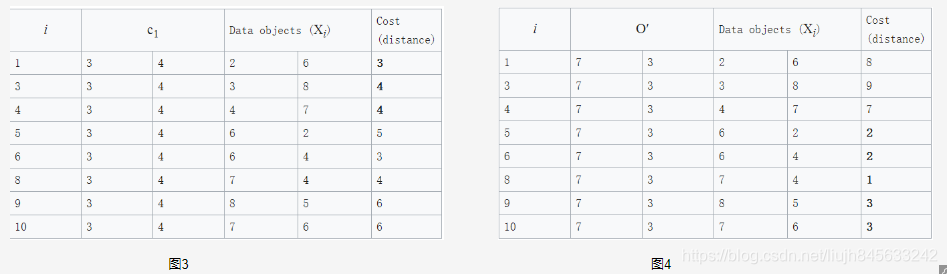
正如图3和图4所见,此时的cost(很好计算,黑体数值的和)变成了: total cost = 3+4+4+2+2+1+3+3 = 22
此时的cost为22,比之前的cost=20要大,所以这次替换的损失变大啦,我们最终不进行这次替换。
这仅仅是尝试 X 7 X_{7} X7 替代c2点,我们应该计算除了c1和c2点外的所有点分别替代c1和c2,将这些替换后的损失都计算出来,看看有没有比20小的损失,如果有那么我们就将这个最小损失对应的中心点对作为新的中心点对。至此才完成了一次迭代。重复迭代直至收敛。
四、python代码
注意:
-
下面代码中用到了scipy.spatial.distance import cdist中的计算各个样本对象距离的函数。这样比自己手写的计算距离的函数,速度快很多。
-
另外,下面代码中用的的 medoids: 中心对象集,是一个字典结构。
medoids =
{
‘0’: 0-cluster 的数据对象索引列表
‘1’: 1-cluster 的数据对象索引列表
…
‘k-1’: 0-cluster 的数据对象索引列表
‘cen_idx’:中心点对象的索引列表
‘t_cost’:总的代价
}
import numpy as np
from scipy.spatial.distance import cdist
import random
import matplotlib.pyplot as plt
import copy
# 两向量的欧式距离
def distEclud(vecA, vecB):
return np.sqrt(np.sum(np.power(vecA-vecB,2)))
# # 计算样本点与k个质心的距离
# def cal_dist(dataMat, centroids, k):
# n = np.shape(dataMat)[0] # 样本点个数,注意这里的dataMat是矩阵
# dist = []
# for i in range(n):
# dist.append([])
# for j in range(k):
# dist[i].append(distEclud(dataMat[i, :], centroids[j]))
# dist_array = np.array(dist)
# return dist_array
def total_cost(dataMat, medoids):
"""
计算总代价
:param dataMat: 数据对象集
:param medoids: 中心对象集,是一个字典,
0: 0-cluster的索引;1: 1-cluster的索引;k: k-cluster的索引;cen_idx:存放中心对象索引;t_cost:存放总的代价
:return:
"""
med_idx = medoids["cen_idx"] #中心对象索引
k = len(med_idx) #中心对象个数
cost = 0
medObject = dataMat[med_idx,:]
dis = cdist(dataMat, medObject, 'euclidean') #计算得到所有样本对象跟每个中心对象的距离
cost = dis.min(axis=1).sum()
medoids["t_cost"] = cost
# 根据距离重新分配数据样本
def Assment(dataMat, mediods):
med_idx = mediods["cen_idx"] #中心点索引
med = dataMat[med_idx] #得到中心点对象
k = len(med_idx) #类簇个数
dist = cdist(dataMat, med, 'euclidean')
idx = dist.argmin(axis=1) #最小距离对应的索引
for i in range(k):
mediods[i] = np.where(idx == i) # 第i个簇的成员的索引
def PAM(data, k):
data = np.mat(data)
N = len(data) #总样本个数
cur_medoids = {}
cur_medoids["cen_idx"] = random.sample(set(range(N)), k) #随机生成k个中心对象的索引
Assment(data, cur_medoids)
total_cost(data, cur_medoids)
old_medoids = {}
old_medoids["cen_idx"] = []
iter_counter = 1
while not set(old_medoids['cen_idx']) == set(cur_medoids['cen_idx']):
print("iteration counter:", iter_counter)
iter_counter = iter_counter + 1
best_medoids = copy.deepcopy(cur_medoids)
old_medoids = copy.deepcopy(cur_medoids)
for i in range(N):
for j in range(k):
if not i == j: #非中心点对象依次替换中心点对象
tmp_medoids = copy.deepcopy(cur_medoids)
tmp_medoids["cen_idx"][j] = i
Assment(data, tmp_medoids)
total_cost(data, tmp_medoids)
if(best_medoids["t_cost"]>tmp_medoids["t_cost"]):
best_medoids = copy.deepcopy(tmp_medoids) # 替换中心点对象
cur_medoids = copy.deepcopy(best_medoids) #将最好的中心点对象对应的字典信息返回
print("current total cost is:",cur_medoids["t_cost"])
return cur_medoids
def test():
dim = 2
N = 100
# create datas with different normal distributions.
d1 = np.random.normal(1, .2, (N, dim))
d2 = np.random.normal(2, .5, (N, dim))
d3 = np.random.normal(3, .3, (N, dim))
data = np.vstack((d1, d2, d3))
# need to change if more clusters are needed .
k = 3
medoids = PAM(data, k)
fig = plt.figure()
rect = [0.1, 0.1, 0.8, 0.8] # figure的百分比,从figure 10%的位置开始绘制, 宽高是figure的80%
ax1 = fig.add_axes(rect, label='ax1', frameon=True)
ax1.set_title('Clusters Result')
# plot different clusters with different colors.
ax1.scatter(data[medoids[0], 0], data[medoids[0], 1], c='r')
ax1.scatter(data[medoids[1], 0], data[medoids[1], 1], c='g')
ax1.scatter(data[medoids[2], 0], data[medoids[2], 1], c='y')
ax1.scatter(data[medoids['cen_idx'], 0], data[medoids['cen_idx'], 1], marker='x', s=500)
plt.show()
if __name__ =='__main__':
test()
运行结果如下

PS: 编码过程中遇到的函数总结:
- dist.argmin(axis=1) 沿着行的方向寻找最小值的索引,这里要求dist必须是数组类型
五、总结
- 1、当存在噪声和离群点时,k-medoids方法比k-means方法更鲁邦。
- 2、k-means每次迭代的复杂度是 O ( n k t ) O(nkt) O(nkt) 其中n是样本总数, k是簇数,t是迭代次数
- 3、k-medoids每次迭代的复杂度是 O ( k ( n − k ) 2 ) O(k(n-k)^{2}) O(k(n−k)2),,显然是非常耗时的。
参考资料
- K中心点算法之PAMhttps://www.cnblogs.com/king-lps/p/7775108.html
- (数据科学学习手札13)K-medoids聚类算法原理简介&Python与R的实现https://www.cnblogs.com/feffery/p/8595315.html
























 419
419

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








