1 重要知识点
在专栏我已经系统总结了遗传算法:*智能优化算法(持续更新中…),下面我们先讲解重要知识点,然后用Python和Matlab分别实现。
1.1 遗传算法
遗传算法是一种模拟达尔文生物进化论和遗传变异的智能算法。这种算法具有鲁棒性(用以表征控制系统对特性或参数扰动的不敏感性)较强,实现的步骤规范、简单通用等优点,在人工智能、多目标决策、社会及经济等领域都有大量运用。但一般遗传算法存在一定的局限性:收敛速度慢、迭代的次数多,易过早收敛,容易陷入局部最优解。
1**.2 量子计算**
量子计算为量子力学与信息科学的综合交叉学科。量子计算具有量子力学的并行性,计算速度更快;同时,量子状态多种多样,在进行最优解的搜索时极少陷入局部的极值。
1.3 量子遗传算法
量子遗传算法将量子的态矢量引入遗传算法,利用量子比特的概率幅应用于染色体的编码。一条染色体是多个量子状态的叠加。并使用量子旋转门实现染色体的变异更新。因此量子遗传算法具有迭代次数少,运行速度快,能以较少种群进行遗传变异,搜索范围广,难以陷入局部的极值等优点。
想更深入了解量子遗传算法,可以阅读以下两篇文章:


简单通过一幅图介绍量子遗传算法的特性,黄色个体上有当前最优值,黑色为某其他个体,迭代一次,黑色个体会旋转并向黄色个体靠近,量子遗传算法用旋转门取代了原有的交叉过程。个体自带两个函数值,寻优收敛速度更快,有旋转过程寻找最优,旋转公式中也有惯性公式,即随着迭代次数增加旋转幅度会降低,整体比较遗传算法,更不易陷入局部最优。

2 操作步骤
2.1 运用量子比特初始化父代染色体
在传统的二进制计算中,用|0》与|1》表示数值,它们即为比特。类似的,在量子计算的过程中,使用与表示量子的两种基本状态,称其为量子比特。量子比特的各种状态,用下式表示:
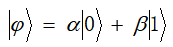
其中,α与β为一复数,称之为概率幅,且α与β满足
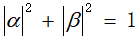
其等效于

量子状态又可表示为:
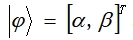
量子遗传算法将染色体上的基因用量子比特表示,从而增加种群多样性。随机生成m个染色体,每个染色体上的基因用量子比特表示 ,且初始化为
,且初始化为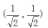 。
。
2.2 在量子遗传算法中,染色体采用量子位的概率幅进行编码,编码方案如下:
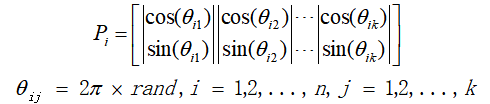
Pi为第i个基因,为量子比特的相位,n为染色体数目,k为量子位的位数即解空间的维数,rand是[0,1]范围内的随机数。每个量子位为分上下两行,分别对应两个量子基本态的概率幅,满足归一化条件,因此每个个体包含上下两条文化基因链,每条基因链是优化问题的一个候选解。由此可知,量子遗传算法在种群规模不变的情况下,候选解个数比遗传算法多一倍,增加了解空间的多样性,提高了寻优成功的概率。
2.3 对初始化种群中的每一个个体进行测量。
随机生成一个数x∈[0,1],若
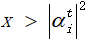
则测量值取1,








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 5199
5199











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








