【论文笔记】| 蛋白质大模型ProLLaMA
ProLLaMA: A Protein Large Language Model for Multi-Task Protein Language Processing
Peking University
Theme: Domain Specific LLM
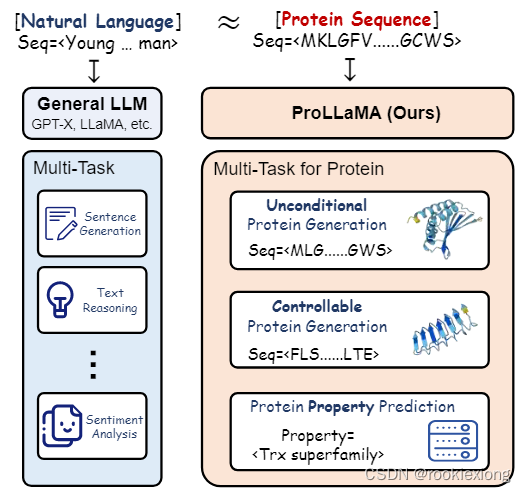
Main work:
当前 ProLLM 的固有局限性:(i)缺乏自然语言能力,(ii)指令理解不足
利用低秩适应(LoRA)并采用两阶段训练方法,将任何通用 LLM 转换为能够同时处理多个 PLP 任务的 ProLLM,在无条件/可控蛋白质序列生成、蛋白质属性预测任务中取得了最先进的结果
Method:
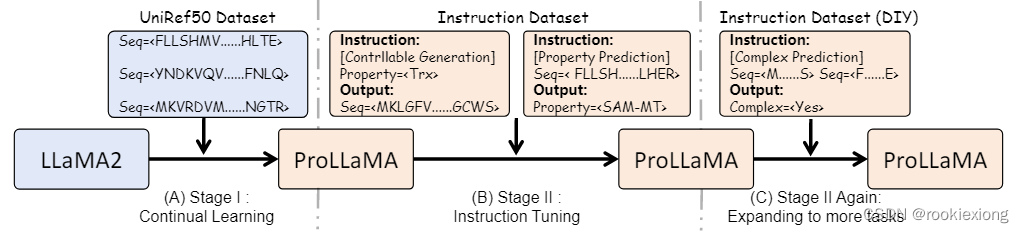
1. Continual Learning on Protein Language
当前的 ProLLM 缺乏自然语言能力,这阻碍了多任务能力,利用预训练的 LLAMA2 对蛋白质语言执行持续学习(类比学习新语言,模型在保留原始自然语言能力的同时学习蛋白质语言)
基于 UniRef50 构建了一个数据集,并用特定的前缀和后缀对每个蛋白质序列进行预处理。
在 LLAMA2 的每个Decoder layer,我们将 LoRA 添加至 W q , W k , W v , W o , W u p , W g a t e , W d o w n W_q,W_k,W_v,W_o,W_{up},W_{gate},W_{down} Wq,Wk,Wv,Wo,Wup,Wgate,Wdown以及 E m b e d Embed Embed和 G e n e r a t i o n H e a d Generation \ Head Generation Head layers (token可能在蛋白质序列和自然语言中具有不同的含义,需要对同一token进行不同的embedding)
LoRA rank-128, AdamW optimizer, peak learning rate(cosine annealing scheduler)-0.05, training epoch-1.
2. Performing Various Tasks
对上一步中获得的 ProLLaMA 执行指令微调,采用自回归方式进行训练
L
(
Θ
)
=
E
x
∼
D
[
−
∑
i
log
p
(
x
i
∣
u
,
x
0
,
x
1
,
…
,
x
i
−
1
;
Θ
)
]
\mathcal{L}(\Theta)=\mathbb{E}_{\boldsymbol{x}\sim\mathcal{D}}\left[-\sum_i\log p(x_i|\boldsymbol{u},x_0,x_1,\ldots,x_{i-1};\Theta)\right]
L(Θ)=Ex∼D[−i∑logp(xi∣u,x0,x1,…,xi−1;Θ)]
LoRA rank-64, AdamW optimizer, peak learning rate(cosine annealing scheduler)-0.05, training epoch-2.
3. Expanding to More Tasks
基于上述模型针对特定任务再次进行指令微调
参考文献
Lv L, Lin Z, Li H, et al. ProLLaMA: A Protein Large Language Model for Multi-Task Protein Language Processing[J]. arXiv preprint arXiv:2402.16445, 2024.






















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








