论文题目:MolCA: Molecular Graph-Language Modeling with Cross-Modal Projector and Uni-Modal Adapter
abstract:
语言模型(LMs)在各种一维文本相关任务中展示了令人印象深刻的分子理解能力。然而,它们固有地缺乏二维图形感知——这是人类专业人员理解分子拓扑结构的关键能力。为了弥合这一差距,我们提出了MolCA:使用跨模态投影器和单模适配器的分子图-语言建模。MolCA通过跨模态投影器使LM(例如Galactica)能够理解基于文本和图形的分子内容。具体而言,跨模态投影器被实现为Q-Former,以连接图形编码器的表示空间和LM的文本空间。此外,MolCA使用单模适配器(即LoRA)使LM有效地适应下游任务。与先前通过跨模态对比学习将LM与图形编码器耦合的研究不同,MolCA保留了LM生成开放式文本的能力,并增加了二维图形信息。为展示其有效性,我们在分子描述、IUPAC名称预测和分子-文本检索任务上广泛测试了MolCA,在这些任务上,MolCA显著优于基线。我们的代码和检查点可以在MolCA找到。
1 Introduction
语言模型(LMs)在各个领域都取得了显著的成就。值得注意的是,LMs预训练数据中丰富的生化文献使得LMs能够获得对生化概念和分子属性的高级理解。这可以从它们在生化和医学问答基准测试中的出色表现中看出。因此,将这些LMs纳入化学和生物学研究变得越来越紧迫。

为此,我们旨在利用LMs来理解分子。如Figure 1a所示,大多数现有的LMs通过SMILES字符串来表示分子,并将它们以类似于文本的方式处理。虽然方便,但将分子视为字符串忽略了对人类专业人士理解分子结构至关重要的分子的二维图表示。为了解决这个问题,最近的工作将分子表示为图,并使用图神经网络作为分子图编码器。图编码器通过跨模态对比学习与LM一起训练,如Figure 1b所示。然而,跨模态对比学习的应用范围有限:它适用于检索任务,但对于开放式分子到文本生成任务来说是不够的,例如分子描述和分子的IUPAC名称预测。这是因为分子到文本生成是一个条件生成任务。它要求LM理解2D图作为生成条件,这是对比学习无法实现的。Su等人(2022年)尝试直接将2D图的表示输入到LMs中,但显示出的改进有限。
为了弥补这一差距,我们设计了MolCA:使用跨模态投影器和单模态适配器的分子图-语言建模。MolCA使LM能够将2D图作为输入来理解,从而有效地调节分子到文本的生成过程。为了使LM能够理解2D图,我们确定关键挑战是跨模态对齐:将2D图的表示转换为文本空间中的一维软提示,以便LM能够理解。这种转换由跨模态投影器促进,它架起了图编码器的表示空间和LM的输入空间之间的桥梁,如Figure 1所示。具体来说,我们由于其在视觉-语言任务中的有效性,将跨模态投影器实现为Q-Former。有了有效的跨模态投影器,我们可以利用现有的大型LM进行分子到文本的生成。然而,鉴于具有数十亿规模参数的大型LM,其下游微调的效率成为了一个新问题。因此,我们将LM与单模态适配器集成,即LoRA,以实现其高效适应。

正如Figure 2所示,MolCA使用一个三阶段的训练流程来整合其组件。两个预训练阶段旨在发展跨模态投影器的跨模态对齐能力。在预训练阶段1中,投影器和编码器被训练以提取与文本最相关的分子特征。这个阶段赋予了生成的模型强大的分子-文本检索能力。在预训练阶段2中,跨模态投影器连接到一个冻结的LM并被训练用于分子描述。这个任务迫使跨模态投影器产生LM能够理解的软提示。在最后一个阶段,MolCA针对下游生成任务进行微调。
我们的贡献可以总结如下:
- 我们提出了MolCA,这是一种开创性的分子语言建模方法。MolCA使LM能够感知2D分子图,从而促进分子到文本的生成任务。
- MolCA在多种基准测试中树立了新的最高标准。它在CheBI-20和我们策划的PubChem324k数据集上的分子描述任务中,分别超过了基线2.1和7.6 BLEU-2。此外,在预测IUPAC名称时,MolCA比基线显示出显著的10.0 BLEU-2优势。在分子-文本检索方面,MolCA在PubChem324k上超过了基线20%的检索准确率,并在PCDes和MoMu数据集上取得了最佳性能。
- 我们进行了消融研究,以展示MolCA将2D图整合到LMs中对分子相关任务的有效性。此外,我们的定量分析表明,整合2D图有助于提高LM计算分子内功能团数量的能力。
2 Model Architecture
在这里,我们介绍了MolCA架构的三个关键组成部分:
1)用于2D结构理解的图编码器
2)用于文本生成的语言模型(LM)
3)连接图编码器和LM的跨模态投影器。
我们将在第3.3节中描述单模态适配器。
Graph Encoder
图编码器(Graph Encoder)是一种基于图神经网络(GNN)的编码器,用于将分子的2D结构信息编码成可以被机器学习模型理解的数值特征。在MolCA架构中,图编码器负责理解分子的2D结构模式。具体来说,采用了一种基于图的编码器来处理分子图,这个编码器是一个五层的GINE(Graph Isomorphism Network),它在ZINC15数据集上预训练了200万个分子,通过对比学习的方法来提取与文本最相关的分子特征。给定一个分子图
g
\mathcal{g}
g,图编码器
f
\mathcal{f}
f能够为
g
\mathcal{g}
g中的每个节点生成结构感知的特征,表示为:
f
(
g
)
=
Z
∈
R
∣
g
∣
×
d
(
1
)
\mathcal{f}(\mathcal{g})=\mathrm {Z} \in\mathbb{R}^{|\mathcal{g}|\times d}(1)
f(g)=Z∈R∣g∣×d(1)
其中
∣
g
∣
|\mathcal{g}|
∣g∣表示分子图中节点的数量。
Language Model
为了实现有效的文本生成性能,我们采用了Galactica作为基础的语言模型(LM)。Galactica在大量科学文献上进行了预训练,涵盖了化学、生物学和医学等领域。它在基于文本的科学问答基准测试中的出色表现突显了其对高级生化概念的理解能力。值得注意的是,Galactica能够处理分子的一维SMILES字符串,这可能对我们的下游任务有益。Galactica是一个基于OPT(Zhang等人,2022年)架构的仅解码器的变换器语言模型。
Cross-Modal Projector

我们将跨模态投影器实现为一个Querying-Transformer(Q-Former),以将图编码器的输出映射到语言模型的输入文本空间。如Figure 3所示,Q-Former在处理2D分子图和1D文本时有不同的程序。给定文本输入,Q-Former在开头插入 ′ [ C L S ] ′ '[CLS]' ′[CLS]′标记,并通过 N N N层自注意力模块和前馈网络处理文本。当预训练任务是文本生成时,自注意力模块采用因果掩码。另一方面,给定一个分子图 g \mathcal{g} g,Q-Former作为一个分子特征提取器工作。具体来说,它维护一组可学习的查询标记 { q k } k = 1 N q {\{\mathrm{q}_k\}}_{k=1}^{N_q} {qk}k=1Nq 作为输入。这些查询标记可以通过交叉注意力模块与图编码器的输出 Z \mathrm {Z} Z交互并提取分子特征。交叉注意力模块每两层添加一次。此外,查询标记也可以通过相同的自注意力模块与文本输入交互。请注意,查询标记和文本输入由不同的前馈网络处理,以保持处理分子和文本的能力。
我们从Sci-BERT初始化Q-Former,Sci-BERT是一个仅编码器的变换器,预训练在科学出版物上。Q-Former的交叉注意力模块是随机初始化的。
3 Training Pipeline
本节详细介绍了MolCA的三阶段训练流程(Figure 2)。前两个预训练阶段利用分子-文本对数据集 D = { ( g 1 , y 1 ) , ( g 2 , y 2 ) , . . . } \mathcal{D}=\{(g_1,y_1),(g_2,y_2),...\} D={(g1,y1),(g2,y2),...}来训练跨模态投影器和图编码器。预训练的目标是将2D分子图转换为冻结的语言模型(LM)能够理解的软提示。微调阶段则专注于高效适应下游生成任务。
3.1 Pretrain Stage 1: Learning to Extract Text Relevant Molecule Representations
在这个阶段,我们的目标是优化跨模态投影器(即Q-Former)以提取与文本输入最相关的分子特征。这个阶段作为跨模态投影器在连接到语言模型(LM)之前的“热身”训练。受BLIP2(Li等人,2023年)的启发,我们同时应用了三个为Q-Former架构量身定制的跨模态预训练任务:分子-文本对比、分子-文本匹配和分子描述。这些预训练任务使Q-Former具备了强大的分子-文本检索能力。因此,我们保存了这个阶段产生的模型,以用于下游的检索任务。现在我们详细说明三个预训练任务:
Molecule-Text Contrasting (MTC)
我们应用跨模态对比学习来训练Q-Former提取与文本相关的分子特征。在这个任务中,查询标记和文本输入被分别送入Q-Former(见Figure 3下方)以获得Q-Former的分子表示和文本表示。
形式上,令
{
(
g
1
,
y
1
)
,
.
.
.
,
(
g
B
,
y
B
)
}
\{(g_1,y_1),...,(g_B,y_B)\}
{(g1,y1),...,(gB,yB)}为一批分子-文本对。我们将
g
i
g_i
gi的Q-Former表示为
{
m
i
k
}
k
=
1
N
q
\{m_{ik}\}_{k=1}^{N_q}
{mik}k=1Nq(每个元素对应于一个query token)。将
y
i
y_i
yi的Q-Former表示为
t
i
t_i
ti([CLS]的表示)。对于任意的
i
,
j
∈
[
1
,
B
]
i,j\in[1,B]
i,j∈[1,B],我们通过计算
t
i
t_i
ti与
{
m
j
k
}
k
=
1
N
q
\{m_{jk}\}_{k=1}^{N_q}
{mjk}k=1Nq中每个元素之间的最大相似度来衡量
t
i
t_i
ti与
{
m
j
k
}
k
=
1
N
q
\{m_{jk}\}_{k=1}^{N_q}
{mjk}k=1Nq之间的相似度。MTC损失
ℓ
M
T
C
\ell _{MTC}
ℓMTC的计算公式如下:

这里的 c o s ( ⋅ , ⋅ ) / τ cos(·, ·)/τ cos(⋅,⋅)/τ是温度缩放的余弦相似度。温度 τ τ τ经验性地设置为0.1。
Molecule-Text Matching (MTM)
MTM是一个二元分类任务,目标是预测一个分子-文本对是否匹配(正样本)或不匹配(负样本)。如Figure 3所示,MTM允许查询和文本通过同一个自注意力模块进行交互。通过这种方式,查询可以从分子和文本中提取多模态信息。对于MTM预测,我们在所有查询的Q-Former表示进行平均池化后附加一个线性分类器。设
ρ
(
g
,
y
)
ρ(g, y)
ρ(g,y)表示MTM预测的
(
g
,
y
)
(g, y)
(g,y)匹配的概率。MTM损失
ℓ
M
T
M
ℓ_{MTM}
ℓMTM可以表示为:

其中 U ( 1 , B ) U(1,B) U(1,B)为均匀分布, y j y_j yj和 g k g_k gk为批次中的随机负样本。
与MTC类似,MTM也计算分子-文本对之间的相似性。不同之处在于,与MTC使用的简单余弦相似性相比,MTM可以通过自注意和交叉注意模块捕获分子和文本之间更细粒度的相似性。因此,在检索实验中,我们首先使用MTC检索前 k k k个样本,然后使用MTM重新排序,从而提高性能。
Molecule Captioning (MCap)
MCap的目标是根据分子表示生成分子的文本描述。为了完成这个任务,我们采用了一种特殊的掩码策略在自注意力模块中,以确保查询学会提取与文本描述相对应的分子特征。具体来说,我们为查询使用了双向自注意力掩码,允许它们相互看到,但看不到文本标记。此外,我们对同一自注意力模块上的文本应用因果掩码,以进行文本描述的自回归解码。每个文本标记可以看到查询和前面的文本,但看不到后面的文本标记。由于文本标记不能直接与图编码器交互,它们必须从查询中获取分子信息,这迫使查询通过交叉注意力模块提取分子信息。设
(
p
1
(
y
∣
g
)
)
( p_1(y|g) )
(p1(y∣g))为Q-Former生成文本
(
y
)
( y )
(y)对于图
(
g
)
( g )
(g)的概率。我们使用以下损失函数:

3.2 Pretrain Stage 2: Aligning 2D Molecular Graphs to Texts via Language Modeling
在这个阶段,我们的目标是将跨模态投影器的输出与冻结的语言模型(LM)的文本空间对齐。如Figure 4所示,我们将跨模态投影器对2D分子图的表示作为输入喂给冻结的LM,并训练模型生成分子的文本描述。这个过程鼓励跨模态投影器提供LM能够理解的表示,以促进文本生成。此外,我们还使用分子的1D SMILES来指导生成(参见Figure 4)。这是因为大多数LM在预训练期间使用SMILES。因此,这些LM已经建立了SMILES和它们的文本上下文之间的一些关联。因此,包含SMILES可以潜在地提示相应的生化知识。另一方面,整合2D图可以帮助捕捉从1D SMILES中难以学习到的结构模式。我们将在后续的实验中展示,结合2D图和1D SMILES可以提升性能。

正式地,考虑一个分子-文本对
(
g
,
y
)
(g, y)
(g,y),
g
g
g的SMILES表示
s
s
s,跨模态投影器对
g
g
g的表示记作
{
m
k
}
k
=
1
N
q
\{m_k\}_{k=1}^{N_q}
{mk}k=1Nq。我们定义
p
2
(
⋅
)
p_2(\cdot)
p2(⋅)为由冻结的语言模型参数化的文本分布。我们通过最小化以下损失函数来优化跨模态投影器和图编码器:

3.3 Fine-tune Stage: Uni-Modal Adapter for Efficient Downstream Adaptation

在这个阶段,我们对MolCA进行微调以适应下游的生成任务。如Figure 5所示,我们在分子表示之后添加了任务描述的文本提示。然后,我们应用语言模型损失来微调MolCA,以用于生成任务,例如分子的IUPAC名称预测。
Uni-Modal Adapter
在MolCA架构中,语言模型(LM)占据了计算开销的很大一部分:它可能拥有大约10亿个参数,而跨模态投影器和图编码器总共只有大约1亿个参数。因此,我们采用了一种单模态适配器来实现LM对下游任务的高效适应。具体来说,我们采用了LoRA(Low-Rank Adaptation)适配器,因为它的实现简单且表现优异。
如Figure 5所示,在LM中选定的权重矩阵(例如,
W
∈
R
d
1
×
d
2
W ∈ \mathbb{R}^{d_1×d_2}
W∈Rd1×d2)中,LoRA会以并行的方式添加一对秩分解矩阵(例如,
B
A
BA
BA,其中
B
∈
R
d
1
×
r
B \in \mathbb{R}^{d_1 \times r}
B∈Rd1×r,
A
∈
R
r
×
d
2
A \in \mathbb{R}^{r \times d_2}
A∈Rr×d2。原始的
h
=
W
x
h = Wx
h=Wx层被更改为:
h
=
W
x
+
B
A
x
,
(
6
)
h=Wx+BAx,(6)
h=Wx+BAx,(6)
其中
W
W
W保持冻结状态,新添加的
B
A
BA
BA在适应过程中进行训练。由于
r
≪
m
i
n
(
d
1
,
d
2
)
r \ll min(d_1, d_2)
r≪min(d1,d2),LoRA可以有效地使LM适应下游任务,同时只需要很少的存储空间来保存梯度。
4 Experiments
4.1 Experimental Setting
这里我们简要地介绍一下实验设置。详情见附录B。
PubChem324k Dataset

我们收集了PubChem-324k数据集,该数据集包含了来自PubChem网站的324k个分子-文本对。如Table 1所示,数据集中包括了许多信息量不大的文本,例如“The molecule is a peptide”。因此,我们从中抽取了一个包含15k对、文本长度超过19个单词的高质量子集,用于下游任务。这个高质量子集进一步被随机划分为训练集/验证集/测试集。剩余的数据集虽然噪声较多,但被用于预训练。此外,我们还过滤了预训练子集,排除了其他下游数据集的验证集/测试集中的分子,包括CheBI-20、PCDes和MoMu数据集。过滤后的数据集总共包含了313k个分子-文本对。
Baselines
在生成任务中,我们将MolCA与以下基线模型进行比较:T5、MolT5和MoMu。在分子-文本检索方面,我们还包括了以下方法:MoleculeSTM、KV-PLM和Sci-BERT。
4.2 Molecule Captioning

在这一阶段,我们对MolCA进行了下游生成任务的微调。如Figure 5所示,我们在分子表示之后添加了任务描述的文本提示。然后,我们应用语言模型损失来对MolCA进行微调,以用于生成任务,例如分子的IUPAC名称预测。具体来说,我们在PubChem324k和CheBI-20数据集上评估了MolCA的性能,使用了基于Galactica 1.3 B _{1.3B} 1.3B、Galactica 125 M _{125M} 125M和MolT5-Large的语言模型,并采用了BLEU、ROUGE和METEOR作为评估指标来评估MolCA性能。实验结果如Table 2所示,MolCA在各项指标上表现优于基准模型,尤其是 G a l a c 1.3 B Galac_{1.3B} Galac1.3B版本表现最佳。此外,MolCA-Galac 125 M _{125M} 125M版本在各项指标上都优于规模较大的基准模型,表明MolCA的优势并非仅限于模型规模。
- MolCA在分子描述任务中的表现始终优于基线模型,优势非常明显。具体来说,使用Galac 1.3 B _{1.3B} 1.3B作为基础语言模型的MolCA在所有评估指标上都取得了最佳性能。在PubChem324k数据集上,MolCA的BLEU-2得分比基线模型高出7.6,在CheBI-20数据集上高出2.1,这表明MolCA在分子描述任务上具有显著的优势。
- MolCA在不同规模的模型中都展现出了优势,其中Galac 125 M _{125M} 125M版本在所有评估指标上都超过了规模更大的基线模型,这说明MolCA的优势并不局限于模型的规模大小。
4.3 IUPAC Name Prediction
国际纯粹与应用化学联合会(IUPAC)建立了一个标准化的化学化合物命名系统,即IUPAC名称。值得注意的是,这个命名系统依赖于识别特定的分子结构,包括烃链和双/三键。因此,正确预测IUPAC名称表明了模型理解分子结构的能力。我们使用PubChem324k的训练集对MolCA和基线模型进行微调,以生成分子的IUPAC名称。如Table 3所示,MolCA在各项指标上一致优于基线模型,BLEU-2的平均提升达到了10.0,突出了MolCA在理解分子结构方面的优势。

4.4 Molecule-Text Retrieval

在PubChem324k、PCDes和MoMu数据集上,我们对MolCA进行了分子-文本检索的评估。具体来说,我们评估了MolCA在第一阶段预训练后的检查点,而没有进行进一步的微调。在所有实验中,MolCA首先使用分子-文本对比(MTC)检索出前128个候选对象,然后利用分子-文本匹配(MTM)模块进行重新排序。我们选择准确率(Accuracy, Acc)和召回率@20(Recall@20, R@20)作为评估指标,并对整个测试集的检索性能进行了报告。如Table 4所示,我们观察到以下几点:
- MolCA在性能上优于基线模型。具体来说,在PubChem324k数据集中,MolCA的准确率比基线模型提高了20%以上。在PCDes和MoMu数据集中,MolCA也持续优于基线模型,证明了其在分子-文本检索方面的有效性。
- 结合MTM显著提升了MolCA的性能。这可以归因于MTM模块能够通过交叉注意力和自注意力模块,对分子特征和文本之间的长期交互进行建模。
- MolCA的优异表现部分可以归因于我们更大的预训练数据集——PubChem324k。如Table 3(a)所示,我们比较了MoMu原始检查点(在15k分子-文本对上预训练)与我们使用PubChem324k数据集重新训练的MoMu的性能。后者的检索准确率提高了25%以上。
4.5 Ablation Study on Representation Types
在这里,我们对分子的两种表示类型进行了消融研究:1D SMILES和2D图形。我们比较了MolCA与其两个变体:1) 1D SMILES:一个仅使用1D SMILES进行预训练和微调的语言模型。为了进行公平比较,我们在这个变体上预训练了PubChem324k的预训练子集,然后进行下游适应;2) 2D Graph:这个变体遵循原始MolCA的训练流程,除了在第二阶段的预训练和微调阶段不使用1D SMILES。
End Task Ablation

Table 5展示了分子到文本生成和分子属性预测任务的结果。我们可以观察到,结合2D图形和1D SMILES能够提高所有比较任务的性能。这一结果证明了MolCA在结合分子的2D图形表示方面的有效性。
Counting Functional Groups (FGs)

我们对MolCA在计算分子中85种功能团(FGs)的能力进行了消融研究。功能团是分子的一个子图,它在不同分子中表现出一致的化学行为。正确计算功能团有助于理解分子的性质。如Figure 6所示,整合2D图形显著提高了MolCA在计算功能团数量上的性能,从而增强了其理解分子结构的能力。
5 Related Works
本节简要回顾了分子相关文献。我们在附录A中讨论了MolCA与视觉-语言预训练方法的关系。
Molecule Understanding via 1D Language Modeling
由于训练语料库中包含大量的生化文献,一些开放域的语言模型(LMs)已经获得了对分子和化学概念的高级理解。这通过它们在与文本相关的生化和医学问答基准测试中的出色表现得到了证明。在这些语言模型中,Galactica表现出了竞争优势,因为它主要使用科学文献组成的语料库进行训练。专注于化学领域,KV-PLM通过在一维SMILES上应用掩码语言建模损失来建模分子。Vaucher等人(2021)提出通过阅读化学反应方程式来预测化学实验动作。MolT5提出了几种基于T5的语言模型,用于SMILES到文本和文本到SMILES的翻译。此外,Christofidellis等人(2023)提出对T5进行微调,以用于化学反应预测和逆合成任务。与这些仅使用一维SMILES来表示分子的方法不同,MolCA旨在使语言模型能够感知分子的二维图形表示。
Molecule-Text Contrastive Learning
由于分子-文本检索系统的需求,Text2Mol采用了跨模态对比学习来训练一个基于图卷积网络(GCNs)的分子图编码器和一个基于Sci-BERT的文本编码器。随后的工作提出了一些改进,包括增加跨模态对比学习损失和将模型应用于基于文本的分子编辑。然而,跨模态对比学习不适合开放式的条件生成任务,因为它专注于学习相似性函数。为了解决这个问题,我们提出了MolCA,以使语言模型能够理解2D分子图,从而促进MolCA进行开放式分子到文本的生成能力。
6 Conclusion and Future Works
在这项工作中,我们提出了MolCA,一种新颖的分子语言建模方法。MolCA旨在使语言模型(LMs)能够感知2D图形,以进行分子到文本的生成。为此,MolCA特色了一个跨模态投影器,将2D图形的表示映射到LMs的文本空间。它还采用了单模态适配器,以实现高效的下游适应。MolCA在分子描述和分子-文本检索基准测试中取得了最先进的性能。展望未来,我们对探索LMs在3D分子建模和药物发现任务中的应用感兴趣。
Limitations
这项工作专注于利用语言模型(LMs)的生成能力来处理分子-文本任务。然而,LMs的其他一些有趣的能力,比如上下文学习(in-context learning)和思维链推理(chain-of-thought reasoning),超出了这项研究的范围。这些有趣的能力将留给未来的探索。
尽管MolCA在性能上相较于基线模型有所提升,但目前分子描述的性能还不足以满足实际应用的需求。这可能归因于预训练数据的规模。据我们所知,我们的PubChem324k数据集是迄今为止最大的分子-文本对数据集。然而,与视觉-语言预训练的大约1000万规模的数据集相比,我们的数据集包含324k个数据点,规模相对较小,这限制了模型的性能。可能的补救措施包括从生化文献中挖掘弱监督数据。
Broader Impacts
我们的工作在分子描述和分子-文本检索方面建立了新的最高标准。它在两个方面产生了更广泛的影响:1)对于化学专业人士来说,我们的分子描述和分子-文本检索方法可能是有用的工具,有可能加快他们的研究进程;2)对于没有专门化学知识的人来说,我们的方法可能提供了一种更经济的方式来获取分子的基本化学信息。
我们的模型也存在大多数语言模型的风险。它可能会生成不准确的信息,并且可能被滥用来产生有偏见的内容。此外,考虑到我们训练数据规模的局限性,我们强烈建议在将我们的模型应用于实际应用之前进行严格的测试。
Acknowledgement
这项研究得到了国家自然科学基金(National Natural Science Foundation of China)的支持,项目编号为92270114。此外,还得到了安徽省大学协同创新计划(University Synergy Innovation Program of Anhui Province)的支持,项目编号为GXXT-2022-040。研究工作还得到了谷歌云研究学分计划(Google Cloud Research Credit program)的支持,奖项编号为6NW8-CF7K-3AG4-1WH1。研究同时得到了NExT研究中心(NExT Research Center)的支持。
A Complete Related Works
我们在这里提供完整的文献回顾。除了本文主体部分讨论的与分子相关的文献,我们还讨论了MolCA与视觉-语言预训练的关系。
Molecule Understanding via 1D Language Modeling
由于训练语料库中包含大量的生化文献,一些开放域的语言模型(LMs)已经获得了对分子和化学概念的高级理解。这通过它们在与文本相关的生化和医学问答基准测试中的出色表现得到了证明。在这些语言模型中,Galactica因其主要使用科学文献组成的语料库而展现出了竞争优势。专注于化学领域,KV-PLM通过在一维SMILES上应用掩码语言建模损失来建模分子。Vaucher等人提出通过阅读化学反应方程式来预测化学实验动作。MolT5提出了几种基于T5的语言模型,用于SMILES到文本和文本到SMILES的翻译。此外,Christofidellis等人提出对T5进行微调,以用于化学反应预测和逆合成任务。
MolCA与这些仅使用一维SMILES来表示分子的方法不同。相反,MolCA旨在使语言模型能够感知分子的二维图形表示。
Molecule-Text Contrastive Learning
在分子-文本对比学习(Molecule-Text Contrastive Learning)领域,Text2Mol利用跨模态对比学习训练了一个基于图卷积网络(GCNs)的分子图编码器和一个基于Sci-BERT的文本编码器。随后的研究提出了一些改进,包括增加跨模态对比学习损失和将模型应用于基于文本的分子编辑。然而,跨模态对比学习不适合开放式的条件生成任务,因为它主要关注学习相似性函数。为了解决这个问题,我们提出了MolCA,以使语言模型能够理解2D分子图,从而增强MolCA在开放式分子到文本生成任务中的能力 。
Vision-Language Pretraining (VLP)
视觉-语言预训练(Vision-Language Pretraining,VLP)和分子语言建模都旨在弥合文本与另一种模态之间的差距。特别是,CLIP等VLP方法使用对比学习来连接视觉编码器和文本编码器。这些方法可以应用于图像-文本检索和零样本图像分类等任务。最近,一系列VLP工作 表明,视觉特征可以与LMs的文本空间对齐。这种跨模态对齐允许LMs利用其语言生成和少样本学习能力来处理多模态任务。MolCA从这些发现中汲取灵感。据我们所知,我们是第一个将2D分子图与LMs的文本空间对齐的。此外,我们引入了一个单模态适配器来提高下游任务的适应效率。
B Experimental Settings
Pretrain Settings
MolCA的第一阶段预训练有50个周期,第二阶段预训练有10个周期。Q-Former有8个查询标记( N q = 8 N_q = 8 Nq=8)。我们的优化器配置遵循(Li等人,2023年)。我们使用AdamW优化器,权重衰减为0.05。学习率由线性预热和余弦衰减的组合来安排。峰值学习率为 1 e − 4 1e^{-4} 1e−4,预热有1000步。
Molecule Captioning
MolCA在微调阶段进行了100个周期的训练,使用了与之前相同的优化器和学习率调度配置。LoRA适配器是通过OpenDelta库和PEFT库实现的。在PubChem324k数据集上,我们将LoRA的秩 r r r设置为8,并将其应用于Galactica模型的 [ q _ p r o j , v _ p r o j ] [q\_proj, v\_proj] [q_proj,v_proj]模块。这样的配置产生了一个具有200万参数的LoRA适配器,占Galactica 1.3 B _{1.3B} 1.3B参数的0.12%。对于CheBI-20数据集,我们将LoRA的秩 r r r设置为16,并将其应用于Galactica模型的 [ q p r o j , v p r o j , o u t p r o j , f c 1 , f c 2 ] [q_proj, v_proj, out_proj, fc1, fc2] [qproj,vproj,outproj,fc1,fc2]模块。这样的配置产生了一个具有1200万参数的LoRA适配器,占Galactica 1.3 B _{1.3B} 1.3B参数的0.94%。
IUPAC Name Prediction
我们为实验中使用的训练集、验证集和测试集中的分子收集了IUPAC名称。实验使用的超参数与分子描述实验相同。我们在分子表示之后添加了文本提示“分子的IUPAC名称是”作为任务描述(参见Figure 5)。
Molecule-Text Retrieval
我们使用MolCA在第一阶段预训练后的检查点进行检索,没有在任何其他数据集上进行微调。这与(Su等人,2022年;Liu等人,2022年)中零样本检索的设置相似。
Molecule Property Prediction
遵循(Hu等人,2020年)的做法,我们对模型进行100个周期的微调,并报告由验证集选择的测试性能。对于分子分类任务,我们在语言模型最后一层隐藏状态的平均池化后附加一个线性分类器。我们使用AdamW优化器,固定学习率为 1 e − 4 1e^{-4} 1e−4,权重衰减为0.05。这个实验使用了与PubChem324k数据集中分子描述实验相同的LoRA配置。
Counting Functional Groups (FGs)
我们使用PubChem324k训练集中的分子进行微调,并使用验证集中的分子进行评估。遵循(Rong等人,2020年)的做法,我们使用RDkit来获取每个分子中功能团(FGs)的真实计数。对于每种功能团类型,我们使用一个单独的线性分类器来回归其数量。我们的模型是使用均方误差(Mean Square Error,MSE)损失函数进行训练的。其他设置,包括优化器和LoRA,与分子属性预测实验相同。
Galactica
遵循(Taylor等人,2022年)中的指示,我们在将SMILES序列输入Galactica之前,用特殊的标记[START_I_SMILES]和[END_I_SMILES]将它们包装起来。
PubChem324k Dataset
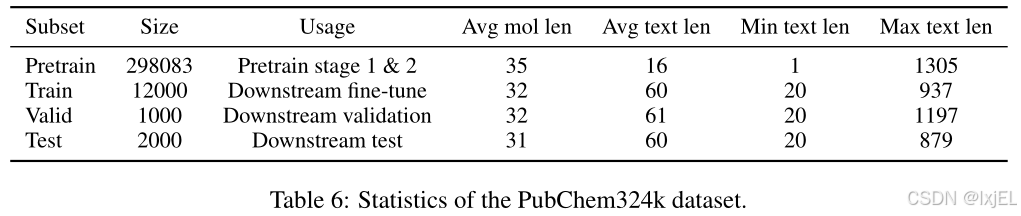
我们的数据处理过程遵循了(Liu等人,2022b)中描述的程序。由于PubChem数据库频繁更新,我们得到的数据集规模更大。对于该网站上的每个分子,我们使用其网页中的“description”字段作为相应的文本描述。为了避免信息泄露,我们将文本开头的任何通用名称或IUPAC名称替换为文本模板(例如,“The molecule”)。PubChem324k的详细统计数据如Table 6所示。
C More Experimental Results
Molecule-Text Retrieval

在这里,我们展示了MolCA在PubChem324k、PCDes和MoMu数据集上的完整的分子-文本检索性能。遵循(Su等人,2022年)的做法,我们报告了在64个随机样本批次中的检索性能以及在整个测试集上的检索性能。正如Table 7所示,我们的结论与第4.4节的结论一致:1)MolCA在分子-文本检索任务中持续优于基线模型;2)应用MTM模块进行重新排序对于MolCA的分子-文本检索性能至关重要。
Ablating the Pretrain Stages
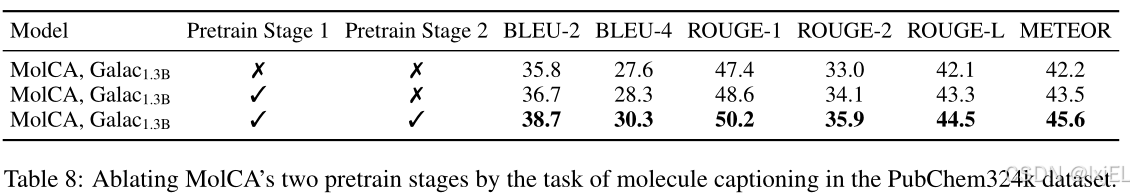
我们对MolCA的两个预训练阶段进行了消融研究。正如Table 8所示,两个预训练阶段对于MolCA在分子描述任务上的性能都有显著的贡献。
Ablating the Cross-Modal Projector

我们比较了所选的跨模态投影器Q-Former与线性跨模态投影器的性能。对于线性跨模态投影器,我们将图编码器的节点表示通过线性投影器层后输入到基础语言模型中。我们调整了图编码器、线性投影器以及基础语言模型的LoRA适配器的权重。实验设置和超参数与MolCA相同。Table 9显示了结果。我们可以观察到:
1)线性跨模态投影器的性能不如Q-Former。我们推测线性层在桥接2D分子和1D文本之间的模态差距方面不是最优的。这与MME基准测试中的发现一致,其中基于Q-Former的方法、MiniGPT-4优于基于线性跨模态投影器的方法;
2)线性跨模态投影器略微优于仅使用SMILES的基线,我们认为这种改进是由于使用了2D分子图,但由于线性投影器的效果有限,所以收益有限。
MolCA’s Generation Results

Figure 7显示了MolCA的分子到文本生成结果。Table 10也给出了分子标注的两个样本。具体来说,我们比较了MolCA(即1D SMILES + 2D Graph)及其变体,该变体仅使用1D SMILES进行预训练和微调。我们可以观察到,使用1D和2D图可以更准确地描述分子结构。

Computational Cost
我们在Table 11中给出了MolCA三个训练阶段的真实训练时间。所有实验均在两台NVIDIA A100 40 GB gpu上进行。值得注意的是,我们观察到,就计算资源而言,微调阶段是可以承受的。


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








