1. read.table()
read.table(file, header = FALSE, sep = "", quote = "\"'",
dec = ".", numerals = c("allow.loss", "warn.loss", "no.loss"),
row.names, col.names, as.is = !stringsAsFactors,
na.strings = "NA", colClasses = NA, nrows = -1,
skip = 0, check.names = TRUE, fill = !blank.lines.skip,
strip.white = FALSE, blank.lines.skip = TRUE,
comment.char = "#",
allowEscapes = FALSE, flush = FALSE,
stringsAsFactors = default.stringsAsFactors(),
fileEncoding = "", encoding = "unknown", text, skipNul = FALSE)
- file 指定读入的文件
- header 是否有列名(默认无)
- seq 指定分隔符(空格、TAB、换行符、回车符)
- quote 制定包围字符型数据的字符。默认情况下,字符串可以被 " 或 ’ 括起,并且两种情况下,引号内部的字符都作为字符串的一部分。有效的引用字符(可能没有)的设置由参数 quote 控制。默认值改为 quote = “”
- dec = “.” 指定小数点数
- colClasses 指定列的数据类型格式
- row.names 指定各行名称,也可以是数字,指定某列为行名col.names
- as.is = !stringsAsFactors as.is 字符向量是否转换成因子(仅仅这个功能),TRUE时保留为字符型
- na.strings = “NA” 指定什么样的字符表示值缺少
- colClasses = NA colClasses运行为输入中的每个列设置需要的类型。注意,colClasses 和 as.is 对每 列专用,而不是每个变量。因此,它对行标签列也同样适用(如果有的话)。
- nrows = -1 最大读入行数,即读入前多少行,“-1”表示都读入
- skip = 0 跳过文件的前n行(skip = n)
- check.names = TRUE # 检查变量名在R中是否有效
- fill = !blank.lines.skip 从一个电子表格中导出的文件通常会把拖尾的空字段(包括?堑姆指舴? 忽略掉。为了读取这样的文件,必须设置参数 fill = TRUE
- strip.white = FALSE 如果设定了分隔符,字符字段起始和收尾处的空白会作为字段一部分看待的。为了去掉这些空白,可以使用参数 strip.white = TRUE
- blank.lines.skip = TRUE 默认情况下,read.table 忽略空白行。这可以通过设置 blank.lines.skip = FALSE 来改变。但这个参数只有在和 fill = TRUE 共同使用时才有效。这时,可能是用空白行表明规则数据中的缺损样本。
- comment.char = “#” 默认情况下,read.table 用 # 作为注释标识字符。如果碰到该字符(除了在被引用的字符串内),该行中随后的内容将会被忽略。只含有空白和注释的行被当作空白行。如果确认数据文件中没有注释内容,用 comment.char = “” 会比较安全 (也可能让速度比较快)。
- allowEscapes = FALSEread.table 和 scan 都有一个逻辑参数 allowEscapes。从 R 2.2.0 开始,该参数默认为否,而且反斜杠是唯一被解释为逃逸引用符的字符(在前面描述的环境中)。如果该参数设为是,以C形式的逃逸规则解释,也就是控制符如 , , , , , , 八进制和十六进制如 40 和 x2A 一样描述。任何其它逃逸字符都看着是自己,包括反斜杠
header参数:逻辑值。数据文件中是否有标题行。如果header设置为TRUE,则要求第一行要比数据列的数量少一列。
col.names:指定列名的向量。缺省情况下是又"V"加上列序构成,即V1,V2,V3......col.names=c("year", "x", "y")
nrows:整型数。用于指定从文件中读取的最大行数。负数或其它无效值将会被忽略。
row.names保存行名的向量。可以使用此参数以向量的形式给出每行的实际行名。或者要读取的表中包含行名称的列序号或列名字符串。在数据文件中有行头且首行的字段名比数据列少一个的情况下,数据文件中第1列将被视为行名称。除此情况外,在没有给定row.names参数时,读取的行名将会自动编号。可以使用row.names = NULL强制对行进行编号。
skip:整型数。读取数据时忽略的行数。-从结果可以看出,前10行数据被忽略了。指定skip参数时应该设置col.names参数,否则第一行数据将作为列名处理。
sep参数:数据的分隔符。默认sep=""。默认情况下,read.table()函数以空白作为数据的分隔符。若读取CSV文件,则需要指定sep=","。
blank.lines.skip:逻辑值,此参数值设置为TRUE时,数据文件中的空白行将被忽略。默认值为TRUE。
skipNul:逻辑值。是否忽略空值。默认为FALSE。
2.添加列名
R语言中,无论是什么数据类型,输出时会用1,2,3…等来做行标(默认的),但是作图时,需要有文字来标记图形,例如:
d <- data.frame(name=c("张三","李四","王五"),score=c("22","24","25"))
d
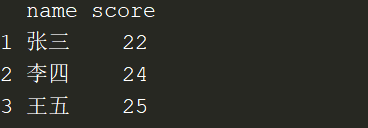
如果想要自己命名列名:
d <- data.frame(name=c("张三","李四","王五"),score=c("22","24","25"))
rownames(d) <- c("person1","person2","person3")
d

3. 找了一个简单的数据进行画热图
1. 自己编辑数据的方式:
txt <- "ID;Zygote;2_cell;4_cell;8_cell
Gene_1;1;2;3;4
Gene_2;6;5;4;5
Gene_3;0.6;0.5;0.4;0.4"
data2 <- read.table(text=txt, sep=";",row.names=1, header=TRUE, quote="", check.names = F)
head(data2)
注:
读入时,增加一个参数
check.names=F也可以解决数字前有一些奇怪的数字
具体的read.tabled的参数的含义:上面的1. read.table();
2. 读取.txt文件的方式:
data3 <- read.table("./test.txt", header=T,row.names=1, quote="", check.names = F)
head(data3)
3. 转换数据格式:
数据读入后,还需要一步格式转换。在使用ggplot2作图时,有一种长表格模式是最为常用的,尤其是数据不规则时,更应该使用。
#需要导入两个包
library(reshape2)
library(ggplot2)
# 转换前,先增加一列ID列,保存行名字
data3$ID <- rownames(data3)
#也就是给你的每一行的名字有一个统称`ID`,也就是纵坐标的标题
#这个是可以随意起名字的
# melt:把正常矩阵转换为长表格模式的函数。工作原理是把全部的非id列的数值列转为1列,命名为value;所有字符列转为variable列。
# id.vars 列用于指定哪些列为id列;这些列不会被merge,会保留为完整一列。
data_m <- melt(data3, id.vars=c("ID"))
#看一下效果
head(data_m)
4. 分解绘图
数据转换后就可以画图了,分解命令如下:
p <- ggplot(data_m, aes(x=variable,y=ID))
# data_m: 是前面费了九牛二虎之力得到的数据表
# aes: aesthetic的缩写,一般指定整体的X轴、Y轴、颜色、形状、大小等
# 在最开始读入数据时,一般只指定x和y,其它后续指定
p <- p + geom_tile(aes(fill=value))
# 热图就是一堆方块根据其值赋予不同的颜色,所以这里使用fill=value, 用数值做填充色。
p
# ggplot2为图层绘制,一层层添加,存储在p中,在输出p的内容时才会出图。
#有时候会出现横轴重叠的情况:
#解决方法就是:一个办法是调整图像的宽度,另一个是旋转横轴标记
theme: 是处理图美观的一个函数,可以调整横纵轴label的选择、图例的位置等
注:
这里选择X轴标签45度。
hjust和vjust调整标签的相对位置,具体见图
简单说,hjust是水平的对齐方式,0为左,1为右,0.5居中,0-1之间可以取任意值。vjust是垂直对齐方式,0底对齐,1为顶对齐,0.5居中,0-1之间可以取任意值
p <- p + theme(axis.text.x=element_text(angle=45, hjust=1, vjust=1))
p
5.读取xlsx文件的时候怎么不让标题出现乱码
无论是用
read.table还是用read.xlsx来读取文件的时候,有时候标题是数字;那么就会遇到输出的时候数字前面或者后面有字母加上:
其实我只想要2,3,4,5,6,7,8,9,10这几个数字的
解决方法就是需要在读取的时候加上check.names=F参数
例:data <- read.xlsx("file.xlsx",row.names = 1,sheetIndex = 1, check.names=F)
6.关于颜色设置,比较全的一个链接:教程链接
7.使用pheatmap来处理数据画热图:
参考链接
链接一:http://www.360doc.com/content/19/0331/18/47588191_825528353.shtml
链接二:https://www.jianshu.com/p/1c55ea64ff3f
链接三(这个写的非常的全面):https://blog.csdn.net/lalaxumelala/article/details/86022722
链接四:https://www.sohu.com/a/129632199_278730
链接五:https://blog.csdn.net/sinat_38163598/article/details/72770404
关于怎么读取xlsx文件:
1.首先要导入
openxlsx包:library(openxlsx)
2.调用read.xlsx函数:
setwd("D:/桌面/")
data <- read.xlsx ("docking_power-binding funnel analysis.xlsx", rowNames = T, check.names = F)
关于读取数据后每一列的前面都会有数字的原因:
如果你没有指定行名(第一列,每一行开始的地方)的话,他就会默认(rowNames = F),就会以数字的形式自动给你补行名。
需要设置rowNames = T,他就会把我们数据中的第一列作为行名显示。

关于颜色中字母和RGB匹配表
参考:https://www.toolbaba.cn/d/ct_color_card
两种代码:
library(pheatmap)
library(openxlsx)
setwd("D:/桌面/")
data <- read.xlsx ("docking_power-binding funnel analysis.xlsx",
rowNames = T, check.names = F)
head(data)
#1.这样就可以输出一张热图
#pheatmap(data)
pheatmap(
#2.一般情况下,我们需要相对基因进行标准化,参数对行进行归一化
data,scale = "row",
#3.列的顺序是递增的序列,不应该对列进行聚类--这样得到的就是正常的顺序了--测试一下
cluster_rows = F, cluster_cols = F,
#4.修改一下配色
color = colorRampPalette(c("Brown", "Turquoise1", "DarkMagenta"))(50),
#5.legend_breaks参数设定图例显示范围,legend_labels参数添加图例标签(注:只能有五个标签) + border_color 表示热图上单元格边框的颜色,如果不绘制边框,则使用NA
#legend = F,
#legend_breaks = -1:4, legend_labels = c("0","1e-4", "1e-3", "1e-2", "1e-1", "1"),
#legend_breaks = c(-2:2), legend_labels = c("0.200", "0.400", "0.600","0.800","1.000"),
#6.去掉每个方格时间的边框
border_color = NA,
#7.在热图的单位格中显示数值
#display_numbers=T,
#8.不显示dendrogram,在聚类之后去掉系统树(和第3条(直接就不聚类,也就没有树)一致,当没有聚类的时候,就没有树了,也就是无需这一步了)
#treeheight_row = 0, treeheight_col = 0,
#9.根据行列将数据分隔开(同时要有聚类才可以,也就是需要关闭第3条)
#cutree_cols=2, cutree_rows=1,
#10.main可设置热图的标题
#main = "RMSD",
#11.filename可直接将热图存出,支持格式png, pdf, tiff, bmp, jpeg,并且可以通过width, height设置图片的大小;
#cellwidth = 15, cellheight = 12, main = "Example heatmap", fontsize = 8, filename = "test.pdf",
)
library("lattice")
library(latticeExtra)
library(openxlsx)
setwd("D:/桌面/")
data <- read.xlsx ("docking_power-binding funnel analysis.xlsx",
rowNames = T, check.names = F)
head(data)
#hc <- hclust(dist(data)) #按行聚类
#dd.row <- as.dendrogram(hc)#保存行聚类树形
#row.ord <- order.dendrogram(dd.row) #保存行聚类顺序
#hc <- hclust(dist(t(data))) #按列聚类
#dd.col <- as.dendrogram(hc) #保存列聚类树形
#col.rod <- order.dendrogram(dd.col) #保存列聚类顺序
#data1 <- data[row.ord,] #只对行聚类(是否对行、列聚类)
levelplot(t(data),aspect="fill",
#图例设置
colorkey=list(space="right",width=1.5), xlab="RMSD",ylab="Software",
legend=list(
#对于右边的树
#right=list(fun=dendrogramGrob,args=list(x=dd.row,rod=row.ord,side='right',size=5)),
#x轴标签旋转60度
scales=list(x=list(rot=60))
),
col.regions = gray(0:100/100),
)








 本文详细介绍了R语言中read.table函数的参数及其用途,包括文件读取、列名设置、数据类型转换等。此外,还讲解了如何自定义列名、处理数据格式以及绘制热图的方法,包括ggplot2库的使用和pheatmap函数的应用。通过实例展示了数据读取、格式转换、热图颜色配置等关键步骤,为R语言的数据分析和可视化提供了实用指南。
本文详细介绍了R语言中read.table函数的参数及其用途,包括文件读取、列名设置、数据类型转换等。此外,还讲解了如何自定义列名、处理数据格式以及绘制热图的方法,包括ggplot2库的使用和pheatmap函数的应用。通过实例展示了数据读取、格式转换、热图颜色配置等关键步骤,为R语言的数据分析和可视化提供了实用指南。

















 7907
7907

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








