Prompt-Guided Injection of Conformation to Pre-trained Protein Model
Summary
针对目前蛋白质预训练模型生成的embedding固定的问题,提出了一种解决思路:在预训练中引入prompt learning,可以针对不同的下游任务选择不同的prompt,将相关知识注入到模型中以便产生多变的embedding
Background
给定一个氨基酸序列,除非蛋白质变性,否则一级结构和二级结构(无序区域除外)将被确定。由于大分子不是刚性的,蛋白质结构可以响应各种生物过程而发生可逆的变化。同一蛋白质的不同结构称为不同构象,它们之间的转变称为构象变化,各种3D构象是从其结构预测蛋白质功能的主要障碍。
native conformation(NC)指蛋白质自然折叠成的3D结构,
interaction conformation(IC)指蛋白质在实现其生物功能时折叠成与其他蛋白质相互作用的对应结构。
蛋白质预训练模型用一个固定的embedding表示一个蛋白质,无法胜任多样化的任务
Significance
解决了固定的蛋白质嵌入难以获得其他知识的问题
Method
使用预训练模型训练相关提示,并将prompt与蛋白质embedding拼接,再经过transformer获得增强后的蛋白质表示
Results
- PTPM可以从不同的prompt中获取不同的知识,进而生成具有不同知识的嵌入,在需要特定知识的蛋白质相关任务中表现不错
- prompt对于模型来说不一定都是有益的,它可能对某些任务是有害的。需要针对不同的任务选择合适的prompt
- prompt可以作为“知识探针”(可以先在具体任务上注入某些prompt,然后根据不同prompt的表现来确定该任务需要哪一种知识)
Limitations
怎么选择合适的prompt对于模型来说可能是一个问题
Questions
-
为了促进正交性和泛化性,提示之间的信息流也被禁止
-
正交性指的是模型的不同部分之间相互独立,这样可以避免在训练过程中出现冗余信息,不会假设输入中的特征之间有任何相互作用,避免模型偏向于某些特定的输入形式,从而提高模型的泛化能力。
-
泛化性指的是模型能够很好地推广到未知的数据上,这样能够更好地应对实际应用中的变化。
阻止提示之间的信息流也有助于防止过拟合,从而提高模型的鲁棒性。
-
详细介绍
-
背景:
蛋白质预训练模型用一个固定的embedding表示一个蛋白质,因此无法胜任多样化的任务
为了使PTPMs产生信息表示,学习可解释、可插入和可扩展的蛋白质提示,将相关知识注入PTPMs
使用掩码语言建模任务的PTPMs优化被解释为学习序列提示(Seq prompt),使PTPM能够捕获氨基酸之间的顺序依赖性。
为了将构象知识结合到PTPM中,我们提出了一种相互作用**构象**提示( interaction-conformation prompt, IC prompt),它通过与蛋白质-蛋白质相互作用任务的反向传播来学习
提出构象感知预训练蛋白质模型,在多任务设置中学习序列和相互作用构象提示
-
贡献:
- 我们建议学习可插入、可解释和可扩展的提示,以将与任务相关的知识注入预训练的蛋白质模型。
- 设计了 ConfProtein 模型,该模型将顺序和构象知识注入到多任务设置中的预训练蛋白质模型中
- 创建了一个新的数据集,其中包含用于接触预测的相互作用构象信息。
- 对蛋白质功能和结构预测任务的综合评估表明,适当的提示可以显着提高预训练模型的性能。
-
本文模型:
将任务相关知识(如构象信息)注入到PTPMs中,以生成更有信息的蛋白质表示
使用prompts避免微调PTLMs,从而提高性能。

ConfProtein 有两个可学习的提示,分别针对蛋白质本身的特性和蛋白质对中的交互构象。
人类仍然不能完全理解生命的语言,即氨基酸序列。因此,基于氨基酸词汇表设计提示语是不可行的。而现有的连续提示是面向任务的,泛化性低。
- 蛋白质本身的属性可以通过氨基酸序列来挖掘,我们利用掩码语言建模(MLM)任务(Devlin et al, 2019)来学习这个提示,称为序列提示(Seq prompt)。
- 对于存在于相互作用对中的构象,利用蛋白质相互作用预测(PPI)任务学习相互作用构象提示(IC提示)。
-
Protein Prompt Learning 参考
传统预训练模型embedding operator: E ( ⋅ ) = E t o k ( ⋅ ) + E s e g ( ⋅ ) + E p o s ( ⋅ ) , t o k e n , s e g m e n t , p o s i t i o n E(·) = E_{tok}(·) + E_{seg}(·) + E_{pos}(·), token, segment, position E(⋅)=Etok(⋅)+Eseg(⋅)+Epos(⋅),token,segment,position
- token embedding 层是要将各个词转换成固定维度的向量。
- segment embeddings 区分一个句子对中的两个句子。Segment Embeddings 层只有两种向量表示。前一个向量是把0赋给第一个句子中的各个token, 后一个向量是把1赋给第二个句子中的各个token。
- position embeddings能够让Bert理解同一个词应该有不同的向量表示
对于Prompt Learning,模型输入有两部分:原始输入序列 S i n S_{in} Sin + 提示 S p t S_{pt} Spt
假设prompt对输入序列 S i n S_{in} Sin施加的影响不受prompt位置的干扰
- S i n S_{in} Sin Embedding: $\mathbf X_{in} = E(S_{in}) $
- S p t S_{pt} Spt Embedding: X p t = E t o k ( S p t ) = E t o k ( s p t 1 ) , . . . , E t o k ( s p t m ) \mathbf X_{pt} = E_{tok}(S_{pt}) = {E_{tok}(s^1_{pt}), . . . , E_{tok}(s^m_{pt})} Xpt=Etok(Spt)=Etok(spt1),...,Etok(sptm)
完整输入: X p r o m p t = X i n ∣ ∣ X p t \mathbf X_{prompt} = \mathbf X_{in} || \mathbf X_{pt} Xprompt=Xin∣∣Xpt
∣ ∣ || ∣∣表示两个向量之间的连接操作
通过transformer中的自注意力机制,整个序列中的每一个token都可以关注到全局信息。
prompt应该为原始输入序列的表示提供任务相关信息,因此只允许从提示到原始输入的单向信息流
为了促进正交性和泛化性,提示之间的信息流也被禁止。
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-XdTChlvW-1670745887829)(images/image-20221119163833360.png)]](https://i-blog.csdnimg.cn/blog_migrate/2f1b4ec707d241bc19b7e117c2a3430b.png)
注意力掩码矩阵M:
m – prompt长度 n – seq长度

输出计算:

损失函数:
-
L C \mathcal L_C LC: knowledge conservation objective L C = L p r ( h ) \mathcal L_C = \mathcal L{pr}(h) LC=Lpr(h) 使预训练的模型保存从预训练任务中学习到的知识
L p r \mathcal L_{pr} Lpr为前一任务的损失函数,eg:预训练期间的 MLM 损失函数
-
L P \mathcal L_P LP: knowledge injection objective L I = ∑ τ ∈ T α τ L τ ( h ) \mathcal L_I = \sum_{\tau \in T} α_τ L_τ (h) LI=∑τ∈TατLτ(h) 指导预训练模型获取新知识
L τ \mathcal L_\tau Lτ是相关任务集合 T T T中某一任务 τ \tau τ的损失
我们假设一种特定类型的知识,如蛋白质构象,可以从多个相关任务中学习,如蛋白质-蛋白质相互作用和结合亲和性预测。
-
L \mathcal L L: $\mathcal L = \mathcal L_C + \lambda \mathcal L_P $
λ \lambda λ是一个超参数,用于平衡新的损失和旧的损失。(balancing the previous and new losses.)
-
ConfProtein
使用预训练MLM任务,在给定的环境中恢复替换的氨基酸,以优化PTPM和序列提示(seq prompt) x S e q x_{Seq} xSeq
通过新任务–预测第p个蛋白和第q个蛋白能否发生相互作用,来学习相互作用构象提示(IC prompt) x I C x_{IC} xIC
-
实验:
-
数据集

-
预训练数据集:
STRING(Szklarczyk et al., 2019),包含用于模型预训练的蛋白质-蛋白质相互作用对。
STRING数据集中的一些相互作用不会形成稳定的构象。为了去除不稳定构象,我们从STRING中选择了物理上唯一的相互作用子集。该子集包含来自14,094个物种的6500万个蛋白质序列和27亿个蛋白质-蛋白质相互作用对。可以定义一个PPI网络,其中一个节点代表一个蛋白质,一条边代表两个相互作用的蛋白质。蛋白质对之间的边缘表明有证据表明它们结合或形成了物理复合体(physical complex)。
-
下游数据集:
SHS27k、SHS148k和STRING Homo Sapiens
Chen等人(2019))基于随机选择的Homo sapiens PPI data创建了SHS27k和SHS148k数据集
STRING-HomoSapiens:使用所有的数据
使用BFS和DFS分别用于划分SHS27k、SHS148k和STRING Homo Sapiens的训练和评估数据集。
利用PPI预测任务来评估IC提示是否可以向PTPM注入构象知识。
在预训练期间,模型会预测两种蛋白质是否可以相互作用;在下游任务上,除了预测相互作用外,模型还会预测两种蛋白质的相互作用类型
-
其他数据集—用于其他任务
-
TAPE是一项旨在评估蛋白质模型普适性的基准。该基准涉及三个主要方面:结构预测、远系同源物检测和蛋白质工程。
-
-
数据集的构建
为了将相互作用构象知识注入到 ConfProtein 中,我们构建了一个 PPI 数据集——一个大规模的物理交互网络。我们使用仅具有物理模式的最新 STRING 数据库,这意味着蛋白质对之间的边表明它们结合或形成物理复合体的证据。该数据库共包含来自 14,094 个物种的 6500 万个蛋白质序列和 27 亿个蛋白质-蛋白质相互作用对。
请注意,来自不同物种的蛋白质之间没有边界。
PPI网络存在分布不均匀的问题,最大的网络包含6万个蛋白质和3.5 × 107条边。这样的数据分布可能导致模型过度关注单一物种的蛋白质。选择具有相当大小的物种网络来预处理数据集。
-
接触图数据
ICProtein 2:接触图的数据集,包含1106个蛋白质复合物,共2212个接触图
ICProtein 2的构建:
- 从Computed structures of core eukaryotic protein complexes中获取所有蛋白质复合物的结构数据
- 使用obabel转换文件格式,计算残差距离,获得基于interaction conformation的接触图
- 上一步得到的接触图为两个相互作用的蛋白质的接触图组成,将之分离
-
预训练
-
实验环境:
Pytorch (Paszke et al., 2019)
Fairseq (Ott et al.,2019)
-
参数设置:
ConfProtein有650M参数,33层,20个注意头。
embedding size = 1280
batch size = 20
learning rate = 0.00001 无权值衰减 固定学习率
氨基酸最大长度为2048
-
baseline
非预训练模型
DPPI (Hashemifar et al., 2018)、 DNN-PPI (H et al.,2018)、 PIPR (Chen et al., 2019)、GNN-PPI (Chen et al., 2019)
前三个基线使用不同的深度学习架构(CNN、RCNN和LSTM)将氨基酸嵌入转换为蛋白质嵌入,并使用线性分类器预测两种蛋白质是否具有相互作用关系。
GNN-PPI利用图形神经网络来关注整个交互图形,并实现SOTA性能。
ProtBert(Elnaggar et al., 2021)、OntoProtein和ESM-1b(Rao等人,2021a)是三种预先训练的模型。
为了公平比较,我们只使用预训练模型来生成氨基酸嵌入,并将这些嵌入输入GNN-PPI
ConfProtein-w/o-IC是我们ConfProtein的变体,仅包括Seq提示,不包括IC提示。
由于ConfProtein的结构与ESM-1b相同,我们通过比较ConfProtein-w/o-IC和ESM-1b来检查Seq提示是否可以保存序列氨基酸信息,以及IC提示是否可以通过比较ConfProtein和ConfProtein-w/o-IC来注入构象知识。
-
-
下游任务定义
- 蛋白质-蛋白质相互作用预测
- 抗体-抗原结合亲和力预测
- 联系预测 Contact Prediction
- 二级结构预测
- 荧光
- Stability
-
-
结果:
- 使用 Seq Prompt不会损害 PTPM 在序列相关任务上的表现,带有Seq提示的PTPMs只能获得氨基酸序列和相关二级结构的知识,
- 结合 IC Prompt可显着提高 PTPM 在交互构象知识重要的任务上的表现,可以有效地获得三维结构的知识。
- 具有恰当的学习提示的PTPMs优于最新的(SOTA)模型,而不恰当的提示将降低其性能。
IC Prompt 可以改善蛋白质预训练模型的PPI预测性能
在最大的STRING Homo Sapiens数据集上,GNN-PPI的性能超过所有PTPM。这是由于内存限制,我们计算氨基酸嵌入序列的平均值作为蛋白质表示。它的性能不如GNN-PPI中的卷积网络。
OntoProtein将知识图中的信息整合到蛋白质表示中,但其性能并不怎么好—>表明并非知识图中的所有信息都对PPI任务有贡献作用,无用的信息对于模型来说可能是有害的。
IC Prompt可以增强具有相互作用构象知识的蛋白质表示
消融实验
数据集:SHS27k + SAbDab
对比: Seq Prompt IC Prompt
预测任务:PPI预测和抗体-抗原结合亲和力预测(SAbDab)

Seq提示在SAbDab数据集中有更大的影响力,在抗体-抗原结合亲和力的预测中,氨基酸的性质是一个关键因素,可以通过Seq prompt得到
IC Prompt在STRING-HomoSapiens数据集中影响力更大,PPI是由蛋白质构象决定的,ConfProtein可以通过IC prompt获得构象知识
解释IC Prompt

a.可视化了有和没有IC提示的TAF4蛋白氨基酸的嵌入
b.一个氨基酸的两次嵌入之间的距离
c.红色标记距离大于100的嵌入对
标记的嵌入都是蛋白质表面的氨基酸,这与与PPI相关的氨基酸几乎都位于蛋白质表面,而不是核心的事实相一致
Prompt对下游任务可能有不利影响
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-o47pUP3J-1670745887848)(images/image-20221121170851567.png)]](https://i-blog.csdnimg.cn/blog_migrate/b0e067ab01c8cced9898f9e9f7b6347e.png)
P@L/2 参考
相同的提示在不同的数据集中对相同的任务产生截然相反的效果,说明使用不适当的知识会产生负面影响。
ConfProtein在长蛋白质中更有效。这是因为较长的蛋白质序列在不同构象的接触图之间差异较大,需要更多的3D信息才能准确预测接触图。
Prompt可以与下游任务无关
利用二级结构预测任务来探索提示如何在不需要它们的任务上执行。
数据集:CB513
MSA Transformer可以利用MSA获得NC
IC提示可以提供具有IC的蛋白质表示

提示可以是未知下游任务的知识探测 Prompts can be knowledge probe for unknown downstream tasks.
由于每个提示在学习后都可以被赋予特定的语义,我们利用提示作为知识探针来确定每个任务所需的信息
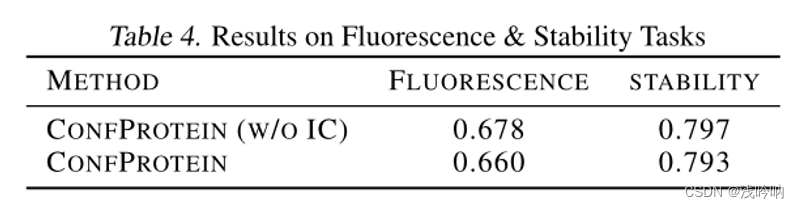
两个蛋白质工程任务:荧光景观预测和稳定性景观预测
- 绿色荧光蛋白在光照下表现出明亮的绿色荧光。
- 荧光景观预测任务的目的是将蛋白质映射到对数荧光强度。

-
蛋白质由氨基酸构成,氨基酸的化学性质决定了蛋白质的整体功能和3D结构
-
预训练语言模型(PTLMs)在语言理解、翻译和对话系统中表现优异,是一种处理序列数据的流行方法。
受到启发,开发了先训练的蛋白质模型(PTPMs),例如:TAPE Transformer、ProtBERT、ESM-1b,来预测蛋白质结构和功能。
在蛋白质二级结构预测、亲和性预测和接触预测等下游任务上表现不错。
存在的问题:蛋白质构象是非常敏感和动态的,受外界因素及其特定功能的显著影响。现有的PTPMs使用固定的embedding来表示一种蛋白质是不合适的。
-
给定一个氨基酸序列,除非蛋白质变性,否则一级结构和二级结构(无序区域除外)将被确定。各种3D构象是从其结构预测蛋白质功能的主要障碍。
同一蛋白质的不同结构称为不同构象,它们之间的转变称为构象变化。
-
构象(conformation):在有机化合物分子中,由C—C单键旋转而产生的原子或基团在空间排列的无数特定的形象称为构象。指一个分子中,不改变共价键结构,仅单键周围的原子放置所产生的空间排布。不同的构象之间可以相互转变,在各种构象形式中,势能最低、最稳定的构象是优势构象。一种构象改变为另一种构象时,不要求共价键的断裂和重新形成。构象改变不会改变分子的光学活性。由氨基酸组成的蛋白质是动态的,可以观察到各种三维结构,称为构象
native conformation(NC)指蛋白质自然折叠成的3D结构,
interaction conformation(IC)指蛋白质在实现其生物功能时折叠成与其他蛋白质相互作用的对应结构。
-
contact map:蛋白质接触图使用二元二维矩阵表示三维蛋白质结构的所有可能的氨基酸残基对之间的距离。参考[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-rM8bVjoH-1670745887855)(C:\Users\WMGray\AppData\Roaming\Typora\typora-user-images\image-20221118162838819.png)]
CDK1的native conformation 和 interaction conformation
-
Prompts:是由人类设计的离散单词序列或通过反向传播学习的连续向量。 -
RSCB术语 参考不对称单元(Asymmetric Unit)是晶体学中的一个概念。定义是:在空间群的对称操作作用下,可以产生出晶胞中全部源自的最少数目的原子或原子团,就叫做不对称单元或不对称单位,也就是晶体学独立单元。也就是说不对称单元可以通过对称操作形成晶胞。我们从PDB数据库上下载的原子坐标文件就是其实是一个不对称单元中的原子数据。
生物学集合体(Biological Assembly)有时候又被称为生物学单元(Biological Unit),从生物学功能上定义的单元。比如有功能的血红蛋是四聚体,这四聚体就称为一个生物学单元。 不对称单元可能包含一个biological assembly;也可能只是biological assembly的一部分;也可能包含多个biological assembly。
-
Prompt learning: 参考Prompt 是一种为了更好的使用预训练语言模型的知识,采用在输入段添加额外的文本的技术。
- 目的:更好挖掘预训练语言模型的能力
- 手段:在输入端添加文本,即重新定义任务(task reformulation)
- prompt construction【Template】
首先我们需要构建一个模版Template,模版的作用是将输入和输出进行重新构造,变成一个新的带有mask slots的文本,具体如下:
-
定义一个模版,包含了2处代填入的slots:[x] 和 [z]
-
将[x] 用输入文本代入
-
answer construction【Verbalizer】
对于我们构造的prompt,我们需要知道我们的预测词和我们的label 之间的关系,并且我们也不可能运行z是任意词,这边我们就需要一个映射函数(mapping function)将输出的词与label进行映射。例如我们的这个例子,输出的label 有两个,一个是😄 ,一个是😟 ,我们可以限定,如果预测词是
fantastic则对应😄,如果是boring则对应😟

-
answer prediction【Prediction】
到了这边我们就只需要选择合适的预训练语言模型,然后进行mask slots [z] 的预测。例如下图,得到了结果
fantastic, 我们需要将其代入[z] 中。

-
answer-label mapping【Mapping】
对于得到的
answer,我们需要使用Verbalizer将其映射回原本的label。

-
Knowledge Graph(知识图谱):知识图谱(Knowledge Graph)主要是用于描述现实世界中的实体(区别于概念,是指客观世界中的具体实物,如张三,李四等)、概念(人们在认识世界过程中形成的对客观事物的概念化表示,如人、动物等)及事件间的客观关系。知识图谱的构建过程即从非结构化数据(图像等)或半结构化数据(网页等)中抽取信息,构建结构化数据(三元组,实体-属性-关系)的过程。参考








 介绍一种新方法,通过引入prompt学习改进蛋白质预训练模型,使其能根据不同的下游任务选择合适prompt,注入相关知识生成多变的embedding。
介绍一种新方法,通过引入prompt学习改进蛋白质预训练模型,使其能根据不同的下游任务选择合适prompt,注入相关知识生成多变的embedding。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








