原文链接HoloView – Tabular Datasets
网格化数据集
import numpy as np
import holoviews as hv
from holoviews import opts
hv.extension('bokeh', 'matplotlib')
在上一个Guide中,我们阐述了如何处理表格化的数据集。
尽管表格数据集非常常见,但许多其他数据集最好由定期采样的n维数组(例如图像、体积数据或更高维的参数空间)表示。在2D屏幕上并使用传统的绘图库,通常很难快速简洁地可视化此类参数空间,但HoloViews允许您快速切片和切割此类数据集,以探索数据并轻松回答有关问题。
网格化
网格数据集通常表示跨多个维度的一些连续变量的观察结果——代表二维表面亮度值的单色图像、三维体积数据、随时间变化的RGB图像序列或任何其他多维参数空间。这种类型的数据在使用空间成像或建模的研究领域中尤其常见,例如气候学、生物学和天文学,但也可以用于表示在多个维度上变化的任何任意数据。
在HoloViews术语中,数据变化的维度是所谓的关键维度(kdims),它定义了底层阵列的坐标,实际值数组由值维度(vdims)描述。像xarray或者iris这些高层的数据类的库允许您使用数组存储坐标,但在这里,我们将自己声明坐标数组,以便更好地理解网格数据接口的工作方式。因此,我们将首先加载一个非常简单的3D阵列:
path = r"C:\Users\samsung\AppData\Local\Programs\Python\Python38\Lib\site-packages\holoviews\examples\getting_started"
import os
os.chdir(path)
data = np.load('../assets/twophoton.npz')
calcium_array = data['Calcium']
calcium_array.shape
这个特定的NumPy数据集包含来自2-photon calcium的数据,该实验提供了通过荧光强度变化编码的神经活动的间接测量。3D阵列表示2D成像平面随时间的活动,在50个时间步长上形成形状为(62,111)的图像序列。如上一篇中一样,我们从HoloView的Dataset开始。然而,为了使HoloViews能够理解原始NumPy数组,我们需要为数据的每个维度(或轴)传递坐标。为了简单起见,这里我们将简单地使用整数坐标表示“Time”、“x”和“y”:
ds = hv.Dataset((np.arange(50), np.arange(111), np.arange(62), calcium_array),
['Time', 'x', 'y'], 'Fluorescence')
ds
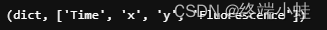
我们建议使用 xarray而不是上面这种手动定义坐标,这使得使用标记的n维数组变得简单。我们甚至可以克隆数据集,并给xarry设置数据类型,将其转化为xarray.Dataset,这是HoloView推荐的处理网格化数据集的格式。在这里,我们可以通过以下方法转换数据格式为 xarray.Dataset,
ds.clone(datatype=['xarray']).data
查看数据
接下来,我们会首先通过.opts()来定制显示的样式
opts.defaults(
opts.GridSpace(shared_xaxis=True, shared_yaxis=True),
opts.Image(cmap='viridis', width=400, height=400),
opts.Labels(text_color='white', text_font_size='8pt', text_align='left', text_baseline='bottom'),
opts.Path(color='white'),
opts.Spread(width=600),
opts.Overlay(show_legend=False))
这个数据集最自然的表现形式可能是在每个时间点显示荧光的图像。使用 .to`接口,我们可以将 Dataset`` 映射到Element的维度上。要显示图像,我们将选取“image”元素,并指定“x”和“y”作为每个图像的两个关键维度。由于我们的数据集只有一个值维度,我们不需要显式声明它:
ds.to(hv.Image, ['x', 'y']).hist()
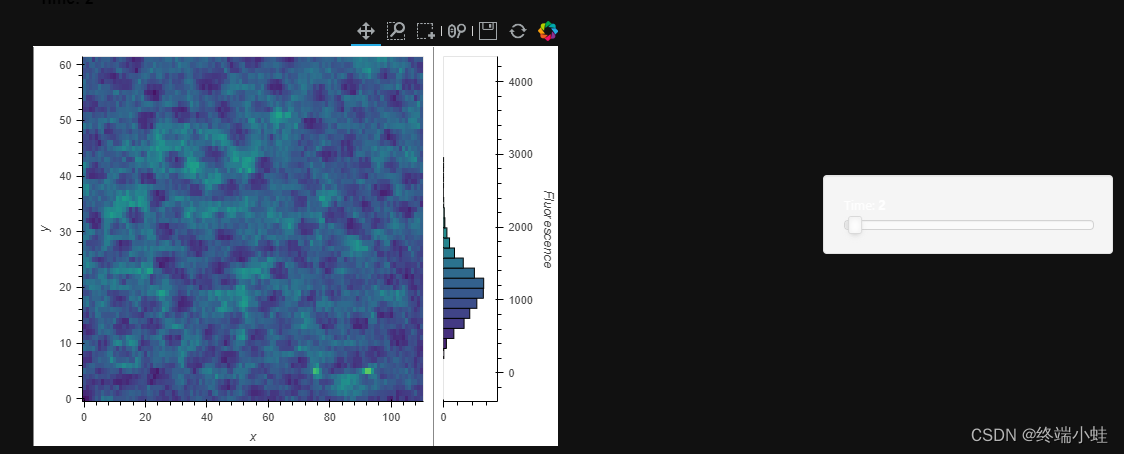
和往常一样,未指定的关键维度"TIME"已成为一个滑块小部件,它允许您每次拖动图像。
一旦选择了一个单独的绘图,您可以通过缩放(这不会给这个特定的降采样数据集带来额外的细节)或通过选择绘图工具栏中的“框选择”工具并在直方图上绘制荧光范围来控制颜色映射范围来与之交互。
在具有许多关键维度或许多不同关键维度值的较大数据集上使用.to或.groupby时,可以使用dynamic=True标志,使您可以动态探索参数空间,而无需提前预计算所有组合。
选择
在处理多维数据集时,我们通常对大参数空间的小区域感兴趣。例如,在处理这样的神经成像数据时,通常会关注较大图像中的感兴趣区域(ROI)。在这里,我们将从之前加载的数据中获取一些边界框。ROI通常是更复杂的多边形,但为了简单起见,我们将使用指定为边界框的左、下、右和上坐标的简单矩形ROI:
ROIs = data['ROIs']
roi_bounds = hv.Path([hv.Bounds(tuple(roi)) for roi in ROIs])
print(ROIs.shape)

在这里,我们有147个ROI,代表数据中147个已识别神经元周围的边界框。为了显示它们,我们将数据包装在“边界”元素中,我们可以将其覆盖在动画的顶部。此外,我们将创建一些“文本”元素来标记每个ROI。最后,我们将使用常规的Python索引语义沿时间维度进行选择,时间维度是第一个关键维度,因此可以简单地指定为“ds[21]”。就像“select”方法一样,这样的索引是按值进行的,而不是按数组索引
labels = hv.Labels([(roi[0], roi[1], i) for i, roi in enumerate(ROIs)])
(ds[21].to(hv.Image, ['x', 'y']) * roi_bounds * labels).relabel('Time: 21')
这里有问题,插入不了图片了
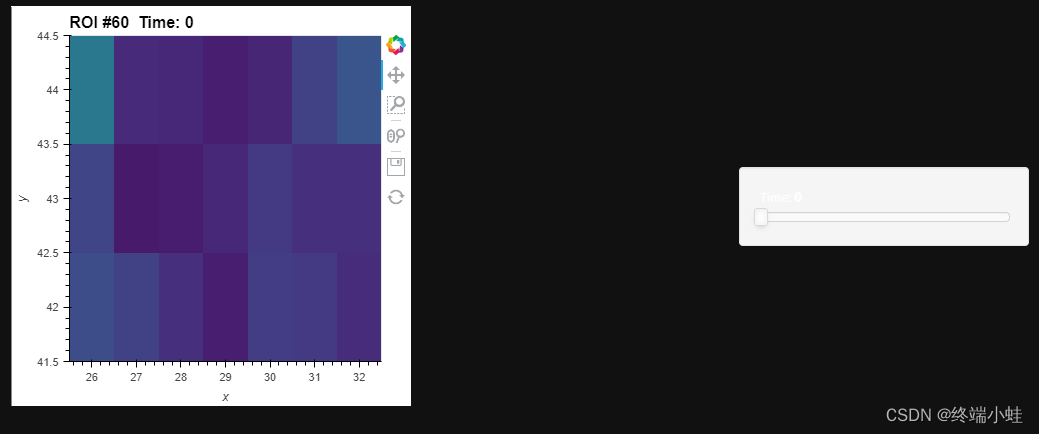
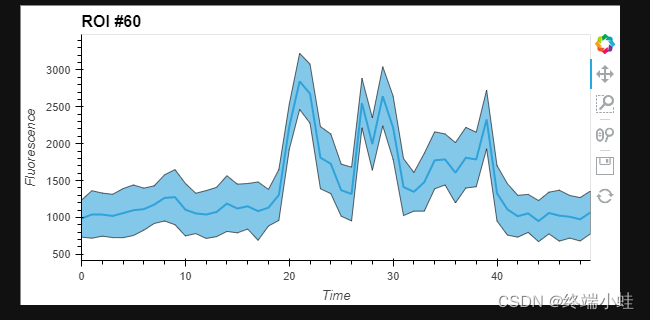
现在我们可以使用这些边界框来选择一些数据,因为它们只是表示坐标。以ROI#60为例,我们可以看到神经元在动画中被强烈激活。使用“选择”方法,我们可以选择ROI的x和y坐标以及看到神经元响应时的大致时间段:
x0, y0, x1, y1 = ROIs[60]
roi = ds.select(x=(x0, x1), y=(y0, y1), time=(250, 280)).relabel('ROI #60')
roi.to(hv.Image, ['x', 'y'])
Faceting
即使我们只选择了很小的一块区域,但还是有非常多的数据。我们可以使用faceting的方法来以不同的方法来显示数据。由于我们现在的数据集中只有几个像素,例如,我们可以绘制ROI中每个像素的荧光随时间的变化。我们可以使用.to接口来显示Curve类型,使用Time当作kdims。
roi.to(hv.Curve, 'Time').grid()
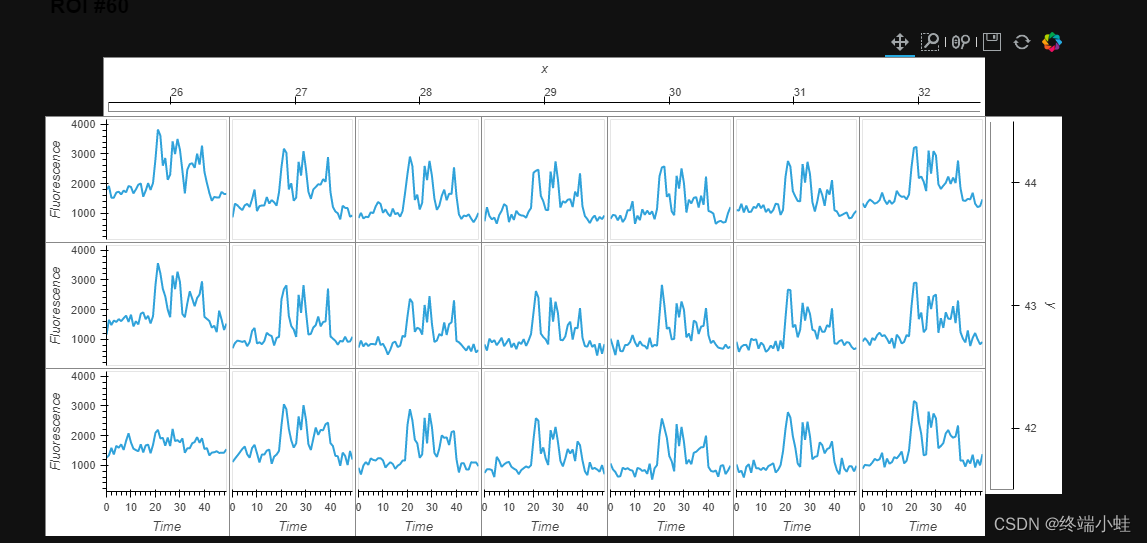
上面的单元格和上一个单元格显示相同的数据,但根据数据映射到屏幕上的方式,显示方式非常不同。
聚合
我们不需要为每个像素单独生成一条曲线,而是需要对x和y方向的数据进行平均,以获得该神经元活动的聚合估计。为此,我们可以使用聚合方法获得ROI窗口内的平均信号。使用spreadfn,我们还可以计算像素之间的标准差,这有助于我们了解信号在该窗口中的变化程度(让我们知道在聚合时掩盖了什么)。我们将平均值和标准差数据显示为“Spread”和“Curve”元素的叠加:
agg = roi.aggregate('Time', np.mean, spreadfn=np.std)
hv.Spread(agg) * hv.Curve(agg)```
当然,我们可以结合所有这些方法并聚合每个ROI,逐个ROI对整个数据集进行分面,以显示各种神经元的活动如何不同。























 747
747











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








