H-DenseUNet: Hybrid Densely Connected UNet for Liver and Tumor Segmentation from CT Volumes
这篇论文发表于2018年的IEEE Transactions on Medical Imaging,来自香港中文大学团队。
论文亮点
摘要
精准而智能的肝区域和肝肿瘤分割方法在临床实践中是高度需要的,它可以帮助医生进行肝癌诊断与治疗计划的确定。在最近的方法中,2D和3D FCN方法充当了立体图形分割的支柱。然而,2D卷积无法利用第三个维度的空间信息,同时,3D卷积则存在高昂的计算代价和GPU内存消耗的问题。为了解决这些问题,我们提出了一种新颖的混合密集连接的U-Net模型(H-DenseUNet),它由一个用于有效提取slice之间的特征的2D DenseUNet模型和一个基于肝脏肿瘤分割的自动上下文算法的分层融合三维上下文的3D DenseUNet。我们将H-DenseUNet的学习过程以一种端到端的方式公式化,其中切片之间的表征和切片内部的特征合一通过一个混合特征融合层联合优化。我们在MICCAI2017的肝分割挑战赛数据集和3DIRCADb数据集上进行了广泛地验证。我们的方法在肿瘤的分割结果上超越了其他SOAT方法,并且甚至在单个模型上,也取得了非常具有竞争力的表现。
介绍
从CT图像中对肝癌做出准确诊断是十分重要的。传统上,肝区域和肝病变区域都是通过医生一个切片一个切片地进行描述的,然而这不仅耗时,而且受到医生主观因素的影响。因此,自动的肝区域分割和肝病变(肿瘤)区域分割在临床应用中是十分必要的。
从CT图像中准确地进行分割出肝区域是非常困难的,而分割出肝肿瘤区域就更困难了。
为了解决这些困难,已经提出了许多分割方法,包括灰度阈值、区域增长和可变模型等。然而这些传统方法依赖于手工制作的特征,并且特征表达能力有限。最近,全卷积神经网络在识别问题上取得了很大的成功。许多研究者在肝区域分割和肝肿瘤分割上使用了深度学习方法,这些方法大概可以分为两类:
【1】2D FCNs,例如U-Net结构,多通道的FCN等;
【2】3D FCNs,用3D卷积代替2D卷积,并且以三维数据作为输入。
在临床诊断中,有经验的医生通常会根据z轴上的相邻切片来观察和分割肿瘤。然而,基于2D FCN的方法忽视了z轴上的上下文信息,这会导致分割精度的下降。具体来说,有些方法使用单张或者三张相邻的slice作为2D FCN的输入,随后将得到的2D 分割图堆叠成三维分割结果。尽管采取三张相邻slices作为输入的方法利用了相邻切片的信息,但是它们仍然不足以提取空间维度上的空间信息,这会使分割结果下降。为了解决这个问题,一些研究者也提出了使用多个平面切片的方法、以及RNN的方法来提取3D信息。相比于2D FCN,3D FCN的计算代价和GPU内存消耗过高。过高的GPU内存消耗也限制了网络的深度和滤波器的大小,而这二者是网络性能的关键。3D卷积沉重的计算代价也妨碍了它应用在大尺度的数据集上。
为了解决上述问题,我们提出了一种新颖的端到端系统,称之为混合密集连接型U-Net(H-DenseUNet),其中intra-slice(切片之间的)特征和3D 上下文信息都很有效地进行了提取,并且进行了联合优化以实现准确的肝区域和病变区域分割。我们的H-DenseUNet有以下两个技术上的成就:
深而高效的网络:首先,为了完全地提取切片间的高层特征,我们设计了一个非常深而高效的网络,称之为2D DenseUNet,将密集连接路径和UNet连接的优势进行融合。密集连接路径来源于DenseNet,它解决了训练非常深的网络的困难。不同于DenseNet,我们增加了UNet连接,即在我们架构编码路径和解码路径之间的大范围跳跃连接。因此,网络可以保留低层空间特征来更好地探索切片之间的上下文信息。
混合特征探索:第二,为了探索三维特征表达,我们设计了一种端到端的训练系统,称之为H-DenseUNet,其中切片间和切片内的特征都被有效地提取,并且联合地通过混合特征融合层(HFF)进行优化。具体而言,3D DenseUNet是由2D DenseUNet通过自动上下文机制组合而成的。有了来自2D DenseUNet的语义概率的指导,在3D DenseUNet上的优化扰动被很好地消除,这有利于3D 上下文信息提取。此外,在端到端系统下,由三维特征和有代表性的高层切片间特征组成的混合特征可以自动地融合,并一起进行优化以更好地进行肝和肿瘤识别。总的来说,这项工作有以下成就:
【1】我们设计了一个DenseUNet来有效地提取切片间的分层特征以进行肝和肿瘤分割,其中对密集连接路径和UNet连接进行了融合来提升肝肿瘤的分割性能。
【2】我们提出了一种H-DenseUNet框架来探索对于肝区域分割和肿瘤分割的混合特征(切片间特征和切片内特征)。这个混合特征学习框架很好地回避了2D网络会忽略三维上下文信息和3D网络存在很大计算代价的问题,并且可以作为一种新的探索3D上下文的新范例。
【3】我们的方法取得了极好的结果。
方法
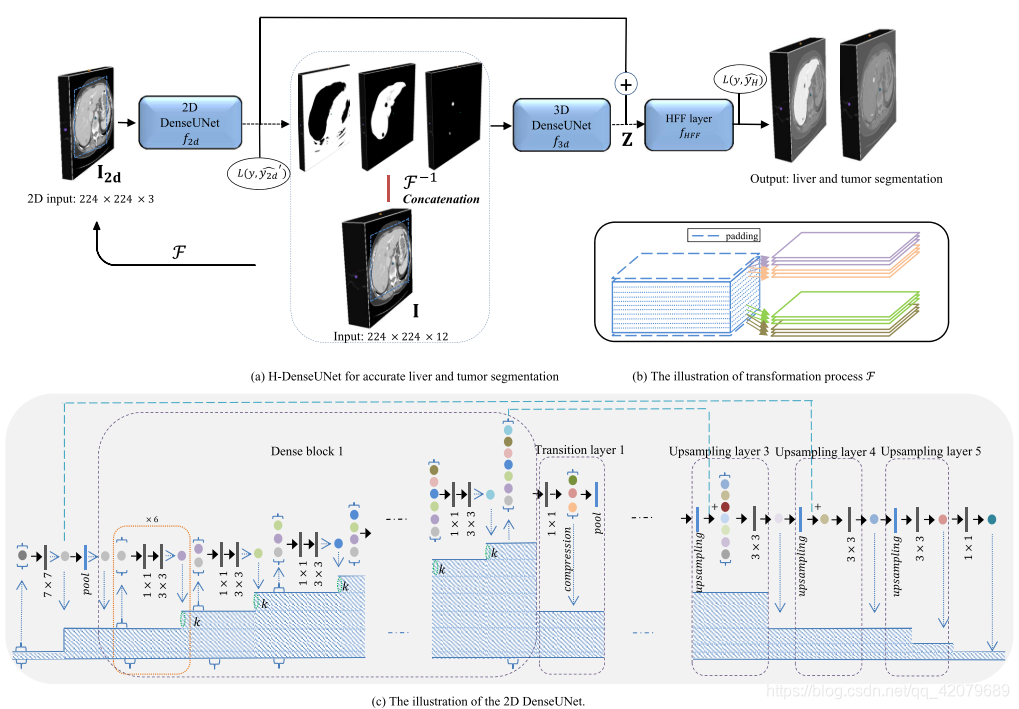
上图展示了我们提出的肝区域和肝病变分割方法的流程。我们采用了级联学习策略来减少总的运算时间,它已经在许多识别任务中使用。首先,一个简单的ResNet框架被训练为获取一个快速但粗略的肝分割(注意,这里未在图中画出!!)。在ResNet提取ROI区域后,我们提出的H-DenseUNet可以通过一个2D DenseUNet 和3D DenseUNet,有效地探索切片内和切片间的特征,并通过混合特征融合层联合地对混合特征进行优化来实现精确肝区域和病变区域分割。
用于切片间特征提取的深度2D DenseUNet
切片间的特征提取路径遵循了DenseNet-161(即DenseNet论文提出的结构)的结构,它由重复的带有不同输出维度的密集连接块组成。在每一个密集连接块中,从任何一层到后面的子层中都有直接的连接,如上图(c)中所示。每一层都能产生k个特征图,k被称之为增长率。层之间的密集连接的一个好处在于它相比传统网络有更少的输出维度,避免学习大量的特征。此外,密集连接路径还能确保层之间的最大信息流动,这可以提升梯度的流动性,并且因此缓解在一个非常深的神经网络搜索全局最优时的扰动。
然而,原始的DenseNet-161用于目标分类任务。此外,由于分割任务的FCN网络常常需要采取池化或上采样操作,这可能会导致低层特征(即高分辨率)的信息丢失。综合这两种考虑,我们提出了一种2D DenseUNet,其中结合了密集连接路径和UNet跳跃连接路径的优点。具体来说,层之间的密集连接应用在每个小块中,来确保最大信息流动。同时,UNet在编码器和解码器之间的大范围跳跃连接用于保留低层信息。
用 I ∈ R n × 224 × 224 × 12 × 1 \mathbf{I} \in R^{n \times 224 \times 224 \times 12 \times 1} I∈Rn×224×224×12×1表示输入训练样本(对应于 224 × 224








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 3718
3718











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








