小罗碎碎念
今日推文主题:人工智能在影像组学/病理组学领域中的最新进展
今天的推文比较有意思,研究影像组学的去看第二、三篇文献,研究病理组学的看第一、四、五篇文献,研究基因组学的,去看第六篇文献。
昨天和一个约翰霍普金斯大学的小伙伴短暂交流了一下,他说他们课题组是做小儿肿瘤的,好家伙,又涉及我的知识盲区了,所以,今天准备的第三、六篇文献都是与小儿肿瘤相关的。对于尝试做多模态的老师/同学来说,这期推文就是为你们量身定制的,能让你们从不同的角度去验证你们的部分想法。(想表示感谢的话,关注转发点赞就行,哈哈)
第一篇文献我也想再提一嘴,老粉应该清楚,之前小罗推荐过生成虚拟单模态数据的推文,但是那是根据转录组数据生成病理切片,哎嘿嘿,你看人家,直接倒过来,发一篇Nature Cancer,23.5分到手。
我是小罗同学,如果想要以最短时间,掌握医学AI领域最新的进展以及一线大佬的动态,建议关注我,哈哈,回见!!

一、ENLIGHT-DeepPT:通过模拟的转录组学数据预测癌症治疗的反应

一作&通讯
| 角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Danh-Tai Hoang | 澳大利亚国立大学生物数据科学研究所,科学学院 |
| 通讯作者1 | Eric A. Stone | 美国国立卫生研究院国家癌症研究所癌症数据科学实验室 |
| 通讯作者2 | Eytan Ruppin | 美国国立卫生研究院国家癌症研究所癌症数据科学实验室 |
文献概述
这篇文章介绍了一种名为ENLIGHT-DeepPT的深度学习框架,该框架能够通过模拟的转录组学数据预测癌症治疗的反应。
ENLIGHT-DeepPT方法包括两个步骤:
- 首先是DeepPT(一个深度学习模型),可以从染色的肿瘤切片预测全基因组肿瘤mRNA表达;
- 其次是ENLIGHT,利用推断出的表达值预测对靶向和免疫疗法的反应。
研究结果表明,DeepPT在16个癌症基因组图谱(The Cancer Genome Atlas, TCGA)的队列测试中成功预测了转录组学,并且能够很好地泛化到两个独立的数据集。ENLIGHT-DeepPT能够成功预测五个独立患者队列中的真实反应者,这些患者队列涉及六种癌症类型的四种不同治疗,总体上,与基线反应率相比,预测反应者的反应率提高了39.5%,整体比值比(odds ratio, OR)为2.28。
此外,该研究还探讨了DeepPT预测的基因表达与肿瘤免疫细胞浸润水平之间的相关性,发现预测的免疫相关基因表达与肿瘤浸润性淋巴细胞(TILs)的数量高度相关。研究还发现,DeepPT推断出的预后特征水平与患者生存相关,且与实际表达值相比,预测值在某些癌症类型中显示出更高的风险比(hazard ratios, HRs)。
研究强调,ENLIGHT-DeepPT的预测准确性在没有接受治疗数据训练的情况下,与直接从图像预测反应的准确性相当。这项工作为使用常规的H&E染色切片预测临床反应提供了一种新的方法,对于快速提供治疗选项具有潜在的临床应用价值。
重点关注
Fig. 1 展示了研究的总体设计和方法,分为三个主要部分:

a. DeepPT 架构的三个主要组成部分:
- 预训练的ResNet50卷积神经网络(CNN)单元:位于左侧,负责从图像瓦片中提取组织病理学特征。
- 自动编码器:位于中间,将2048个特征压缩到较低维度的512个特征,以减少噪声和计算需求,同时避免过拟合。
- 多层感知器(MLP):位于右侧,将这些组织病理学特征整合起来,预测样本的基因表达。
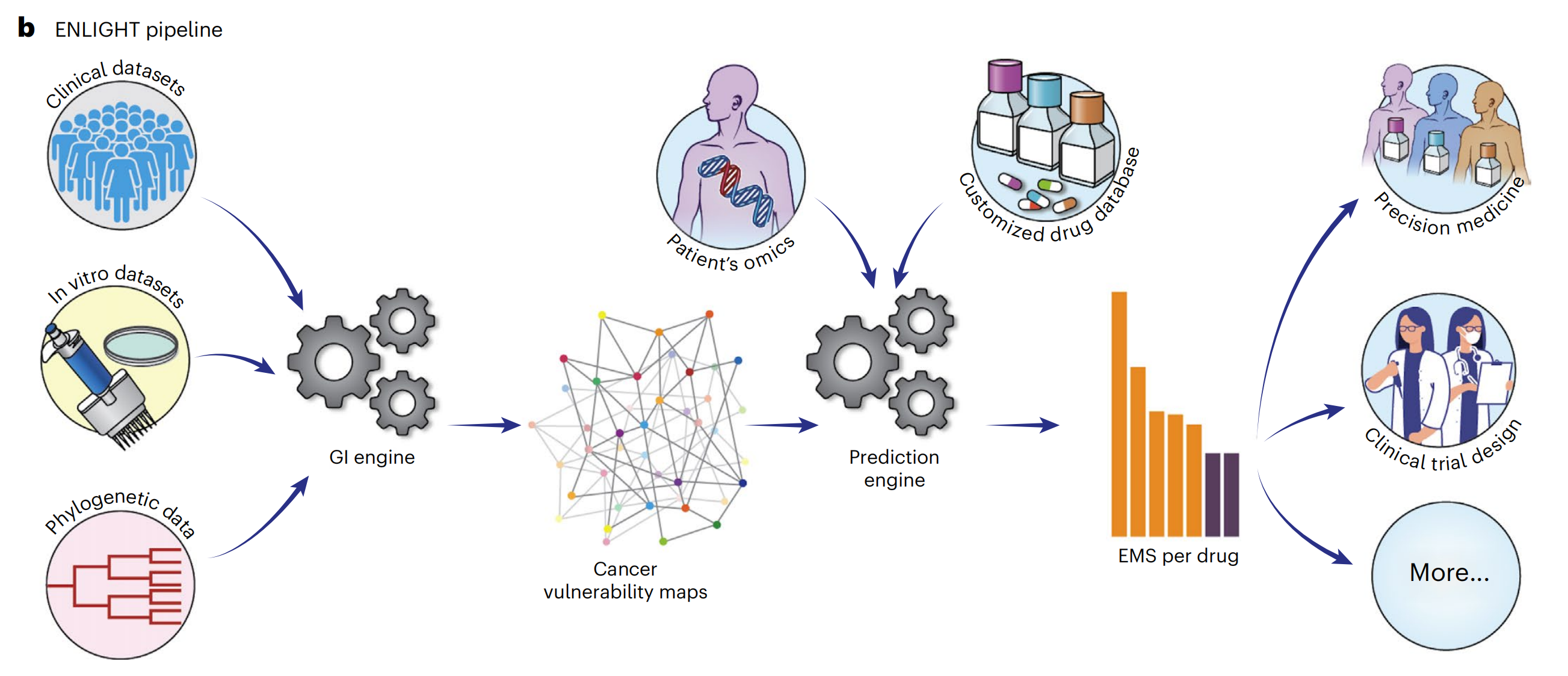
b. ENLIGHT 流程概述:
- ENLIGHT 首先从各种癌症体外和临床数据源推断出给定药物的基因交互(GI)伙伴。
- 给定患者的合成致死(SL)和合成救援(SR)伙伴以及转录组学数据,ENLIGHT 计算药物匹配得分,该得分用于预测患者对治疗的反应。
- 在本研究中,ENLIGHT 使用 DeepPT 预测的表达数据来为每个研究的患者生成药物匹配得分。

c. 使用 DeepPT 和 ENLIGHT 的分析概述:
- 顶部行:DeepPT 使用来自 TCGA 的多种不同癌症类型的 FFPE 切片图像及其匹配的转录组学数据进行训练。
- 中间行:训练阶段之后,模型被应用于预测内部(保留的)TCGA 数据集以及两个它们未经过训练的外部数据集上的基因表达。
- 底部行:在五个独立的测试临床数据集中,预测的肿瘤转录组学作为输入提供给 ENLIGHT,以预测患者对治疗的反应并评估整体预测准确性。
这种设计允许研究者通过深度学习模型从组织病理学图像预测基因表达,并进一步预测患者对特定癌症治疗的反应。通过这种方法,研究者可以在没有直接的治疗反应数据的情况下,为广泛的癌症类型和治疗提供预测。
二、在结直肠息肉的光学诊断中,人工智能(AI)辅助诊断(CADx)系统与人类内镜医师之间的信任度研究

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Quirine E.W. van der Zander | 荷兰马斯特里赫特大学医学中心胃肠病学和肝病学系 |
| 通讯作者一 | Chris C.P. Snijders | 荷兰埃因霍温理工大学人类技术互动系 |
| 通讯作者二 | Erik J. Schoon | 荷兰马斯特里赫特大学医学中心胃肠病学和肝病学系以及埃因霍温Catharina医院胃肠病学和肝病学部门 |
文献概述
这篇文章探讨了在结直肠息肉的光学诊断中,人工智能(AI)辅助诊断(CADx)系统与人类内镜医师之间的交互作用,以及这种交互作用对医师对CADx系统信任度的影响。
研究发现,当CADx预测与内镜医师初步诊断不一致时,提供校准化的算法置信度分数可以改善信任度。研究使用了60个结直肠息肉的光学诊断案例,让23名内镜医师在有无CADx辅助的情况下进行诊断,并比较了不同的CADx变体对医师信任度的影响。
主要发现包括:
- 内镜医师在初步诊断时的准确率为69.3%,而在考虑CADx预测后的最终诊断准确率提高到了76.6%。
- 当CADx预测与医师初步诊断不一致时,医师在36.5%的情况下会采用CADx的预测。
- 当CADx预测附有置信度分数时,如果分数超过60,医师更倾向于信任CADx的预测。
- 医师对CADx的信任度在可选性CADx(即医师可以选择是否使用CADx)中最高,特别是当没有置信度分数时。
- 医师的信任度受到CADx预测正确性的影响,如果CADx预测错误而医师未采纳,他们的置信度会显著下降。
文章强调了理解人工智能与人类交互的重要性,并指出为了在临床实践中成功实施CADx,需要更好地理解并改善医师与AI的协作方式。
研究还指出,尽管内镜医师在CADx预测后的表现有所提高,但联合性能(医师和CADx)仍未达到美国胃肠内镜学会设定的治疗策略阈值。因此,需要进一步研究来重复和确认这些发现,并探索如何通过提供额外的决策信息(如可解释AI)来提高对AI的信任度。
三、使用人工智能自动化儿童Wilms瘤(肾母细胞瘤)的分割

一作&通讯
| 角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Olivier Hild | 法国贝桑松大学医院小儿外科 |
| 通讯作者 | Yann Chaussy | 法国贝桑松大学医院小儿外科及弗朗什孔泰大学 |
文献概述
这篇文章是一篇关于使用人工智能自动化儿童Wilms瘤(肾母细胞瘤)分割的研究论文。
背景:
- Wilms瘤是儿童中最常见的恶性肿瘤之一,手术治疗在治疗中发挥着重要作用。
- 3D重建肿瘤肾脏有助于手术规划和风险评估,但手动分割过程耗时且易出错。
- 人工智能(AI)和深度学习技术,特别是卷积神经网络(CNN),在医学图像分析中显示出潜力。
方法:
- 研究者使用14名患者的CT扫描构建数据库,并由两位专家进行手动分割。
- 利用CNN U-Net及其变种OV2ASSION方法自动进行Wilms瘤和肿瘤肾脏的分割。
- 通过Dice相似性指数评估自动分割结果,并与专家手动分割结果比较。
结果:
- 两位专家手动分割的Dice指数分别为肿瘤0.95和肾脏0.87。
- 仅使用CNN U-Net进行全自动分割的Dice指数较低,肿瘤为0.69,肾脏为0.27。
- 使用OV2ASSION方法训练的CNN U-Net在不同手动分割间隔(gap)下,Dice指数在肿瘤为0.97至0.94,肾脏为0.94至0.86之间变化。
结论:
- 尽管使用已开发的神经网络如U-Net,但全自动分割在医学图像处理领域仍然是一个挑战,尤其是在儿童肿瘤病理学中。
- 研究者开发了一种新的CNN U-Net训练方法OV2ASSION,能够以与专家相同的精度分割肾脏及其肿瘤,同时将专家的干预时间减少了80%。
讨论:
- 医学图像的AI工具越来越多地被用于图像分析和处理,但儿童病理情况的全自动分割面临挑战。
- 儿童图像质量通常不如成人,且Wilms瘤与成人肾肿瘤在形态上有很大不同。
- 医学图像深度学习的主要障碍之一是缺乏大型、多样化的数据集。
- OV2ASSION方法通过从少量专家手动分割的患者切片中学习,显著减少了专家的干预时间。
结论:
- 尽管没有完全实现全自动分割,但OV2ASSION方法提供了一种提高分割精度和减少专家工作量的途径。
- 未来的工作可能包括增加数据集大小、使用数据增强技术、提供解剖学知识以优化AI工具的性能,以及开发半自动化方法以进一步减少专家干预。
重点关注
Fig. 2展示了使用不同方法进行肾脏和肿瘤分割的结果。

- 第一行:展示了肾脏的分割结果。
- 第二行:展示了肿瘤的分割结果。
每个部分都有四个列,分别代表:
- Column A:CT横截面图像,这是原始的医学图像数据,用于展示肾脏和肿瘤的形态。
- Column B:由人类专家执行的手动分割结果。这些结果是通过专家对CT图像进行分析后,手动标记出的肾脏和肿瘤的区域。
- Column C:使用CNN U-Net自动获得的分割结果。这是没有应用OV2ASSION方法,仅使用U-Net网络进行自动分割的结果。
- Column D:使用CNN U-Net加上OV2ASSION训练方法获得的分割结果。这是应用了OV2ASSION方法后的改进自动分割结果。
从这个图表中,我们可以观察到以下几点:
- 手动分割(Column B)提供了专家级别的精确度,是评估自动分割方法性能的基准。
- 自动分割(Column C)可能在某些情况下无法达到与手动分割相同的精确度,特别是在复杂的病理情况下。
- 应用OV2ASSION方法后的自动分割(Column D)应该在保持较高精确度的同时减少了专家的干预,这可以从Dice相似性指数的提高中看出。
具体来说,通过比较Column B和Column D,我们可以评估OV2ASSION方法对提高自动分割精确度的效果。如果Column D中的分割结果与Column B中的非常接近,这表明OV2ASSION方法有效地提高了分割的准确性,并且能够减少专家的工作量。此外,通过比较Column C和Column D,可以直观地看出OV2ASSION方法对自动分割性能的提升作用。
四、使用集成深度学习技术进行乳腺癌亚型鉴别和侵袭性诊断

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Aadhi Aadhavan Balasubramanian | 东北大学计算机科学学院,波士顿,美国 |
| 通讯作者 | Saeed Amal | 东北大学生物工程学院,波士顿,美国 |
文献概述
这篇文章是关于使用集成深度学习技术进行乳腺癌亚型和侵袭性诊断的研究。
研究团队开发了基于卷积神经网络(CNN)的集成深度学习模型,用于从全切片图像(WSI)组织病理学切片中对乳腺癌亚型和侵袭性进行分类。
研究使用了两个广泛使用的独立数据集:BACH和BreakHis。在BACH数据集中,研究者提出了一种集成策略,结合了VGG16和ResNet50架构,以实现对乳腺癌组织病理学图像的精确分类。此外,研究还引入了一种新颖的图像分块技术来预处理高分辨率图像,使得能够专注于分析图像中的局部感兴趣区域。
研究的关键结果包括:
- 在BACH数据集上,通过图像分块技术,实现了95.31%的分块分类准确率。
- 在BreakHis数据集上,集成模型实现了98.43%的WSI图像分类准确率。
这项研究显著地推动了利用人工智能进行乳腺癌诊断的进程,有潜力改善患者结果并减轻医疗保健负担。研究还讨论了计算效率、算法进步以及未来在临床实践和其他领域利用这些方法的方向。
研究的结论强调了深度学习技术,特别是集成学习在乳腺癌组织病理学图像分类中的有效性,并指出了人工智能在提高乳腺癌诊断和管理效率方面的潜力。
重点关注
Figure 2 展示了来自BreakHis数据集的代表性显微图像,这些图像分别代表了良性和恶性乳腺组织(放大40倍)。

该数据集由9109张乳腺肿瘤组织的显微图像组成,这些图像来自82位患者,并在不同的放大倍数(40倍、100倍、200倍和400倍)下捕获。
数据集包括2480个良性样本和5429个恶性样本。每个样本是一个700×460像素、3通道RGB图像,每个通道具有8位深度,并以PNG格式存储。数据集将肿瘤分为良性和恶性类别,每个类别包含几个子类。
对于良性肿瘤,子类包括:
- 腺病(Adenosis)
- 纤维腺瘤(Fibroadenoma)
- 管状腺瘤(Tubular adenoma)
- 叶状腺瘤(Phyllodes adenoma)
对于恶性肿瘤,子类包括:
- 导管癌(Ductal carcinoma)
- 叶状癌(Lobular carcinoma)
- 粘液癌(Mucinous carcinoma)
- 乳头状癌(Papillary carcinoma)
Figure 2 中的图像以两列展示,第一列代表四种类型的恶性肿瘤组织,第二列代表四种类型的良性组织。这些图像对于病理学家进行诊断至关重要,因为它们提供了组织结构、形态和细胞分布的视觉信息,有助于识别与乳腺癌相关的病理特征。
知识点补充
我估计大家对上面的有一句话,可能看着有点迷糊,所以单独拎出来解释一下——每个样本是一个700×460像素、3通道RGB图像,每个通道具有8位深度,并以PNG格式存储。
这句话描述的是图像的数字化表示和存储格式。
-
每个样本是一个700×460像素的图像:这指的是图像的分辨率,即图像的宽度是700像素,高度是460像素。像素是图像的基本单元,所以这个尺寸定义了图像在宽度和高度上由多少个点组成。
-
3通道RGB图像:这指的是图像使用RGB色彩模式,即红色(Red)、绿色(Green)、蓝色(Blue)三种颜色的组合来表示图像中的所有颜色。每个像素点由这三种颜色的强度值组成,可以合成出各种不同的颜色。
-
每个通道具有8位深度:这通常被称为图像的位深度或颜色深度。8位意味着每个颜色通道用8个二进制位来表示,即每个通道可以有2^8(即256)种可能的值。因此,一个RGB图像可以表示出256×256×256种不同的颜色组合,也就是大约1677万种颜色。
-
以PNG格式存储:PNG(Portable Network Graphics)是一种图像文件格式,用于网络上的图像存储和传输。PNG支持无损压缩,意味着图像在压缩时不会失去质量,适合保存高质量的图像数据。
总的来说,这句话描述的是一种高质量的图像存储方式,其中图像具有较高的分辨率和颜色深度,使用通用的RGB色彩模式,并通过PNG格式进行无损压缩存储,以确保图像数据的完整性和高质量。
五、使用深度学习来预测葡萄膜黑色素瘤(UM)的分子亚型

一作&通讯
| 角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Farhan Akram | 病理学和临床生物信息学系,荷兰鹿特丹伊拉斯姆斯医学中心 |
| 通讯作者 | Jan H. von der Th€usen | 病理学和临床生物信息学系,荷兰鹿特丹伊拉斯姆斯医学中心 |
文献概述
这篇文章是关于使用深度学习来预测葡萄膜黑色素瘤(UM)的分子亚类。
研究的目的是评估基于常规的H&E染色组织切片的深度学习模型在预测UM患者预后方面的有效性,并与传统的分子检测方法进行比较。
研究方法:
- 回顾性研究了113名在1993年至2020年间接受眼球摘除手术的UM患者的肿瘤组织,进行了分子分类分析。
- 使用H&E染色的常规切片,通过扫描获得全切片图像(WSI),然后提取1024x1024像素的图像块进行分析。
- 进行了消融研究以选择最佳表现的深度学习模型,比较了EfficientNet、Vision Transformer和Swin Transformer三种先进的深度学习模型。
研究结果:
- Swin Transformer模型在验证队列中的准确率达到了83%,在测试队列中达到了75%。
- 在分子亚类中,该模型在测试集中正确预测了70%的BAP1突变、61%的SF3B1突变和80%的EIF1AX突变的UM。
- 研究表明,深度学习方法能够使用H&E染色的WSI来预测UM的分子亚类,为不需要分子或免疫组化检测的UM患者提供了高级预后预测的潜力。
结论:
- 这项研究展示了深度学习在多类别预测中预测UM分子亚类的潜力,这可能对没有分子检测能力的实验室尤其有用。
- 研究还表明,每个分子亚类可能具有独特的组织病理学特征,这可能有助于UM的预后预测和肿瘤生物学的理解。
重点关注
Figure 1展示了在这项研究中,如何将患者样本分配到训练集、验证集和测试集,并描述了基于人工智能(AI)的策略来使用苏木精-伊红(HE)染色的全切片图像(WSIs)对UM样本进行分子亚类分类的工作流程。

(A) 样本分布:
- 共有114名患者参与了这项研究。
- 由于一个样本的大小不足且提取的图像块数量不足,该样本被排除在外。
- 剩下的113名患者被进一步分为训练集(60名患者)和测试集(48名患者)。
- 在训练和验证之后,测试集用于评估模型的性能。
- 另外,还有5名同时携带EIF1AX和SF3B1突变的患者被纳入第二项测试,以检验模型对于非典型分子类别的性能。
(B) AI分类策略的工作流程:
- 感兴趣区域(ROIs)的提取:从H&E染色的WSIs中提取出感兴趣的区域。
- 图像预处理:对提取的图像块进行预处理,包括可能的尺寸调整和染色标准化。
- 图像块分类:使用Swin Transformer模型对预处理后的图像块进行分类。
- 患者级别预测:通过将各个图像块的预测结果重组成整个感兴趣区域的预测图,利用多数投票机制确定主导的突变类型。
- 最终预测:基于多数投票的结果,为每个患者确定其UM的分子亚类。
这个流程说明了如何从组织切片图像中提取信息,并使用深度学习模型来预测UM的分子亚类,从而为临床诊断和预后评估提供了一种可能的新方法。
六、比较Wilms瘤和正常胎儿肾脏的基因表达谱以识别潜在的分子生物学机制,并开发新的预后标记和分子靶点

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Qiang Zeng | 江西省儿童医院小儿外科,江西,中国 |
| 通讯作者 | Junfeng Tao | 江西省儿童医院小儿外科,江西,中国 |
文献概述
这篇文章是一项关于Wilms瘤(WT)的研究,这是一种最常见的儿童期肾脏胚胎性肿瘤。
文章的主要目的是通过比较Wilms瘤和正常胎儿肾脏的基因表达谱来识别潜在的分子生物学机制,并开发新的预后标记和分子靶点。
研究方法包括:
- 利用GEO和TARGET数据库中的Wilms瘤转录组数据进行基因表达差异分析。
- 通过基因本体(Gene Ontology, GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路富集分析进行生物学功能分析。
- 通过单变量Cox回归分析识别与预后相关的基因。
- 使用LASSO回归分析增强模型的预测能力。
- 在GSE73209数据集中验证关键枢纽基因,并在WiT-49细胞中进行细胞功能实验以识别这些基因在Wilms瘤细胞中的功能。
研究结果:
- 发现差异表达基因(DEGs)显著参与血管生成和细胞分化的调控。
- 通过PPI网络和MCODE算法识别出24个枢纽基因,其中9个与WT患者的预后相关。
- EMCN和CCNA1被识别为关键枢纽基因,与WT的进展相关。
- 功能上,EMCN和CCNA1的过表达或敲低抑制了Wilms瘤细胞的活力、增殖、迁移和侵袭。
结论:
- EMCN和CCNA1被识别为Wilms瘤的关键预后标记,表明它们作为治疗靶点的潜力。
- 差异基因表达和富集分析显示在血管生成和细胞分化中的重要角色。
重点关注
Fig. 8 展示了在 WiT-49 细胞系中对 EMCN 和 CCNA1 基因功能的验证实验。

-
EMCN 和 CCNA1 基因在细胞系中的表达分析:
- 研究者对 WiT-49 细胞系中 EMCN 和 CCNA1 基因的表达水平进行了分析。
-
转染实验:
- WiT-49 细胞被转染了 EMCN 模拟物(mimic)或阴性对照(mimic NC)以及 CCNA1 的小干扰 RNA(siRNA)或非靶向对照 siRNA(siNC)。
-
转染效率监测:
- 转染后 48 小时,通过绿色荧光蛋白(GFP)的荧光来监测转染效率。
-
mRNA 和蛋白表达水平检测:
- 利用逆转录定量聚合酶链反应(RT-qPCR)和西方印迹(Western blot)技术检测 EMCN 或 CCNA1 的相对 mRNA 和蛋白表达水平。
-
细胞活性测定:
- 使用 CCK-8 实验来确定转染了 EMCN 模拟物或阴性对照的 WiT-49 细胞的活性(E),以及转染了 CCNA1 siRNA 或 siNC 的细胞的活性(F)。
-
细胞迁移和侵袭能力测定:
- 利用 Transwell 实验来评估转染了 EMCN 模拟物或阴性对照的 WiT-49 细胞的迁移和侵袭能力(G),以及转染了 CCNA1 siRNA 或 siNC 的细胞的迁移和侵袭能力(H)。
-
细胞增殖能力测定:
- 通过集落形成实验来评估转染了 EMCN 模拟物或阴性对照的 WiT-49 细胞的增殖能力(I),以及转染了 CCNA1 siRNA 或 siNC 的细胞的增殖能力(J)。
这些实验结果有助于理解 EMCN 和 CCNA1 在 Wilms 瘤细胞行为中的作用,特别是在细胞增殖、迁移和侵袭方面。通过这些功能验证实验,研究者能够进一步探究这两个基因作为潜在治疗靶点的可能性。


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








