小罗碎碎念
本期推文主题:人工智能在病理组学领域的最新进展
厦门开会期间随手写的两篇推文,没想到还挺受欢迎,今天这期算是前两期的一个延续。
我当时提到过可以将领域自适应技术应用于病理AI的研究,今天的第一篇文献就是关于这个的,很有意思的。此外,还有两篇文献值得重点关注——第二篇&第四篇。
第二篇文献系统的比较了八种WSI配准算法的性能,如果正在从事这方面研究的老师/同学,不要错过。第四篇文献用了K近邻和线性回归算法,但是发的NC,恰好我最近又看到了算法与数据的28法则,这篇文献再一次引发了我的思考……暂时没想明白,还需要时间的沉淀。
我是罗小罗同学,下期推文见!!

一、提升医学图像模型泛化能力:一种新颖的领域自适应主动学习策略

一作&通讯
| 角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Dwarikanath Mahapatra | Inception Institute of AI, Abu Dhabi, United Arab Emirates | 阿布扎比起始人工智能研究所,阿拉伯联合酋长国 |
文献概述
这篇文章是关于一种新的医学图像分类方法,名为ALFREDO(Active Learning with FeatuRe disEntanglement and DOmain adaptation),它结合了主动学习(Active Learning)和领域适应(Domain Adaptation)技术来提高模型在分布偏移情况下的泛化能力。
研究背景与动机:
- 深度神经网络(DNNs)在大量标记数据集上表现出色,但难以将学到的知识泛化到新的领域(target domains)。
- 在现实世界中,重新训练网络以适应新应用的大规模数据集收集是不切实际的。
- 医学图像分析中,手动注释数据需要高度的临床专业知识,这限制了模型的实用性。
ALFREDO方法:
- 提出了一种新颖的流水线,通过特征解耦方法将图像特征分解为领域特定(如扫描仪、供应商或医院)和任务特定(如分类、分割任务的判别特征)的组成部分。
- 定义了多个新颖的成本函数来识别在领域偏移下的信息样本。
- 在一个组织病理学数据集和三个胸部X光数据集上测试了所提出的方法。
- 实验表明,与其他领域适应方法以及主动领域适应方法相比,ALFREDO实现了最先进的结果。
方法详解:
- 介绍了特征解耦网络(FDN),使用自动编码器来获取不同组件的特征,并通过不同的损失函数进行训练。
- 提出了基于多个标准的样本信息性评分方法,包括不确定性、领域性、密度和新颖性,以选择信息量大的样本。
实验结果:
- 展示了ALFREDO与其他主动学习、对抗性领域适应方法以及基于不确定性的采样方法的比较结果。
- 进行了消融研究,以展示ALFREDO方法中不同组件的重要性。
结论与未来工作:
- ALFREDO方法有效地结合了主动学习和领域适应,减少了监督设置中的注释成本,并在无监督领域适应中减少了训练准确系统所需的样本数量。
- 未来的工作计划包括在其他医学图像数据集上测试模型,以及评估其对不同分类架构的鲁棒性和泛化能力。
重点关注
Fig. 1 展示了所提出方法 ALFREDO 的工作流程,分为两个主要部分:

(a) 特征解耦(Feature Disentanglement):
- 训练数据通过一个自动编码器(autoencoder)来获取不同的特征组件,记为 ( z_{dom} ) 和 ( z_{task} )。
- 这些特征组件随后用于计算不同的损失函数,以此来训练模型。
- 训练完成后,得到领域特定(domain-specific)和任务特定(task-specific)的特征。
(b) 信息样本选择(Informative Sample Selection):
- 对于未标记的数据(unlabeled data),获取其解耦后的特征表示。
- 计算批次中每个样本的信息性得分(informativeness score)。
- 选择信息性得分最高的样本进行标记(在监督领域适应中),并将这些样本添加到已标记样本中,以启动领域适应步骤。
分析:
- 特征解耦的目的是将输入数据的特征分离成与领域相关的部分和与任务相关的部分,这有助于模型更好地理解和适应不同的数据分布。
- 信息样本选择的目的是识别出最能提供对模型性能改进的样本,这样可以在减少所需标记数据量的同时,保持或提高模型的准确性和鲁棒性。
- 整个流程结合了主动学习的思想,即通过智能地选择样本来优化学习过程,特别适用于领域适应的场景,其中标记数据可能稀缺或成本高昂。
二、乳腺癌组织全切片图像自动配准的前沿技术

一作&通讯
| 角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Philippe Weitz | Department of Medical Epidemiology and Biostatistics, Karolinska Institutet, Stockholm, Sweden | 瑞典卡罗林斯卡医学院医学流行病学与生物统计系 |
文献概述
本文介绍了ACROBAT 2022挑战,旨在通过比较不同算法对乳腺癌组织全切片图像进行自动配准的性能,推动计算病理学在临床诊断中的应用。
摘要
文章讨论了在研究和临床应用中,组织在全切片图像(WSI)之间的对齐的重要性。ACROBAT挑战,基于迄今为止最大的WSI配准数据集,包括1152名乳腺癌患者的4212个WSI。
挑战的目的是将常规诊断免疫组化(IHC)染色的WSI与其相应的H&E染色WSI进行对齐。研究比较了八种WSI配准算法的性能,并调查了不同的WSI属性和临床协变量对性能的影响。
结果
展示了不同的WSI配准方法,包括基于特征和基于强度的方法。讨论了深度学习技术在WSI配准中的广泛应用,并根据目标配准误差(TRE)对算法进行了评估和排名。
分析了当前多染色WSI配准算法的性能,并测试了当前解决方案对来自临床常规数据集的适用性。讨论了最佳性能方法、特征基础配准的有效性以及使用外部数据的影响。
重点关注
Fig. 1 展示了一个经过H&E(苏木精-伊红)染色的组织切片的全切片图像(WSI)以及相应的免疫组化(IHC)染色的组织切片。

这个图由两行组成,第一行(a-e)显示了整个WSI的概览,而第二行(f-j)则展示了在更高放大倍数下的相应组织区域。
具体来说:
- a) 和 f) 展示了H&E染色的WSI,这种染色通常用于显示细胞和组织的形态结构。
- b) 和 g) 展示了使用雌激素受体(ER)抗体进行IHC染色的组织,这有助于识别和评估乳腺癌细胞的激素受体状态。
- c) 和 h) 展示了使用人表皮生长因子受体2(HER2)抗体进行IHC染色的组织,这有助于评估癌细胞的HER2蛋白表达水平,对乳腺癌的分型和治疗有重要意义。
- d) 和 i) 展示了使用增殖细胞核抗原(KI67)抗体进行IHC染色的组织,KI67是一种常用于评估肿瘤细胞增殖活性的标记物。
- e) 和 j) 展示了使用孕激素受体(PGR)抗体进行IHC染色的组织,这有助于了解癌细胞对孕激素的反应性。
通过这种对比展示,研究人员可以更直观地观察和分析不同染色方法对同一组织样本的显示效果,进而用于病理诊断和研究。图像的高倍放大部分有助于观察特定细胞或组织结构的细节,这对于理解肿瘤的生物学特性和制定治疗策略至关重要。
三、数字病理学中的深度学习应用:前景与挑战
一作&通讯
| 作者角色 | 作者姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Peter Boor | 亚琛工业大学病理学研究所 |
文献概述
这篇文章讨论了深度学习(Deep Learning, DL)在数字病理学中的应用前景及其面临的挑战。
- 深度学习的潜力:深度学习通过多层人工神经网络改善疾病诊断的精确性和速度,对病理学和放射学等医学领域具有革命性的潜力。
- 当前应用:DL在病理学中主要用于支持癌症诊断,如在淋巴结中检测癌症微转移、辅助前列腺癌分级和精确量化肿瘤生物标志物。
- 监管合规性:尽管一些工具在病理实验室中得到使用,但只有少数解决方案获得了FDA批准或符合欧盟体外诊断规定。
- 临床肾脏病理学:目前还没有在临床肾脏病理学领域广泛采用DL解决方案,但可以预见DL工具在自动计数肾小球、检测全球性硬化肾小球等方面的应用。
- 未来展望:DL有望实现肾脏组织学的全面量化,从而提供更多有意义的定量参数,可能对疾病分类和临床指南有所贡献。
- 技术挑战:DL集成到病理工作流程中进展缓慢,部分原因是实施新技术的挑战,包括数字化工作流程的初始投资、适应性、维护成本等。
- 经济效益:虽然数字化病理学和DL工具可能带来长期节省,但目前与模拟工作流程相比,成本效益不高,且大多数国家的医疗体系不报销与DL相关的成本。
- 技术集成问题:即使在完全数字化的实验室中,大多数DL解决方案也无法无缝集成到现有工作流程中,因为缺乏标准化接口和互操作性。
- 生态影响:DL解决方案的巨大计算需求可能对环境产生不利影响,需要在开发初期就考虑生态可持续性。
- 专业方法的挑战:肾脏病理学使用的专业方法在其他病理学领域中很少使用,因此需要为这些方法特别开发DL解决方案或调整方法以适应常用技术。
- 数据集的挑战:构建DL模型所需的样本数量是一个挑战,尤其是在罕见肾脏疾病领域,需要国际合作和大型数据集的建立。
文章强调了DL和数字病理学的变革潜力,但也指出了实现这一潜力所面临的挑战,并呼吁采取行动以克服这些挑战。
四、跨越23种人类组织类型的形态学特征与空间RNA表达的自监督学习研究

一作&通讯
| 作者类型 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Francesco Cisternino | 人类技术中心(Human Technopole) |
| 通讯作者 | Craig A. Glastonbury | 人类技术中心(Human Technopole) |
文献概述
这篇文章利用自监督学习技术,通过视觉变换器在大量健康组织图像上训练,实现了对组织亚结构的自动分割、病理特征的检测量化,以及从组织学图像中直接预测和空间定位RNA表达水平。
研究背景:
随着大量组织学档案的数字化,需要能够将特定的组织亚结构和病理变化与疾病结果联系起来,而不需要繁琐的注释。
本文利用视觉变换器(Vision Transformer)在1.7百万组织学图像上进行自监督学习,这些图像涵盖了来自Genotype Tissue Expression (GTEx)联盟的23种健康组织和838名捐赠者。
自监督学习:
通过自监督学习,研究者能够自动将组织分割成其组成的组织亚结构和病理比例,并且在数千张全切片图像上的表现优于其他自监督方法(轮廓系数增加了43%)。
此外,他们还能检测和量化存在的组织病理变化,例如动脉钙化(AUROC = 0.93),并识别缺失的钙化诊断。
RNAPath模型:
为了将基因表达与组织形态联系起来,研究者引入了RNAPath,这是一组在23种组织类型上训练的模型,可以直接从H&E组织学图像预测和空间定位单个RNA表达水平(显著回归的基因数为5156,FDR为1%)。
通过与匹配的免疫组织化学的地面真实数据验证RNAPath的空间预测,重现了它们已知的空间特异性。
组织学图像表示:
通过自监督学习获得的组织学图像表示能够区分组织亚结构和病理特征,而无需标签。
研究者使用了13,898张全切片图像(WSI)进行训练,这些图像来自GTEx的23种组织,涵盖了838名捐赠者。通过训练Vision Transformer(ViT-S)模型,研究者发现学习到的表示能够清晰地捕捉和分离细胞类型(例如脂肪细胞)、病理特征(例如动脉钙化)和组织亚结构(例如胫骨血管层:内膜、中膜、外膜)。
组织亚结构和病理特征的自动分割:
研究者手动标记了一小部分图像瓦片,并由临床病理学家验证。利用这些标记,他们训练了一个k-最近邻(kNN)模型,用于自动分割所有GTEx的WSI,并获得了每个捐赠者样本的亚结构和病理比例。
基因表达与组织形态的关联:
研究者发现,基因表达受到组织亚结构变化的影响。他们通过差异表达分析,观察到组织内及其组成组织亚结构之间的广泛差异表达,中位数为1753个基因(FDR1%)在不同样本中差异表达。
基因与组织亚结构的关联:
通过全基因组关联分析(GWAS)和交互eQTL分析,研究者发现组织亚结构和病理特征的比例与常见的遗传变异有关。
他们还发现,许多组织共享的eQTL效应可能由于实验采样变异和/或捐赠者组织中组织亚结构和病理特征的存在而产生。
RNAPath模型的验证:
研究者在TCGA-BRCA(乳腺癌)数据集上验证了RNAPath模型。他们发现,即使没有在肿瘤数据上训练,RNAPath也能够预测正常乳腺和乳腺癌中的基因表达。
文章展示了如何通过自监督学习从大量组织学档案中提取有用的生物学信息,并展示了这种方法在理解组织病理学、其空间组织以及形态组织变异与基因表达之间相互作用方面的潜力。
重点关注
Fig. 1 展示了如何使用从全切片图像组织学(WSI)中学习到的自监督表示来进行组织亚结构的分割、病理特征的识别,以及利用 RNAPath 理解形态学-表达-遗传关联的示意图。

A. 组织学图像预处理:全切片图像(WSI)首先通过分割和瓦片化处理,被划分成 63 × 63 微米平方的区域。
B. 自监督特征提取:使用自监督学习方法从这些瓦片中提取形态学特征。

C. 瓦片分类与表型分析:利用学习到的特征,通过 K-最近邻(kNN)模型对瓦片进行分类,并根据样本中检测到的区域范围推导出表型。
D. RNAPath 模型预测:RNAPath 模型接受瓦片嵌入作为输入,预测局部(瓦片级别)和全局(样本级别)的基因表达,并生成热图来可视化预测的空间基因活性。
这个流程图说明了如何将自监督学习应用于组织学图像分析,以实现对组织结构的深入理解和基因表达的精确预测。通过这种方式,研究人员可以在不需要大量手动注释的情况下,自动识别和量化组织中的不同亚结构和病理特征,并进一步探索这些特征与基因表达之间的关系。
五、利用cfDNA甲基化特征提高肿瘤检测的准确性

一作&通讯
| 作者角色 | 作者姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Xu Hua | 哥伦比亚大学欧文医学中心癌症遗传学研究所 |
| 第一作者 | Hui Zhou | 哥伦比亚大学欧文医学中心癌症遗传学研究所 |
| 通讯作者 | Zhiguo Zhang | 哥伦比亚大学欧文医学中心 |
文献概述
这篇文章通过分析血浆中无细胞DNA的对称和半甲基化模式,开发了一种新的多癌种检测方法。
研究背景:
癌症是全球主要的公共健康问题,早期肿瘤检测对于改善癌症患者的预后至关重要。液体活检,尤其是基于血浆cfDNA的检测,因其非侵入性和能够克服肿瘤异质性带来的挑战,显示出早期癌症检测的潜力。
研究目的:
开发并验证一种新方法,通过分析血浆cfDNA的甲基化模式(包括对称甲基化和半甲基化)来检测肿瘤。
研究方法:
- 使用了改进的甲基化DNA免疫沉淀测序(MeDIP-Seq)技术,包括针对基因组DNA的ssg-MeDIP-Seq和针对血浆cfDNA的sscf-MeDIP-Seq。
- 通过这些方法,研究者能够分析cfDNA中的对称甲基化区域(DMRs)和半甲基化区域(DHMRs)。
研究发现:
- 大多数肝肿瘤DNA或血浆cfDNA中的DHMRs与同一样本的DMRs不重叠,表明DHMRs可以作为独立的生物标志物。
- 通过分析215个样本的cfDNA甲基化组,训练了使用DMRs、DHMRs或两者结合的机器学习模型。结合DMRs和DHMRs的模型显示出较单独使用DMRs或DHMRs的模型有更好的性能。
研究结果:
- 利用DMRs和DHMRs的模型在区分健康对照组、肝癌和脑癌方面表现出色,验证队列中的AUROC值分别达到0.978、0.990和0.983。
- 研究表明,使用sscf-MeDIP-Seq程序作为生物标志物,可以提高多癌种检测的准确性。
研究意义:
这项研究支持了利用DMRs和DHMRs进行多癌种检测的潜力,为癌症的早期发现和治疗提供了新的方法。
这篇文章提供了一种新的癌症检测方法,通过分析血浆cfDNA的甲基化模式,有助于提高癌症早期检测的准确性和可靠性。
重点关注
表1提供了271个用于本研究的cfDNA样本的患者信息,包括癌症类型、性别和年龄分布。

-
样本总数:共有271个cfDNA样本被用于研究。
-
性别分布:
- 男性:127个样本在训练队列中,34个样本在验证队列中。
- 女性:87个样本在训练队列中,22个样本在验证队列中。
- 未知性别:训练队列中有1个样本,验证队列中没有。
-
年龄分布:
- 年轻(小于30岁):训练队列中有13个样本,验证队列中有6个样本。
- 中年(30至60岁):训练队列中有107个样本,验证队列中有25个样本。
- 老年(超过60岁):训练队列中有95个样本,验证队列中有25个样本。
- 未知年龄:没有样本的年龄信息未知。
-
癌症类型:
- 脑癌:训练队列中有77个样本,验证队列中有20个样本。其中IDH野生型(IDH WT)34个,IDH突变型(IDH mutant)43个。
- 肝癌:训练队列中有58个样本,验证队列中有15个样本。
- 健康对照组:训练队列中有80个样本,验证队列中有21个样本。
-
队列分布:
- 训练队列:共215个样本,用于模型的训练。
- 验证队列:共56个样本,用于独立验证模型的准确性。
表1的信息对于理解研究的样本选择和代表性至关重要,它显示了研究团队试图在不同癌症类型、性别和年龄组之间保持平衡,以确保研究结果的广泛适用性和准确性。
六、染色质图像的无监督学习揭示导管原位癌细胞状态变化
一作&通讯
| 作者角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Xinyi Zhang | 麻省理工学院电子工程与计算机科学系 |
| 通讯作者 | Caroline Uhler | 麻省理工学院电子工程与计算机科学系&哈佛-麻省理工学院博德研究所 |
| 通讯作者 | G. V. Shivashankar | ETH 苏黎世瑞士联邦理工学院健康科学与技术系,保罗谢勒研究所纳米生物实验室 |
文献概述
这篇文章通过无监督表示学习技术,利用染色质图像分析,揭示了导管原位癌(DCIS)中细胞状态和组织结构的变化,为乳腺癌的早期诊断和治疗提供了新的生物标志物。
DCIS是一种可能发展成侵袭性乳腺癌的前侵性肿瘤,研究者们生成了一个大规模的组织微阵列图像数据集,包含560个样本,来自122名女性患者的3个疾病阶段和11个表型类别。
研究者使用表示学习技术,仅依靠染色质图像,无需多重染色或高通量测序,成功识别了8种不同的细胞形态状态和组织特征,这些特征标志着DCIS的变化。所有细胞状态在所有疾病阶段都有所观察,但比例不同,表明在正常乳腺组织中以小比例存在的富含侵袭性癌症的细胞状态。组织层面的分析揭示了细胞状态空间组织在不同疾病阶段的显著变化,这可以预测疾病的阶段和表型类别。
研究表明,染色质成像是衡量DCIS细胞状态和疾病阶段的有力工具,提供了一种简单有效的肿瘤生物标志物。研究还发现,通过简单的染色质染色结合图像自编码器(一种无监督学习神经网络),可以获取有意义的细胞状态。此外,研究者还利用神经网络分类器,基于自编码器的潜在表示来预测细胞所属的表型类别,进一步验证了细胞状态的区分度。
文章强调,通过使用计算工具对核形态进行定量和一致的评估,可以补充病理注释,有助于更好地理解DCIS的机制,从而为患者推荐适当的治疗方案。研究结果不仅展示了使用染色质图像进行大规模研究的可行性,还表明了这种方法在预测疾病阶段和表型类别方面具有高度准确性。
重点关注
Fig. 1 展示了在研究中使用的大规模高分辨率染色质成像数据集的结构和组织方式,这些数据集使得对DCIS的疾病阶段和表型类别进行分析成为可能。
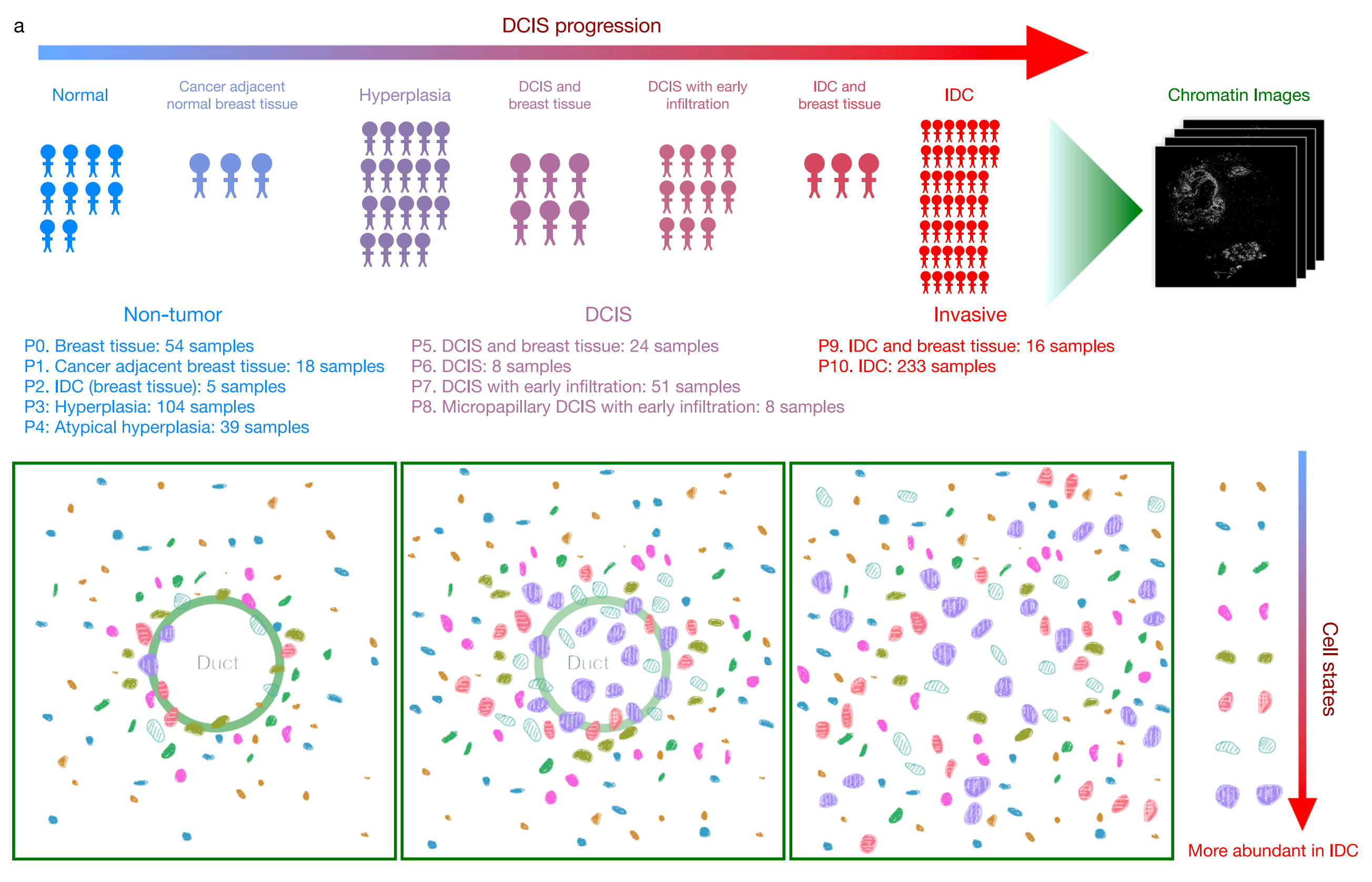
a. 描述了11种不同的表型类别,这些类别分布在3个疾病阶段:
- 非肿瘤阶段
- 导管原位癌(DCIS)
- 侵袭性导管癌(IDC)
类别按照从非肿瘤阶段的正常乳腺组织(P0)到IDC阶段(P9和P10)的顺序排列。列出了每个阶段成像的样本数量。特别地,IDC(乳腺组织)(P2) 指的是位于IDC附近的非肿瘤组织的样本;癌症相邻的乳腺组织(P1)指的是紧挨着IDC的非癌症组织;IDC和乳腺组织(P9)指的是主要包含癌症组织但也包含一些正常乳腺组织的样本。

b. 样本被组织进多个组织微阵列(TMAs),每个TMA都进行了染色质和其他蛋白的染色。使用角蛋白染色来分割乳腺导管。成像的分辨率为0.18微米/像素,这允许对细胞核的详细观察和分析。
整体来看,Fig. 1 提供了研究数据集的概览,包括样本的分类、组织方式以及成像的分辨率,这些信息对于理解研究方法和结果至关重要。


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








