小罗碎碎念
文献精读:AI在三阴性乳腺癌肿瘤浸润性淋巴细胞评估中的应用
这篇文章发表于《CLINICAL CANCER RESEARCH》,目前IF=10,医学&肿瘤学1区。
文中提到了一种开源的、自动化的肿瘤浸润性淋巴细胞(Tumor-Infiltrating Lymphocyte, TIL)算法,用于三阴性乳腺癌(Triple-Negative Breast Cancer, TNBC)的预后评估。

一作&通讯
| 角色 | 姓名 | 单位(英文) | 单位(中文) |
|---|---|---|---|
| 第一作者 | Yalai Bai | Department of Pathology, Yale School of Medicine | 耶鲁大学医学院病理学系 |
| 通讯作者 | Balazs Acs | Department of Oncology-Pathology, Karolinska Institutet | 卡罗林斯卡医学院肿瘤病理学系 |
| 通讯作者 | David L. Rimm | Department of Pathology, Yale School of Medicine | 耶鲁大学医学院病理学系 |
研究团队使用QuPath开源软件构建了一个基于神经网络的分类器,用于在苏木精-伊红(H&E)染色切片上识别肿瘤细胞、淋巴细胞、成纤维细胞和其他细胞。
通过分析分类器得出的TIL测量值,研究者们构建了五个独特的TIL变量,并使用171例TNBC病例的回顾性收集作为发现集,以确定机器读取的TIL变量与患者预后之间的最佳关联。此外,研究者们还评估了包含749名TNBC患者的四个独立验证子集的回顾性收集。
研究结果显示,所有五个机器TIL变量都与预后有显著的关联(所有比较的P值≤0.01),但在验证集中显示出细胞特异性的变化。Cox回归分析表明,所有五个TIL变量在调整了包括阶段、年龄和组织学等级在内的临床病理因素后,仍然与改善的总体生存率独立相关(所有分析的P值≤0.0003)。
研究结论是,由神经网络驱动的细胞分类器定义的TIL变量是几个独立验证队列中TNBC患者的稳健和独立的预后因素。这些客观的开源TIL变量可以免费下载,并且现在可以考虑在前瞻性设置中进行测试,以评估其临床效用。
文章还讨论了TIL评估在乳腺癌患者护理日常实践中的临床效用,由于评估的主观性和缺乏标准化,目前仍受限。
国际免疫肿瘤生物标志物工作组已努力标准化TIL评估,并引入了达到良好但非完美可重复性的TIL评估指南。自动化数字分析和机器学习算法的使用提供了解决标准化和操作者变异问题的可能方案。
研究还提到,尽管已开发出基于机器学习的TIL分析算法并显示出预后潜力,但其中只有少数在TNBC队列中进行了测试,并且几乎所有算法都是“黑箱”操作,即算法无法还原为特定细胞或细胞特征的数量,且算法既不是开源的,也不容易为现实世界的区域病理部门所获得。
文章最后指出,这项研究的目的是识别和验证一种透明、可访问的、操作者独立(可重复的)和在TNBC队列中具有预后价值的基于细胞类型的变量的收集和使用方法。
研究还提到了该算法的构建过程、TIL变量的构建、统计分析方法以及研究的转化相关性。
一、引言
近期临床试验表明,宿主抗肿瘤免疫,通过基质肿瘤浸润淋巴细胞(sTIL)测量,在原发性三阴性乳腺癌(TNBC)中具有重要的临床意义(参考文献1–4)。
多项研究数据显示,sTIL水平升高与早期和转移性TNBC的无复发生存期延长和对新辅助治疗的更好反应相关(参考文献1, 4–8)。然而,由于主观性和缺乏标准化,sTIL在实际乳腺癌患者护理中的临床应用仍然受限。
因此,国际乳腺癌免疫肿瘤生物标志物工作组致力于标准化TIL评估,并引入了TIL评估指南,该指南在国际环形研究中已达到良好但非完美的可重复性(参考文献9–12)。然而,在真实世界临床实践中,由于定量评估组织学特征的困难和主观性,观察者内和观察者间的变异性可能仍然是一个问题(参考文献13–15)。
利用机器学习衍生算法的自动化数字分析可能为标准化和操作者主观性差异问题提供解决方案(参考文献13)。
最近开发的基于机器学习的TIL分析算法已显示出预后潜力,这些研究中,TILs在肿瘤内基质或整个肿瘤区域进行分析(参考文献16–21),模拟病理学家评估TILs的方式,每个标记反映了给定隔室中独特的TIL空间信息。
此外,只有少数报道的TIL评分算法在TNBC队列中进行了测试(参考文献16, 18, 20),且几乎所有算法都是“黑箱”式的,即算法不能被翻译回特定细胞或细胞特征的数目,这些算法既不是开源的,也不易被当前现实世界地区的病理科实现。
在此,我们使用开源软件平台(QuPath)构建了一个基于识别特定细胞类型的机器学习乳腺癌TIL评分算法。
通过对细胞类型进行数学处理,可以得到可能具有意义的TIL测量变量,这些变量不易由病理学家计算。
我们首先定义四种细胞类型(肿瘤细胞、TILs、成纤维细胞和其他细胞),然后数学地结合它们,创建评估不同细胞群体中TIL比例或变量组织隔室中TIL密度的变量。
本研究的目的是识别并验证一种透明、可访问的方法,用于收集和使用基于细胞类型的变量,该变量操作者独立(可重复)且在TNBC队列中具有预后价值。
二、材料与方法
2-1:患者队列
本研究回顾性收集了920例TNBC患者的数据,包括五个独立队列:
- 三个来自耶鲁大学医学院病理科
- 一个来自癌症基因组图谱(TCGA)
- 一个来自瑞典南部斯科讷医疗保健区的瑞典国家乳腺癌质量注册中心(NKBC)数据。
表1提供了发现集和验证集中患者的临床病理信息。

-
患者总数:发现集(WTS Yale)有171例患者,而四个独立的验证集(TMA Yale1、TMA Yale2、WTS TCGA、WTS Sweden)总共有749例患者。
-
年龄分布:
- 在WTS Yale中,28.7%的患者年龄小于50岁,53.8%的患者年龄在50岁或以上,17.5%的年龄数据缺失。
- 其他验证集中年龄小于50岁的患者比例从22.7%到38.8%不等,年龄在50岁或以上的患者比例从54.3%到77.3%不等。
-
种族分布:
- WTS Yale中,71.3%的患者为白人,22.2%为非洲裔美国人,5.3%为其他种族,1.2%的数据缺失。
- 在其他验证集中,白人患者的比例从53.2%到89.2%不等,非洲裔美国人的比例从6.5%到28.0%不等。
-
肿瘤大小(厘米):
- WTS Yale中,43.3%的患者肿瘤大小小于2厘米,37.4%的患者肿瘤大小在2厘米以上,19.3%的数据缺失。
- 其他验证集中,肿瘤大小小于2厘米的患者比例从38.1%到46.8%不等,肿瘤大小在2厘米以上的患者比例从37.4%到56.9%不等。
-
组织学分级:
- WTS Yale中,1.2%的患者为分化良好(Well-DI),22.8%为中度分化(Moderate-DI),71.3%为分化不良(Poor-DI),4.7%的数据缺失。
- 其他验证集中,分化良好的患者比例从0%到1.4%不等,中度分化的患者比例从10.2%到30.2%不等,分化不良的患者比例从35.3%到88.4%不等。
-
临床分期:
- WTS Yale中,35.1%的患者为I期,45.6%为II期,7.6%为III期,4.1%为IV期,7.6%的数据缺失。
- 其他验证集中,I期患者的比例从13.7%到36.7%不等,II期患者的比例从36.7%到62.0%不等,III期患者的比例从7.6%到38.1%不等,IV期患者的比例从1.0%到22.7%不等。
-
化疗情况:
- WTS Yale中,32.7%的患者接受了化疗,3.5%未接受化疗,63.7%的数据缺失。
- 其他验证集中,接受化疗的患者比例从49.6%到73.1%不等,未接受化疗的患者比例从2.5%到11.2%不等。
-
随访时间(月):
- WTS Yale的中位随访时间为66.1个月,范围从0.5到233.1个月。
- 其他验证集中,中位随访时间从13个月到64.8个月不等。
表1的数据为研究人员提供了每个队列的详细基线特征,这些信息对于评估TIL评分算法的预后潜力至关重要,因为它们可以帮助识别可能影响患者结果的其他因素。
TNBC定义
在耶鲁大学和TCGA队列中,TNBC定义为雌激素受体(ER)、孕激素受体(PR)免疫组化(IHC)染色细胞小于1%,HER2 IHC染色评分小于2,或对于IHC 2+的患者,无扩增的ISH状态。
在瑞典,TNBC的定义是ER和PR IHC染色细胞小于10%(包括1%–9%染色的细胞)和HER2 IHC染色评分小于2,或对于IHC 2+的患者,无扩增的ISH状态。
TMA Yale1和TMA Yale2队列的代表肿瘤区域由病理学家根据H&E染色切片选择。肿瘤核心由耶鲁病理组织服务设施(YPTS)打孔并排列到接收块中(每个直径0.6毫米)。
在TMA Yale1队列中,所有病例都有重复的TMA核心,而在TMA Yale2队列中,具有1倍、2倍和3倍冗余的组织核心数量和百分比分别为76(27%)、124(45%)和78(28%)(补充表S1)。
TMA队列中每个肿瘤的平均多个核心(每个肿瘤0.57 mm²)进行了分析。对于整体组织切片集,由病理学训练的研究科学家为本研究选择了每个患者的整张切片。WTS Yale、WTS TCGA和WTS Sweden的平均评估面积分别为51.2 mm²、59 mm²和96.1 mm²(补充表S2)。我们的分类器训练集包含来自多个较老耶鲁队列的随机选择的95例乳腺癌患者的97个TMA点。
2-2:数字图像分析
在耶鲁队列中,使用Aperio ScanScope CS2平台(Leica Biosystems)以20倍放大率扫描H&E染色切片,像素大小为0.4986 mm × 0.4986 mm。
WTS TCGA图像从NIH CDC标本存储库下载(参考文献23;https://portal.gdc.cancer.gov/repository)。在WTS Sweden队列中,使用NanoZoomer 2.0-HT(Hamamatsu Photonics K.K.)平台以20倍放大率数字化H&E染色切片,像素大小为0.4537 mm × 0.4537 mm。
使用QuPath开源软件平台(版本0.1.2)构建自动化的TIL评分算法(参考文献24, 25)。
由于H&E染色的日期在队列之间和队列内部都有所不同,我们使用QuPath中的“估计染色向量”功能对每个数字化的切片进行了H&E染色估计的优化。
使用watershed细胞检测方法(25)对图像中的细胞进行分割,以下为设置参数:
- 检测图像:苏木精OD;
- 请求的像素大小:0.5 mm;
- 背景半径:8 mm;
- 中值滤波器半径:0 mm;
- σ:1.5 mm;
- 最小细胞面积:10 mm²;
- 最大细胞面积:400 mm²;
- 阈值:0.1;
- 最大背景强度:2。
- 细胞扩展:5 mm。
细胞分割的质量控制由两名病理学家(DR和BA)执行。为了将检测到的细胞分类为肿瘤细胞、免疫细胞(TILs)、成纤维细胞和其他(错误检测、背景;补充图S1),我们使用了具有八个隐藏层的神经网络作为机器学习方法(最大迭代次数:100)。
分类中使用的特征已先前描述(参考文献21)。为了帮助算法进行准确的分类,我们还添加了25和50 mm半径的平滑对象特征,以补充单个细胞的现有测量。对分类器训练集进行了多轮细胞分类审查和修正,以实现最准确的算法,最终得到名为“CNN11”的算法。
2-3:构建乳腺癌TIL量化算法
基于开源软件,建立了一个用于组织图像定量分析的流程图(图. 1)。
图1展示了从算法训练到算法验证的流程图,分为三个主要部分:
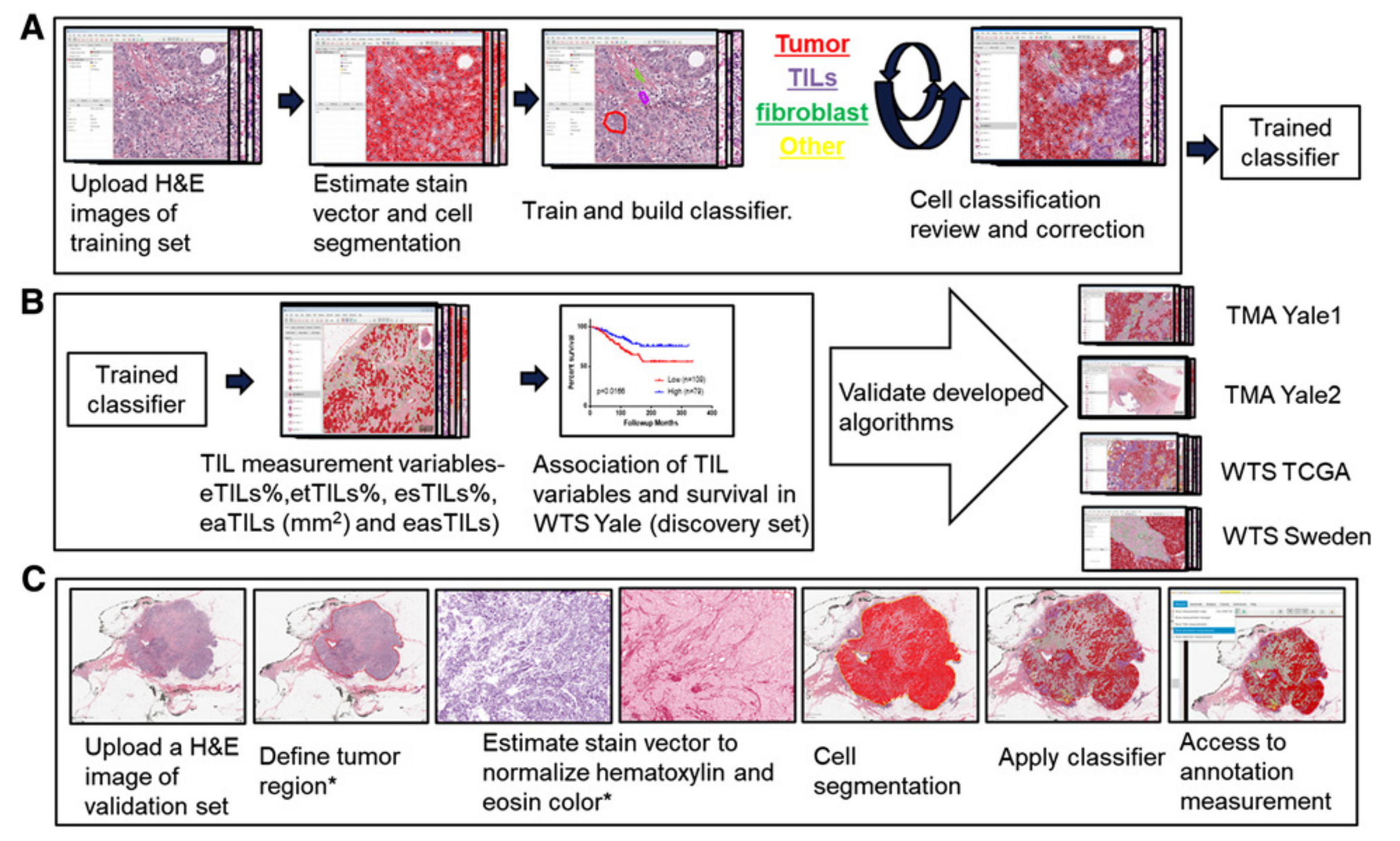
A. 算法训练:
- H&E图像上传:首先上传苏木精-伊红(H&E)染色的图像。
- 染色矢量估计:上传后首先定义染色矢量,以标准化不同批次的染色特性。
- 细胞分割:使用标准化的分水岭(watershed)细胞检测参数进行细胞分割。
- 分类器训练:使用神经网络对肿瘤细胞、TILs、成纤维细胞和其他细胞类型(错误检测或背景)进行分类,并为每种类型进行颜色编码。
- 临时分类器构建:构建一个临时分类器,并将其应用于分类器训练集中的其余图像。
- 多轮审查和校正:经过多轮细胞分类审查和校正,直到大多数图像的形态学判断准确率达到至少95%。
- 训练完成:一旦分类器(CNN11)在大多数图像上达到至少95%的准确率,就锁定该分类器。
B. TIL变量的计算与应用:
- 分类器应用:应用训练好的分类器,得到TIL测量值。
- TIL变量计算:根据得到的测量值,计算以下TIL变量:eTILs%、etTILs%、esTILs%、eaTILs(每平方毫米)、easTILs(具体定义见材料与方法部分)。
- 结果关联分析:在WTS Yale(发现集)中使用X-tile软件确定的最佳切割点,确定TIL变量与患者预后之间的关联。
- 验证集测试:所有TIL变量随后在验证集中进行测试,包括TMA Yale1、TMA Yale2、WTS TCGA和WTS Sweden。
C. 整个组织图像中TIL量化的工作流程:
- 肿瘤区域定义:定义肿瘤区域的步骤,然后估计染色矢量以标准化H&E颜色。
- 细胞分割与分类:使用标准化的分水岭细胞检测参数进行细胞分割,然后使用训练好的分类器进行细胞分类。
- TIL测量分析:最后,将TIL测量值分析为构建的变量。
- 病理学家监督:整个过程需要病理学家的监督。
整个流程图强调了算法的透明度和可重复性,以及病理学家在图像分析和质量控制中的关键作用。通过这种方法,研究者能够将传统的病理学评估与现代图像分析技术结合起来,以提高TIL评估的准确性和一致性。
应用CNN11后,程序能够提供图像中四种类型细胞的数量,包括:
- (i)肿瘤细胞
- (ii)TILs
- (iii)成纤维细胞
- (iv)其他细胞
对于整体组织切片(WTS)集,肿瘤区域注释是根据国际免疫肿瘤生物标志物工作组引入的指南定义的(参考文献10),包括:
- (i)包括在侵袭性肿瘤边界内的TILs,包括“中心肿瘤”和“侵袭性边缘”;
- (ii)所有单核细胞(包括淋巴细胞和浆细胞)都应计分,但多形核白细胞被排除;
- (iii)排除肿瘤边界外的TILs;
- (iv)排除围绕DCIS和正常小叶的TILs;
- (v)排除压碎伪影、坏死、退行性硬化区域以及之前的芯活检部位。
两名病理学家(DR和BA)对算法分类检测到的细胞进行了质量控制。使用细胞分类掩模的工作流程和图像示例分别显示在图. 1C和图. 2中。
图2展示了四个样本案例的代表性图像,包括苏木精-伊红(H&E)染色图像和相应的细胞分类蒙版。

-
H&E图像与细胞分类蒙版对比:
- 图A、C、E和G显示了四个不同样本的H&E染色图像。
- 图B、D、F和H展示了与A、C、E和G相对应的细胞分类蒙版。
-
细胞分类颜色编码:
- 肿瘤细胞用红色表示。
- 肿瘤浸润性淋巴细胞(TILs)用紫色表示。
- 成纤维细胞用绿色表示。
- 其他类型(可能包括背景或错误检测)用黄色表示。
-
图像准确性:
- 图E和F展示了样本中细胞分类不准确的代表性图像。这些罕见的领域最终被审查并排除(censored)。
- 这表明算法在某些情况下可能无法准确分类所有细胞,需要进一步的审查和可能的人工校正。
-
图像选择:
- 图G和H展示了只选择和分析侵袭性乳腺癌区域的图像。这意味着在进行TIL分析时,研究者专注于与癌症侵袭性相关的区域。
-
比例尺:
- A至F图中的比例尺为20毫米,这表明这些图像是局部放大的,用于展示特定区域的细胞分类细节。
- G至H图中的比例尺为200毫米,这表明这些图像可能展示了更大范围的组织,用于展示侵袭性癌症区域的选择。
-
图像质量与算法限制:
- 尽管算法旨在实现高准确度,但图E和F中的示例显示,在某些情况下可能需要人工审查来纠正分类错误。
图2通过视觉方式展示了算法在实际应用中的表现,包括其准确性和在特定情况下的局限性。这强调了在自动化图像分析中结合人工审查的重要性,以确保最终结果的准确性和可靠性。
我们在运行算法之前排除了组织伪影区域,例如坏死。算法不能替代病理学家,因为它不能选择正确的分析区域,也不能消除常见伪影。最后,在细胞分配后对算法输出进行了质量控制。
在验证集中,肿瘤区域注释是由不同的病理学家(DR、BA和JH)定义的。此外,为了证明用户独立性,不同的观察者(YB和BA)运行了算法。我们观察到在1%-2%的病例中出现了灾难性的分割失败,这些病例必须标记为重复分析或如果在某些异常伪影导致失败的情况下被排除。
这在浸润性小叶癌中最常见,在一小部分高比例iTIL的病例中也很常见。Yale WTS和Sweden WTS队列的基质TILs(sTIL)也根据国际免疫肿瘤生物标志物工作组发布的指南,由美国(KC)和瑞典(JH)的专家乳腺病理学家传统视觉评估。
2-4:构建TIL变量
基于CNN11的乳腺癌分类算法得到的组织注释测量包括:
-
(i)分配到上述定义的四种细胞类型之一,
-
(ii)注释区域(mm²)(整个肿瘤区域)
-
(iii)每种细胞类型的累积面积(mm²)。
TIL测量在以下构建的变量中进行分析(补充图S1):
- eTILs% = 100 × [TIL数量/(肿瘤细胞数量 + TIL数量)];代表TILs占肿瘤细胞的比例。
- etTILs% = 100 × (TIL数量/总细胞数量);代表TILs占所有检测细胞的比例。
- esTILs = 100 × [TIL数量/(总细胞数量 - 肿瘤细胞数量)];代表TILs占基质细胞的比例。
- eaTILs (mm²) = TIL数量/分析的肿瘤区域总面积(mm²);代表TILs在肿瘤区域的密度。
- easTILs = 100 × [TIL面积总和(mm²)/基质面积(mm²)];模拟国际TIL工作组变量,如病理学家所读。
需要注意的是,在变量#5中,基质面积(mm²)= 分析的肿瘤区域总面积(mm²) - 肿瘤细胞面积(mm²);easTILs代表TILs在基质面积的密度,模拟国际免疫肿瘤生物标志物工作组关于乳腺癌的指南中病理学家对sTIL的评分。然而,所有变量都包括了iTILs的测量。排除了高iTIL比例的病例。
2-5:统计分析
总生存期(OS)定义为从肿瘤原发诊断日期到因任何事件导致的死亡日期之间的时间,如果患者仍然存活,则为最后一次审查的日期。
我们使用X-tile软件可视化连续数据及其与患者预后的关联(图. 1B;参考文献26)。
X-tile软件确定的每个TIL变量的统计学显著阈值(截点)在验证集中进行了测试。
三、结果
3-1:CNN11 TIL算法在WTS Yale发现队列中的开发
机器学习通常用于定义与人类TIL评分或结果相关的黑盒算法,而与传统病理学的参数无关。
在本研究中,我们采取了不同的方法来训练算法,定义熟悉的细胞类型,并对细胞类型计数进行数学运算,如材料和方法部分所述(图1;补充图S1)。因此,从单一细胞发现(分割)算法CNN11,我们生成了五个候选变量,我们可以测试和比较,以选择未来使用的最佳变量。
值得注意的是,这种方法,虽然自动化,但结果变量对传统病理学来说是熟悉的。这五个变量被选择来测试预后价值和潜在的未来用途作为客观生物标志物。
接下来,我们测试了每个变量的数值与患者预后的关联。
这不再是一个训练过程,而是一个寻找最佳截点的最优过程,因此我们获得了与TNBC病例(WTS Yale集)整个组织切片集相关的预后信息,该集与CNN11细胞分割算法训练集是独立的。
这个发现集被用来为每个TIL变量找到最佳截点。为了测试这些变量在发现集中的预后价值,我们使用X-tile软件来可视化每个可能的截点以及每个TIL变量与患者预后的关联。
图3显示了每个变量的最佳截点以及为评估队列最佳截点得出的未调整P值。
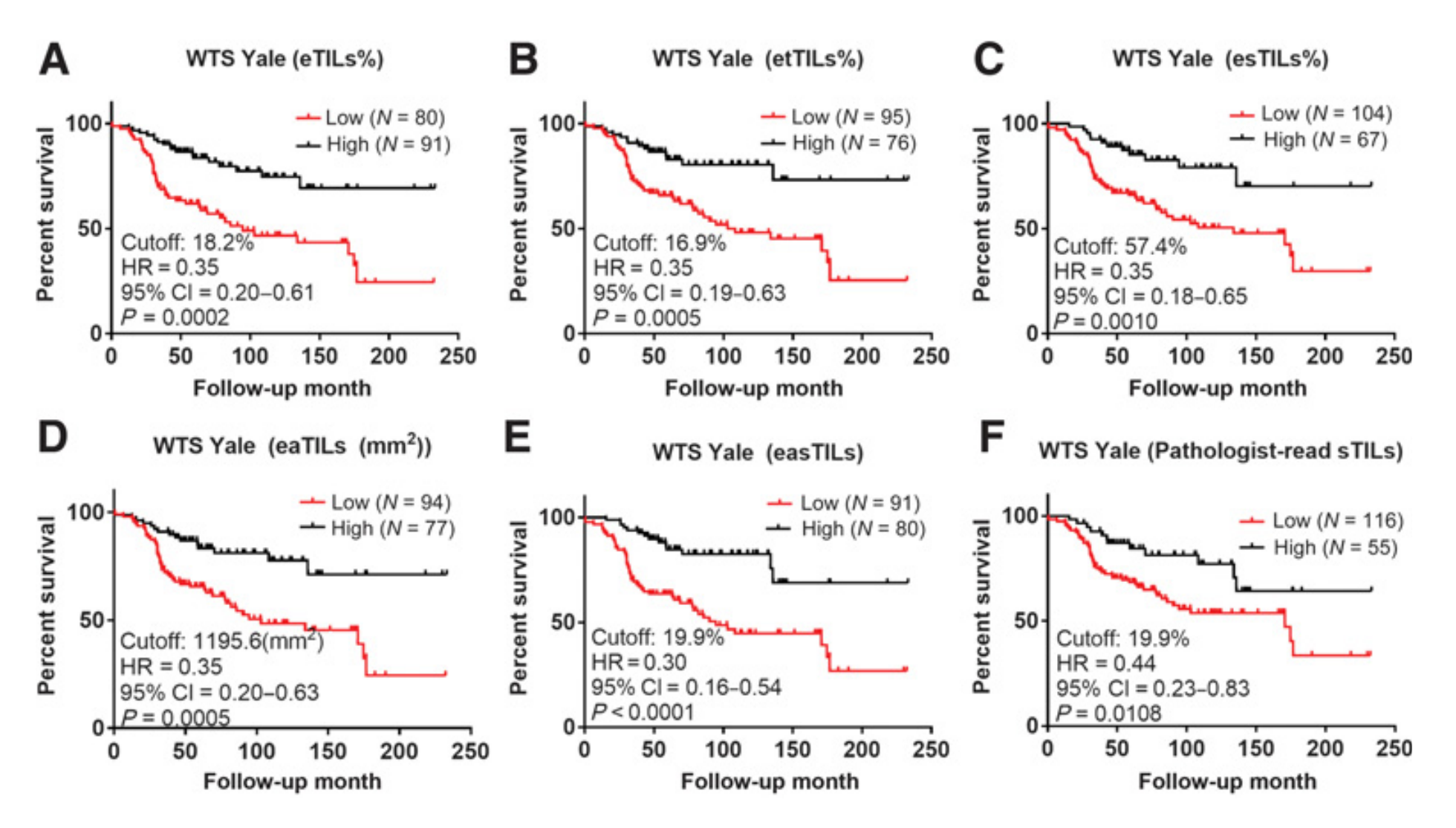
每个图中的截点表示为细胞百分比或细胞/mm²,显示在图的插图中。
我们选择从开始就尝试五个变量,但可以生成的变量数量要大得多。例如,高eTILs%(≥18.2%)与低eTILs%的患者相比,具有统计学上显著更好的OS率[危险比(HR):0.35,置信区间(CI):0.20–0.61,P:0.0002]。
WTS Yale队列中其他四个TIL变量也观察到类似的临床关联,每个变量都有自己的最佳截点:
- etTILs%(阈值:≥16.9%,HR:0.35,95% CI:0.19–0.63,P:0.0005);
- esTILs(阈值:≥57.4%,HR:0.35,95% CI:0.18–0.65,P:0.001);
- eaTILs(mm²)(阈值:≥1195.6/mm²,HR:0.35,95% CI:0.20–0.63,P:0.0005)
- easTILs(阈值:≥19.9%,HR:0.30,95% CI:0.16–0.54,P<0.0001)。
病理学家读取的sTIL评分也与OS显著相关(阈值:≥19.9%,HR:0.44,95% CI:0.23–0.83,P:0.01;图3)。
3-2:CNN11 TIL算法变量的性能在验证集中的表现
接下来,为了测试每个变量的预后价值,我们在四个完全独立的队列中进行了验证;TMA Yale 1、TMA Yale 2、WTS TCGA和WTS Sweden。使用上述定义的特定TIL变量的截点,TMA Yale1集的患者,所有五个变量都被显著地分为有利的和不良的预后亚组[eTILs%:HR = 0.64,95% CI = 0.43–0.94,P = 0.025;etTILS%:HR = 0.51,95% CI = 0.32–0.81,P = 0.004;esTILs:HR = 0.48,95% CI = 0.25–0.89,P = 0.02;eaTILs (mm2):HR = 0.48,95% CI = 0.31–0.74,P = 0.0009;easTILs:HR = 0.65,95% CI = 0.43–0.98,P = 0.04](补充图S2)。
在TMA Yale2队列中,eTILs%,etTILs%,和esTIL评分与OS显著相关,而eaTILs和easTILs则不相关[eTILs%:HR = 0.43,95% CI = 0.26–0.69,P = 0.0005;etTILS%:HR = 0.47,95% CI = 0.28–0.77,P = 0.003;esTILs:HR = 0.42,95% CI = 0.24–0.76,P = 0.004;eaTILs (mm2):HR = 0.62,95% CI = 0.37–1.01,P = 0.06;easTILs:HR = 0.78,95% CI = 0.48–1.26,P = 0.31;补充图S3],显示了变量的固有不同特性。
在临床环境中,TIL评估仅在WTS切片上进行;因此,我们在两个外部和独立的WTS队列上应用了CNN11算法变量。使用发现集得出的最优截点在WTS TCGA队列上,高eTILs%的患者与低eTILs%组相比,OS有显著改善(eTILs%:HR = 0.09,95% CI = 0.01–0.70,P = 0.02)。
同样,高etTILs%或高eaTILs (mm2)的患者也表现出显著的有利结果[etTILs%:HR = 0.10,95% CI = 0.01–0.80,P = 0.03;eaTILs (mm2):HR = 0.10,95% CI = 0.01–0.76,P = 0.03](图4)。

在WTS Sweden队列中,只有easTILs与OS显著相关(easTILs:HR = 0.54,95% CI = 0.31–0.92,P = 0.02;补充图S4)。瑞典队列中有更多的高期患者,这可能是导致五个TIL变量算法性能不同的原因。未来的研究将评估这些变量与肿瘤期之间的相互作用。
eTILs%和etTILs%变量与临床结果显著相关,在三个验证集中得到了验证。同时,esTILs,eaTILs (mm2)和easTILs变量在两个验证集中得到了验证(补充表S3)。当将所有验证集合并为一个单一队列时,所有五个TIL变量与OS显著相关,无论是否根据多变量Cox回归分析调整了分期状态、年龄和病理学分级(表2)。
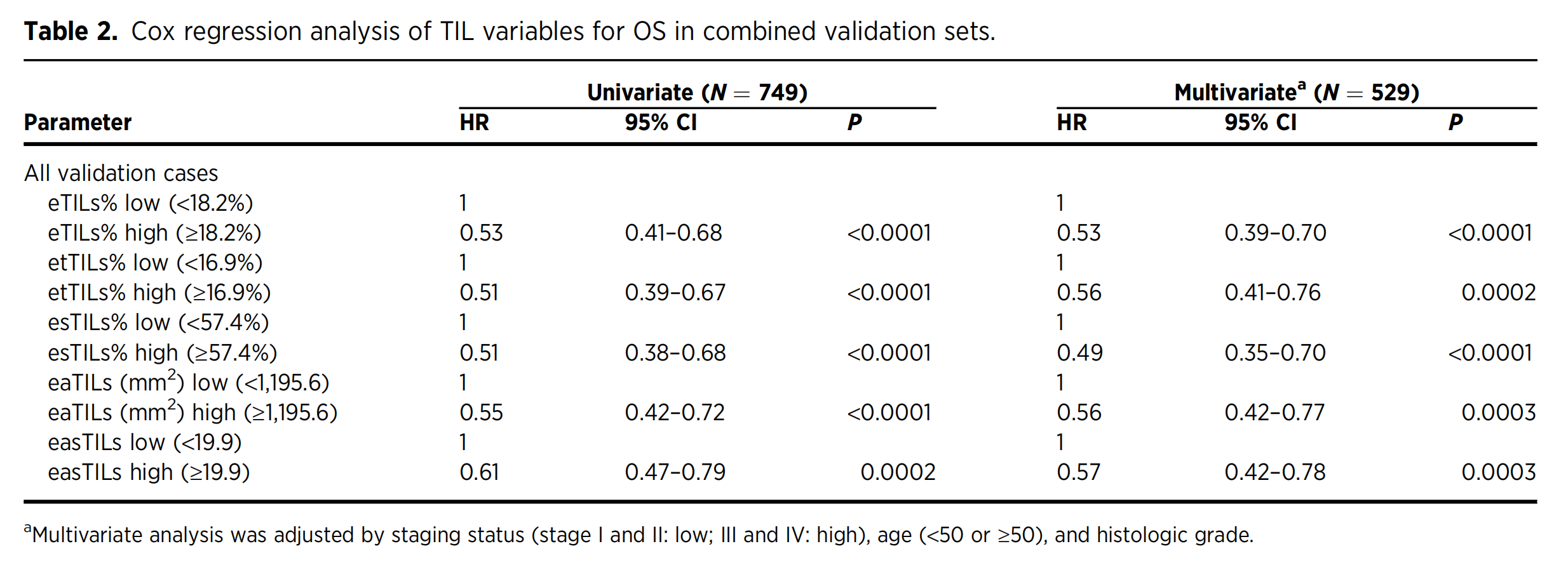
多变量Cox回归模型分析是为了测试机器读取算法变量的独立预后潜力,调整了分期、年龄和病理学分级。所有CNN11衍生的TIL算法变量在所有比较中均保持显著,具有相似的HR和重叠的CI值(HR < 0.57,95% CI = 0.35–0.78,P ≤ 0.0003,对于所有比较;表2)。
值得注意的是,eTILs%和esTILs%似乎是两个更稳健的标记(eTILs%:HR = 0.53,95% CI = 0.39–0.70,P < 0.0001;esTILs%:HR = 0.49,95% CI = 0.35–0.70,P < 0.0001;表2)。尽管从统计学上讲,比较P值是不合理的,但危险比可以进行比较。如表2所示,五个TIL变量的危险比相似,但eTIL%和esTIL%在所有比较中始终表现更好。
进一步分析WTS Sweden队列中病理学家读取的sTIL评估,发现高sTILs的患者比低sTILs的患者有更好的结果(HR = 0.52,95% CI = 0.30–0.90,P = 0.02)。
这一观察结果提出了一个关于比较TIL算法变量与病理学家读取的问题。当我们比较CNN11衍生的easTILs变量评分与病理学家读取的sTIL评估时,在WTS Yale(斯皮尔曼r系数 = 0.61,P < 0.0001)和WTS Sweden队列(斯皮尔曼r系数 = 0.63,P < 0.0001)中都观察到了良好的相关性。进一步分析显示,高sTILs的患者在这两个队列中easTILs显著更高(补充图S5)。
最后,我们还比较了CNN11衍生变量在TMAs与WTS上的表现。我们发现TMAs与WTS样本之间存在中等的相关性(补充表S4;补充图S6)。
综上所述,CNN11算法在多个独立队列中表现出良好的预后预测能力,且与病理学家读取的sTIL评估具有较好的相关性。这些发现支持了CNN11算法在临床实践中的应用潜力,尤其是在评估TNBC患者的预后和治疗反应方面。未来的研究需要进一步验证这些算法变量的临床效用,并探索它们与其他临床参数和治疗策略的相互作用。
四、讨论
近年来,TILs被认为在早期或转移性TNBC患者中具有重要的预后和预测价值(参考文献1, 4–8)。然而,病理学家读取的TIL评估可能会产生显著的变异性(参考文献14, 15)。
此外,由于难以进行此类评估,相邻的微环境细胞群体并未被考虑在内。为了量化TILs,计算研究要么专注于模仿国际免疫肿瘤生物标志物工作组引入的指南(参考文献16–18, 20),要么生成黑箱算法(参考文献19, 27)。我们用于定义细胞类型的算法(CNN11)同样是一个黑箱。然而,用于预后生物标志物的输出变量是基于透明细胞类型计算的,因此对病理学家更为熟悉。
在过去几年中,提出了不同的机器学习方法来评分抗肿瘤免疫力,从而产生了多种具有潜在临床应用价值的TIL生物标志物。其中一些机器学习工具基于补丁分类,而其他一些主要依赖于对象(细胞)检测/分类。
另一种广泛采用的方法是实现组织模式识别,区分组织区域,并在不同的组织隔室中评估TILs(例如,在肿瘤内基质;参考文献16–20)。然而,许多基于机器学习的TIL生物标志物缺乏广泛的验证,这对于临床采用是必要的。此外,需要进行比较各种TIL变量的研究,重点关注不同的空间方面。在本研究中,我们展示了基于机器学习的TIL评分能够提供TNBC肿瘤微环境中TILs的全面信息,这些信息难以由病理学家评估得出。
在我们的研究中,使用QuPath平台,我们开发了一个细胞分类器来评分TIL测量,产生了五个TIL变量[eTILs%,etTILs%,esTILs,eaTILs (mm2),和easTILs],这些变量代表了TILs与整个肿瘤区域内细胞计数之间的关系,以及不同肿瘤区域(例如,肿瘤内基质)的面积。
我们使用的数字分析方法是一种无监督的核分割,然后是神经网络为基础的机器学习细胞分类。这种概念的优势在于它需要相对较小的训练集。然而,它的局限性在于分割敏感性和分类性能依赖于生物和技术的图像变异,这可能导致过拟合分类器。
为了解决这个问题,我们在训练集上验证了分类器。为了处理这个问题,我们在四个独立的TNBC队列中进行了验证,这些队列来自不同的机构,诊断时间不同,格式也不同,既包括TMA也包括WTS格式。此外,在我们的评估协议中,我们保持了病理学家在选择肿瘤和相关基质以及排除灾难性的算法失败方面的作用。
我们的结果显示,所有五个TIL变量在独立TMA和WTS集合中的成功验证中具有预后作用。我们在黑色素瘤(参考文献21)中观察到eTIL%的类似预后结果。
尽管在本试点研究中尚未得到证实,但客观TIL评估的潜在临床用途可能是使临床医生能够识别可能受益于免疫治疗(参考文献28)或化疗可以省略的患者群体,其中复发疾病的发生率极低(参考文献29)。
尽管我们在独立队列中验证了具有预定义截点的机器读取TIL评分,但这些截点在一般情况下可能不适用,因为它们没有调整阶段、肿瘤类型和治疗类型。因此,我们建议将这种机器读取评分视为一个连续变量,并且需要进行调整上述因素的临床效用研究,以开发特定指征的具体截点。
此外,国际免疫肿瘤生物标志物工作组尚未推荐一个普遍适用的TIL阈值用于临床实践;然而,在最近发表的研究中,sTIL对预后有影响的阈值在10%至30%之间(参考文献5, 12)。在我们的验证集中,应用发现集(WTS Yale集合)中得出的截点后,我们发现在同一大致范围内找到了最优的预后截点,但变异范围更窄,并且观察者独立可重复。
本试点工作存在一些潜在的局限性。
我们工作的一个局限性是算法分配错误作为机器读取评分的一个潜在陷阱。例如,在含有凋亡形态、中性粒细胞和具有单调均匀核的低级别肿瘤的区域中,检测到假阳性TILs。
此外,我们的模型没有细调以区分iTILs和sTILs,因为iTILs在大多数情况下只占TILs的1%至3%。这些错误在罕见情况下可能是灾难性的,需要病理学家审查病例以排除或重新分析。
然而,病理学家细胞分配审查验证是过程中的合理步骤,因为我们认为计算算法在短期内将协助,而不是取代病理学家。我们的目标不是构建一个完全自动化的TIL评分应用程序,而是一个计算机辅助、开源的工具,可能有助于病理学家提高可重复性。因此,病理学家对于质量控制和系统性能评估仍然至关重要。
本工作的最显著局限性是所有队列都是回顾性收集的。因此,它们具有异质的治疗方法和可变纳入标准。此外,组织处理和H&E染色的差异可能会进一步导致队列之间的变异性。然而,机器读取的TIL评分在验证集中的预后表现尽管队列之间的变异,进一步支持了其稳健性。在本研究中,预后关联未特别考虑给予的治疗,因为不同队列之间的治疗方式不同。
为了真正评估预后价值,临床试验的严谨性是最好的。然而,临床试验的组织和数据是非常珍贵和有限的资源。因此,需要使用回顾性队列进行概念证明的试点、发现研究,如本研究。
最后,需要提到的一个变量,尽管不一定是局限性,但瑞典WTS队列使用的是不同的幻灯片扫描仪品牌(NanoZoomer,而不是Aperio)。不可能重新扫描所有瑞典队列使用Aperio扫描仪,而且超出本工作的范围比较不同幻灯片扫描仪对最终结果的影响。
然而,这个变量可能会产生变异,应被视为本工作的潜在局限性,并应在瑞典队列中考虑结果的变异。未来的努力将比较幻灯片扫描硬件。
总的来说,我们证明了基于机器学习的TIL算法变量与TNBC患者的预后显著相关。它们也在几个验证队列中显示出客观和独立的预后因素。随着在临床试验中的进一步研究,我们相信这个客观工具可以在临床环境中用于客观量化TILs。
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








