小罗碎碎念
本期推文主题:胃癌
这期推文时间跨度比较大,包括了24年1月~8月发表的一系列文献,其中重点推荐第五篇文献(作者来自北京大学),涉及到了影像组学、病理组学和临床信息的整合,并且代码开源,有兴趣的可以一起研究交流。

本期推文代码&数据集
第二篇
- 数据集:
- TCGA-STAD: 文章中提到了使用TCGA(The Cancer Genome Atlas)项目的胃癌(Stomach Adenocarcinoma,简称STAD)数据集。
- ENCODE: 文章提到了使用ENCODE(Encyclopedia of DNA Elements)项目的数据。ENCODE是一个公共项目,旨在识别基因组中的所有功能元素,数据可通过ENCODE门户网站获取。
- 代码和分析工具:
- gkm-SVM: 文章中提到了使用gkm-SVM(gapped k-mer support vector machine)方法进行机器学习分析。这是一种用于序列分析的机器学习模型,通常用于基因组数据的模式识别。
第三篇
-
代码链接:
- GitHub 链接: https://github.com/Yangzi-Chen2023/GC_NC-Res
- Code Ocean 链接: https://codeocean.com/capsule/5496369/tree
-
数据集链接:
- 数据集链接: Supplementary Data 1
这个链接指向的是文章的补充材料中的原始数据集。数据集是进行代谢组学分析的基础,包含了用于训练和验证机器学习模型的血浆样本的代谢物数据。公开数据集有助于提高研究的透明度和可重复性,同时也允许其他研究者使用相同的数据进行新的分析或与自己的数据进行比较。
第五篇
- 代码链接:
- MuMo模型的源代码:https://github.com/czifan/MuMo
- 这是文章中提出的多模态模型(MuMo)的代码库,用于预测HER2阳性胃癌患者对抗HER2疗法或抗HER2联合免疫疗法的反应。在文章中,这个代码库是实现和验证研究方法的关键,允许其他研究人员复现和利用该模型。
第六篇
-
代码链接:
- GitHub 链接: https://github.com/baofengguat/RFLM-project/tree/master
- 作用:代码库提供了实现鲁棒联合学习模型(RFLM)所需的源代码。这使得其他研究人员可以复现研究结果,验证模型的有效性,并在此基础上进行进一步的研究和开发。
-
数据集:
- LIDC-IDRI 数据集:https://wiki.cancerimagingarchive.net/display/Public/LIDC-IDRI 这是一个公开的肺癌诊断数据集,由肺影像数据库联合会(LIDC-IDRI)提供。在文章中,该数据集被用来展示 RFLM 算法在其他任务(如肺癌诊断)中的适用性。
一、胃癌治疗耐药性的空间多组学研究

一作&通讯
| 作者类型 | 作者姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Gang Che | 浙江大学医学院附属第一医院 浙江省胰腺疾病重点实验室 |
| 第一作者 | Jie Yin | 浙江省肿瘤医院 结直肠肿瘤科 中国科学院杭州医学研究所 |
| 通讯作者 | Chao Wu | 浙江大学医学院附属第一医院 浙江省胰腺疾病重点实验室 |
| 通讯作者 | Jianpeng Sheng | 重庆大学 国家教育部指定智能肿瘤学中心 重庆大学附属肿瘤医院 重庆大学医学院 |
| 通讯作者 | Qing Li | 重庆大学 国家教育部指定智能肿瘤学中心 重庆大学附属肿瘤医院 重庆大学医学院 |
| 通讯作者 | Jian Liu | 浙江大学医学院附属第一医院 浙江省胰腺疾病重点实验室 |
文献概述
这篇文章通过多组学和空间数据分析,探索了胃癌对化疗和免疫治疗反应的异质性,并揭示了药物耐药性的细胞和分子机制。
研究的目的是探索胃癌对化疗和免疫治疗的异质性反应,并试图通过空间多组学方法来阐明药物耐药性的细胞和分子基础。
研究方法包括:
- 对接受化疗和免疫治疗的胃癌患者的术后组织进行多组学探索。
- 开发图像深度学习模型来预测治疗反应。
研究结果发现:
- 顶端膜细胞与对氟尿嘧啶和奥沙利铂(治疗的关键成分)的耐药性相关。
- 这些细胞与驻留巨噬细胞之间的大量相互作用,强调了细胞间通信在塑造治疗耐药性中的作用。
- 配体-受体分析揭示了特定的分子对话,如 TGFB1-HSPB1 和 LTF-S100A14,为耐药性潜在的信号通路提供了见解。
- 支持向量机(SVM)模型结合多组学和空间数据,显示出显著的预测能力。
研究结论:
- 通过整合 mIHC 测定、特征提取和机器学习,成功揭示了药物耐药性背后的复杂细胞相互作用。
- 这一预测模型可能作为个性化治疗策略和提高胃癌治疗结果的有价值工具。
文章还提到了研究的局限性,包括样本大小有限,以及需要在更大的队列和不同的治疗方式中验证发现。
二、机器学习识别RUNX/AP-1在胃癌中的关键调控作用

一作&通讯
| 角色 | 姓名 | 单位名称(中文) | 单位名称(英文) |
|---|---|---|---|
| 第一作者 | Milad Razavi-Mohseni | 约翰霍普金斯大学,生物医学工程系和遗传医学系 | Department of Biomedical Engineering and McKusick-Nathans Department of Genetic Medicine, Johns Hopkins University |
| 通讯作者 | Anders J Skanderup | 新加坡基因组研究所,计算癌症基因组学实验室 | Laboratory of Computational Cancer Genomics, Genome Institute of Singapore (GIS), Agency for Science, Technology and Research (A*STAR) |
| 通讯作者 | Michael A Beer | 约翰霍普金斯大学,生物医学工程系和遗传医学系 | Department of Biomedical Engineering and McKusick-Nathans Department of Genetic Medicine, Johns Hopkins University |
文献概述
这篇文章通过机器学习分析揭示了在胃癌中,RUNX/AP-1的激活驱动了间充质和纤维化调控程序,对胃癌异质性和进展具有潜在的驱动作用。
文章通过机器学习方法,利用ATAC-seq和RNA-seq数据,研究了胃癌细胞系和原发性肿瘤中调控状态变化及其转录反应的驱动因素。
研究发现,与上皮型(Epi-like)相比,间充质型(Mes-like)胃癌亚型更具侵袭性。研究者们开发了一种机器学习方法来确定调控胃癌亚型的转录因子(TFs),并识别了驱动间充质状态的TFs(如RUNX2、ZEB1、SNAI2、AP-1二聚体)以及上皮状态的TFs(如GATA4、GATA6、KLF5、HNF4A、FOXA2、GRHL2)。此外,研究还发现了与这些TFs失调相关的DNA拷贝数变化,特别是GATA4的缺失和MAPK9的扩增。
通过与bulk和单细胞RNA-seq数据集的比较,研究发现Mes-like GC中向成纤维细胞样表观基因组和表达特征的激活。这种间充质纤维化程序的激活与间充质基因旁的DNA顺式调控元件的差异化可及性相关。研究结果为胃癌中TF活性提供了一幅地图,并强调了拷贝数驱动的改变在塑造表观遗传调控程序中的潜在作用,这些程序可能是胃癌异质性和进展的驱动因素。
文章还讨论了胃癌的流行病学、分子亚型、以及与治疗反应相关的基因表达特征。此外,研究者们还探讨了通过个性化治疗反应来确定不同治疗方法的疗效,并研究了转录因子在胃癌侵袭性中的作用。研究结果为理解胃癌的异质性和侵袭性提供了新的见解,并可能为胃癌的治疗提供新的靶点。
三、利用机器学习揭示胃癌代谢特征并构建诊断预后模型

一作&通讯
| 角色 | 姓名 | 单位名称(中文) | 单位名称(英文) |
|---|---|---|---|
| 第一作者 | Yangzi Chen | 清华大学药学院 | School of Pharmaceutical Sciences, Tsinghua University |
| 通讯作者 | Zeping Hu | 清华大学药学院 & 清华大学-北京大学生命科学联合中心 | School of Pharmaceutical Sciences, Tsinghua University & Tsinghua-Peking Joint Center for Life Sciences |
Zeping Hu是清华大学药学院的教授,同时在清华大学-北京大学生命科学联合中心担任职务。他在药物化学和生物医学研究领域有着深入的研究,特别是在药物发现、药物作用机制以及疾病治疗策略等方面做出了重要贡献。
他的研究工作通常涉及跨学科的合作,包括化学、生物学、药理学和临床医学等多个领域。Zeping Hu教授在国际学术期刊上发表了多篇研究论文,并获得了多项科研资助和奖项,是该领域内具有重要影响力的科学家之一。
文献概述
这篇文章通过靶向代谢组学分析和机器学习技术,开发了用于胃癌早期诊断和预后预测的新型生物标志物模型。
研究团队分析了来自多个中心的702个血浆样本,通过靶向代谢组学分析,揭示了胃癌的代谢特征。他们利用机器学习算法,特别是最小绝对收缩和选择算子(LASSO)回归和随机森林模型,从数据中筛选出10种代谢物,构建了一个胃癌诊断模型(10-DM模型)。这个模型在外部测试集上的敏感性达到了0.905,超过了传统的癌症蛋白标志物检测方法。
此外,研究者还开发了一个预后模型(28-PM模型),它利用机器学习分析181名胃癌患者的代谢组学数据,以预测临床结果。与传统的临床参数模型相比,这个预后模型显示出更高的准确性,并且能够有效地将患者分为不同的风险组,以指导精确的干预措施。
文章强调了代谢组学在揭示胃癌代谢特征和识别潜在治疗靶点方面的应用,并指出机器学习在解释复杂组学数据、开发预测模型和精确医学中的潜力。研究结果不仅揭示了胃癌的代谢特征,而且识别了两个不同的生物标志物组,分别用于早期检测和预后预测,从而有助于胃癌的精准医疗。
四、泛癌分析揭示无细胞免疫相关miRNAs作为早期癌症检测的生物标志物

一作&通讯
| 作者角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | 吴 | 中国医学科学院北京协和医学院肿瘤医院 胸外科 |
| 第一作者 | 张 | 中国医学科学院北京协和医学院肿瘤医院 胸外科 |
| 第一作者 | 唐 | 中国医学科学院北京协和医学院肿瘤医院 胸外科 |
| 通讯作者 | 孙 | 中国医学科学院北京协和医学院肿瘤医院 胸外科 |
| 通讯作者 | 何 | 中国医学科学院北京协和医学院肿瘤医院 胸外科 |
文献概述
这篇文章报道了一项基于大规模样本的泛癌分析研究,开发了一种新的无细胞免疫相关微小RNA(cf-IRmiRNAs)签名,用于早期癌症的非侵入性检测。
研究分析了来自15,832名参与者的循环miRNA(微小RNA)档案,包括患有13种癌症类型的个体和对照组。数据被随机分为训练集、验证集和测试集(比例为7:2:1),还有一个额外的外部测试集包括684名参与者。
在发现阶段,研究者们鉴定了100个在恶性肿瘤和非恶性之间差异表达的cf-IRmiRNAs,并通过最小绝对收缩和选择算子(LASSO)方法保留了39个。研究采用了五种机器学习算法来构建cf-IRmiRNAs签名,基于XGBoost算法的诊断分类器在验证集上显示出卓越的癌症检测性能(AUC:0.984,CI:0.980–0.989),这是通过5折交叉验证和网格搜索确定的。
在测试集和外部测试集的进一步评估确认了分类器的可靠性和有效性(AUC:0.980至1.000)。该分类器成功检测到早期癌症,特别是肺癌、前列腺癌和胃癌。它还能区分良性和恶性肿瘤。这项研究代表了迄今为止最大规模和最全面的关于cf-IRmiRNAs的泛癌分析,提供了一个有前景的非侵入性诊断生物标记物,用于早期癌症检测,并可能对临床实践产生影响。
文章还讨论了癌症作为严重的公共健康问题,以及早期检测对于改善治疗结果的重要性。研究指出,尽管治疗取得了进步,但癌症的预后仍然不容乐观。因此,寻找实用的、微创的方法对于早期癌症检测至关重要。无细胞miRNAs(cf-miRNAs)因其稳定性和丰度而被视为液体活检标记物的有希望的候选者。研究还探讨了炎症反应和生物标记物可能在癌症诊断前几年就出现,并且慢性炎症导致的免疫抑制微环境可能促进癌症的发展和激活。
文章最后提到,尽管这项研究基于最大的样本量队列揭示了cf-IRmiRNAs在癌症检测中的价值,并突出了cf-IRmiRNAs小组在准确非侵入性检测早期恶性肿瘤方面的潜力,但鉴于研究的回顾性设计,需要在大规模前瞻性和多中心试验中进一步验证。
五、多模态数据分析在预测HER2阳性胃癌治疗反应中的应用

一作&通讯
| 作者类型 | 作者姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Zifan Chen | 北京大学数据科学中心 |
| Yang Chen | 北京大学肿瘤医院胃肠肿瘤科 | |
| Yu Sun | 北京大学肿瘤医院病理科 | |
| Lei Tang | 北京大学肿瘤医院放射科 | |
| 通讯作者 | Xiaotian Zhang | 北京大学肿瘤医院胃肠肿瘤科 |
| Bin Dong | 北京大学数学科学学院;北京国际数学研究中心 | |
| Lin Shen | 北京大学肿瘤医院胃肠肿瘤科 |
通讯作者介绍:
- Xiaotian Zhang:张晓天,北京大学肿瘤医院胃肠肿瘤科的研究人员,专注于胃癌等消化道肿瘤的治疗和研究。在胃癌的抗HER2治疗和免疫治疗方面有深入的研究和丰富的临床经验。
- Bin Dong:董彬,北京大学数学科学学院教授,北京国际数学研究中心成员,主要研究领域包括机器学习和人工智能在医学图像分析和生物信息学中的应用。
- Lin Shen:申琳,北京大学肿瘤医院胃肠肿瘤科的研究人员,专注于胃癌的临床治疗和转化研究,尤其在胃癌的个体化治疗和新型治疗策略的开发方面有显著贡献。
文献概述
这篇文章通过应用多模态数据分析和深度学习模型MuMo,成功预测了HER2阳性胃癌患者对抗HER2疗法或联合免疫疗法的反应,并可能有助于个性化医疗的实施。
研究使用了多模态数据,包括放射学、病理学和临床信息,来自429名患者的数据。研究团队引入了一个深度学习模型,称为多模态模型(MuMo),整合这些数据以精确预测治疗反应。
MuMo在预测抗HER2疗法和联合免疫疗法的反应方面表现出色,分别达到了0.821和0.914的曲线下面积(AUC)得分。此外,MuMo将患者分类为低风险,这些患者的无进展生存期和总生存期显著延长。
研究还强调了多模态数据分析在增强HER2阳性胃癌治疗评估和个性化医疗中的重要性,以及该模型的潜力和临床价值。研究结果不仅突出了多模态数据分析在增强治疗评估和个性化医疗中的重要性,还展示了该模型的潜力和临床价值。
研究还进行了一些消融研究,以评估MuMo融合模块对多模态信息的贡献。研究结果表明,整合放射学和病理学数据提高了模型的预测AUC得分。此外,包括临床报告和患者特定信息在内的综合数据显著增强了MuMo的预测能力。
最后,研究还探讨了MuMo的可解释性,包括图像聚焦区域的定性分析和临床信息权重的定量分析。MuMo能够从临床报告和患者信息中提取适当的知识,以准确预测治疗反应,其识别的关键决策变量与当前的临床发现一致,证实了MuMo的可靠性和临床相关性。
重点关注
Fig. 5中,展示了多模态模型(MuMo)的可解释性分析。
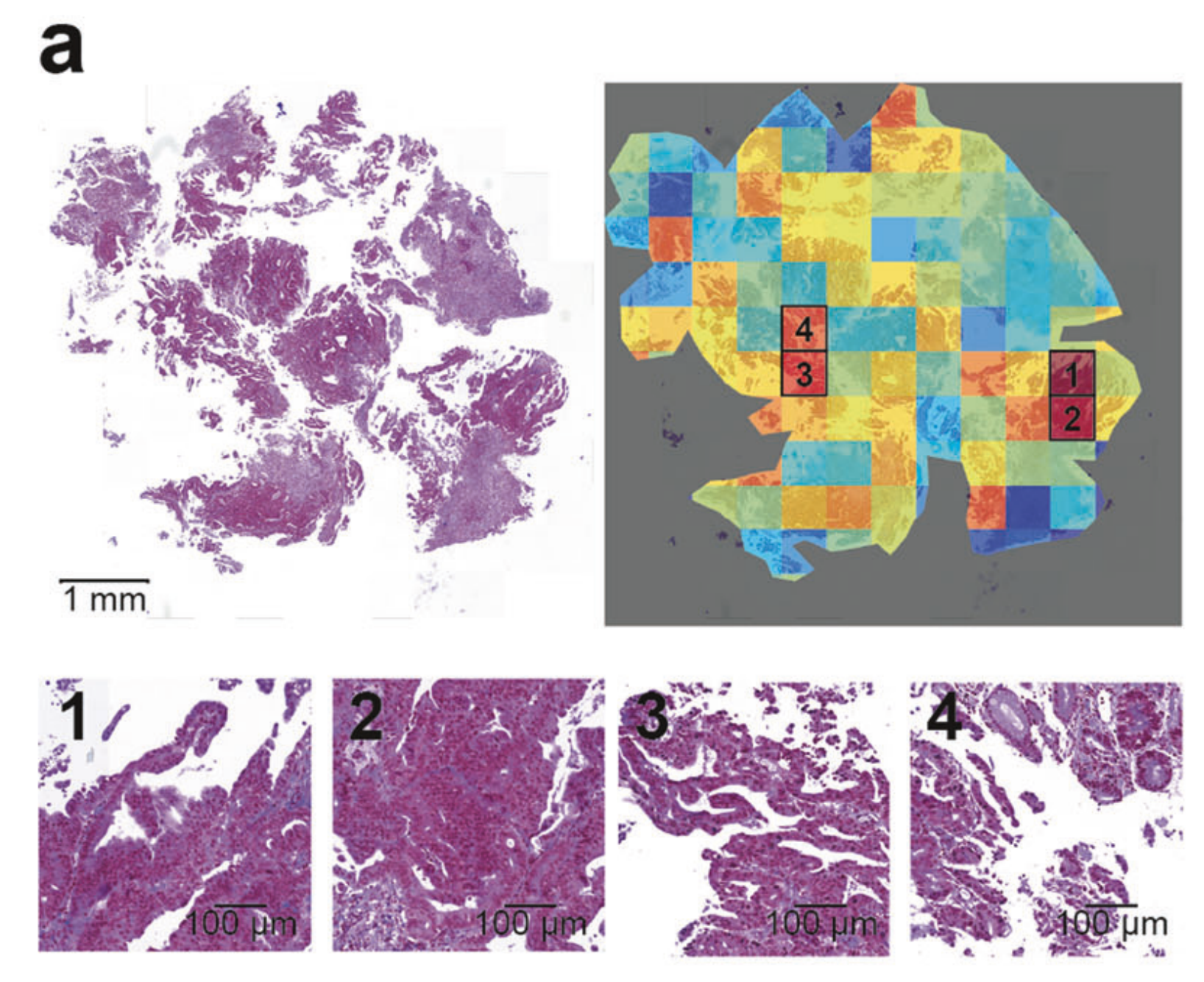
a. 病理切片图像中“bags”的重要性得分可视化:
- “Bags”是指在病理全切片图像中划分的较大区域,这些区域在进行病理分析时被特别关注。
- 图像中颜色的深浅代表了模型在进行治疗反应预测时,各个区域的重要性。红色区域表示这些区域对预测结果的贡献较大,而蓝色区域则表示贡献较小或影响较小。
- 第二行显示了切片图像中四个最重要的“bags”,这表明这些区域在模型的预测中起到了关键作用。
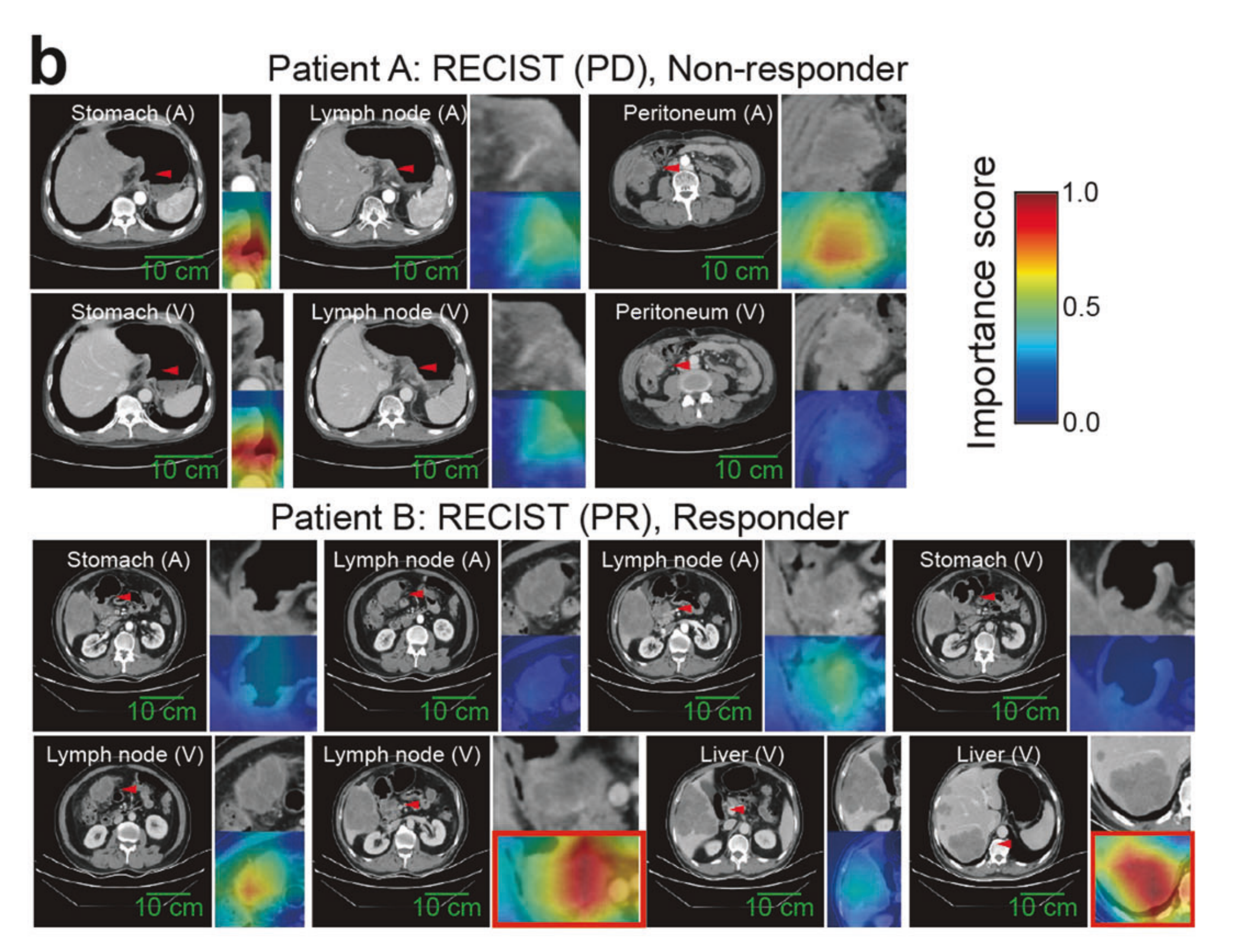
b. 放射学病变图像上的注意力图可视化(使用Grad-CAM算法):
- Grad-CAM(Gradient-weighted Class Activation Mapping)是一种用于可视化深度学习模型中哪些区域对最终决策影响最大的技术。
- 在这里,红色区域表示MuMo模型在分析时特别关注的区域,如淋巴结和肝脏肿瘤,这些区域对治疗反应的预测至关重要。
- 蓝色区域则表示模型关注较少的区域。
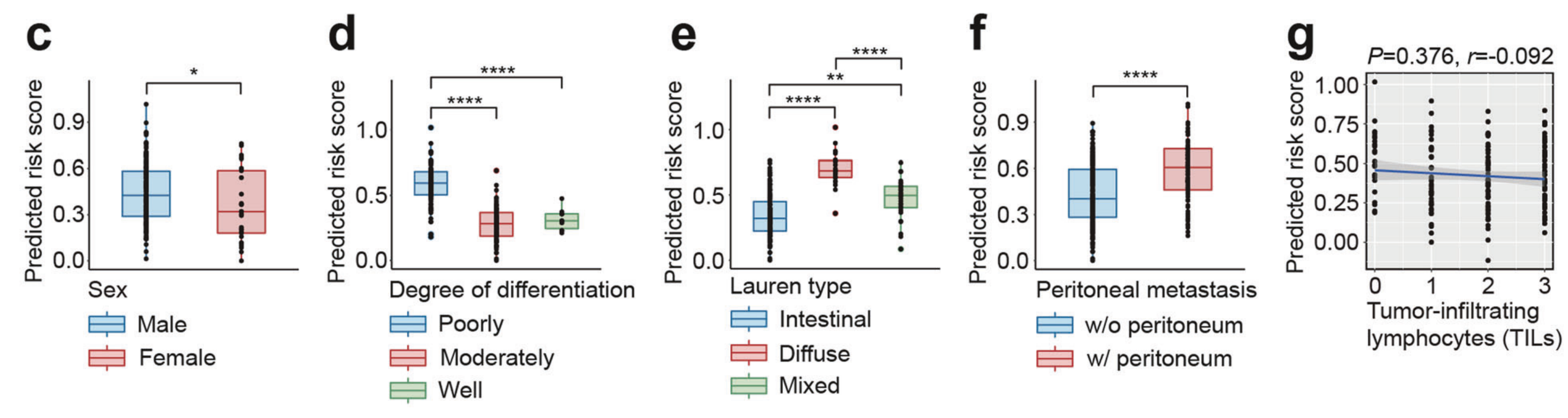
c–g. 抗HER2队列中不同临床信息亚组的预测风险评分评估:
- 这部分展示了在抗HER2治疗组中,根据临床信息(如性别、肿瘤分化程度、Lauren分类、腹膜转移等)对患者进行分组,并评估了模型预测的风险评分。
- 这些评估有助于理解模型是如何根据临床信息来预测治疗反应的,以及哪些因素对预测结果影响较大。
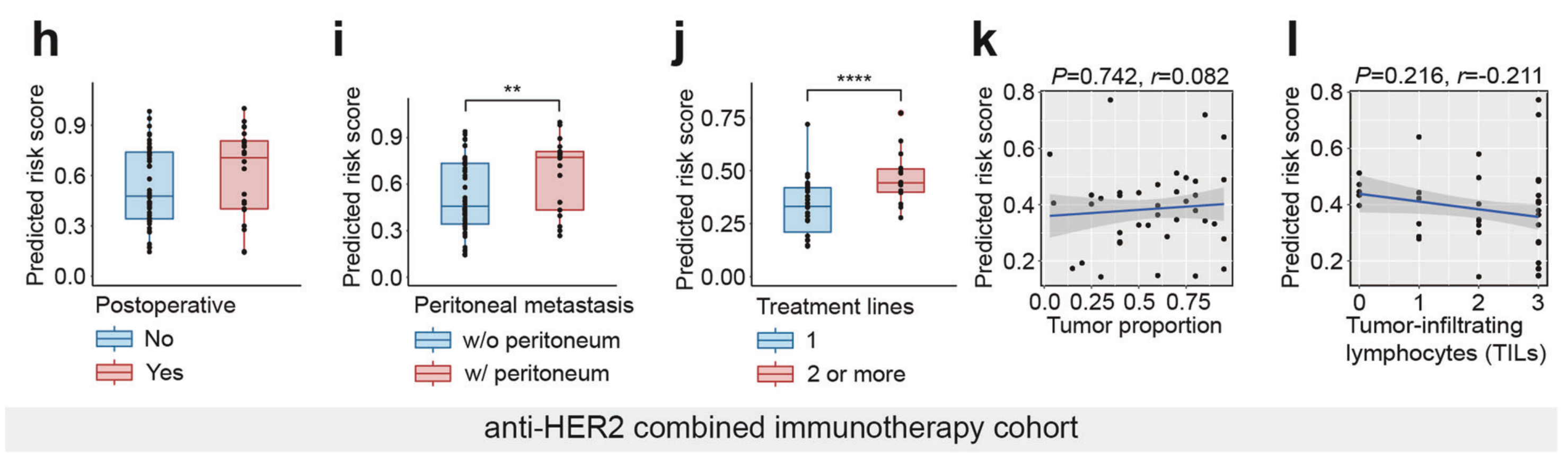
h–l. 抗HER2联合免疫治疗队列中不同临床信息亚组的预测风险评分评估:
- 与c–g部分类似,这一部分针对的是接受了抗HER2联合免疫治疗的患者群体。
- 通过评估不同临床信息亚组的预测风险评分,可以了解模型在这种联合治疗方案中的预测能力和准确性。
总体而言,Fig. 5通过可视化和评估的方式,展示了MuMo模型在处理病理和放射学数据时的关注点,以及如何结合临床信息来预测胃癌患者对抗HER2治疗或联合免疫治疗的反应。这不仅展示了模型的预测能力,也增强了模型的可解释性,有助于临床医生理解和信任模型的预测结果。
六、基于鲁棒联合学习的胃癌术后复发高危患者识别

一作&通讯
| 作者类型 | 作者姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Bao Feng | 江门市中心医院放射科,中国 |
| 第一作者 | Jiangfeng Shi | 桂林航天工业学院智能检测与信息处理实验室,中国 |
| 通讯作者 | Yehang Chen | 桂林航天工业学院智能检测与信息处理实验室,中国 |
| 通讯作者 | Wansheng Long | 江门市中心医院放射科,中国 |
文献概述
这篇文章介绍了一种鲁棒的联合学习模型(Robust Federated Learning Model, RFLM),用于在保护隐私的同时,通过多中心数据识别胃癌术后复发的高危患者。
- 研究背景:胃癌是最常见的恶性肿瘤之一,许多患者在晚期才被诊断出来。尽管手术切除是晚期胃癌的主要治疗方法,但由于复发率高,患者的生存率并不理想。目前使用的TNM分期系统虽然对治疗规划和预后评估很重要,但它缺乏关于肿瘤相关因素和肿瘤边缘特征的信息,这些信息对于预测胃癌患者的术后复发至关重要。
- 研究目的:本研究旨在建立一个鲁棒的联合学习模型,以克服数据孤岛问题,并在多中心、跨机构的环境中识别胃癌术后复发的高危患者,从而实现具有显著价值的稳健治疗。
- 数据收集:研究从四个独立的医疗机构收集数据进行实验。
- 方法:提出的RFLM算法在四个数据中心的接收者操作特征曲线下面积(AUC)值分别为0.710、0.798、0.809和0.869。此外,通过分析评估了算法的有效性,并识别了自适应和常见特征。
- 结果:RFLM在预测胃癌患者术后复发方面表现出更高的诊断效率。与临床模型相比,RFLM在所有四个中心的测试数据集上的整体准确性提高了32.36%,并且成功将胃癌局部复发的误诊率降低了42.23%。
- 与其他算法的比较:RFLM与其他四种传统的联合学习算法(FedAvg、FedProx、Moon、HarmoFL)以及两种鲁棒联合学习算法(pFedMe和pFedFSL)进行了比较。RFLM在所有四个中心中均实现了最高的AUC结果。
- 鲁棒性测试:通过在多个中心进行数据集分布的随机置换,验证了算法的鲁棒性。此外,通过三重交叉验证进一步验证了模型的鲁棒性。
- 特征分析:研究分析了RFLM的联合特征,包括共同特征和自适应特征。共同特征是指在有无胃癌复发的患者之间共享的类别特征,而自适应特征是指特定于不同数据中心的独特特征。
- 应用到其他任务:为了展示RFLM算法在其他任务中的适用性,研究还使用了来自肺影像数据库联合会(LIDC-IDRI)的肺癌诊断数据集进行了测试。
- 讨论:文章讨论了准确的预后评估在确定胃癌患者的个性化和精确治疗中的关键作用,并指出了TNM分期系统的局限性。此外,文章还讨论了RFLM在预测胃癌患者术后复发方面的有效性,并与其他联合学习算法进行了比较。
重点关注
图 4 提供了鲁棒联合学习模型(RFLM)结果的详细分析。
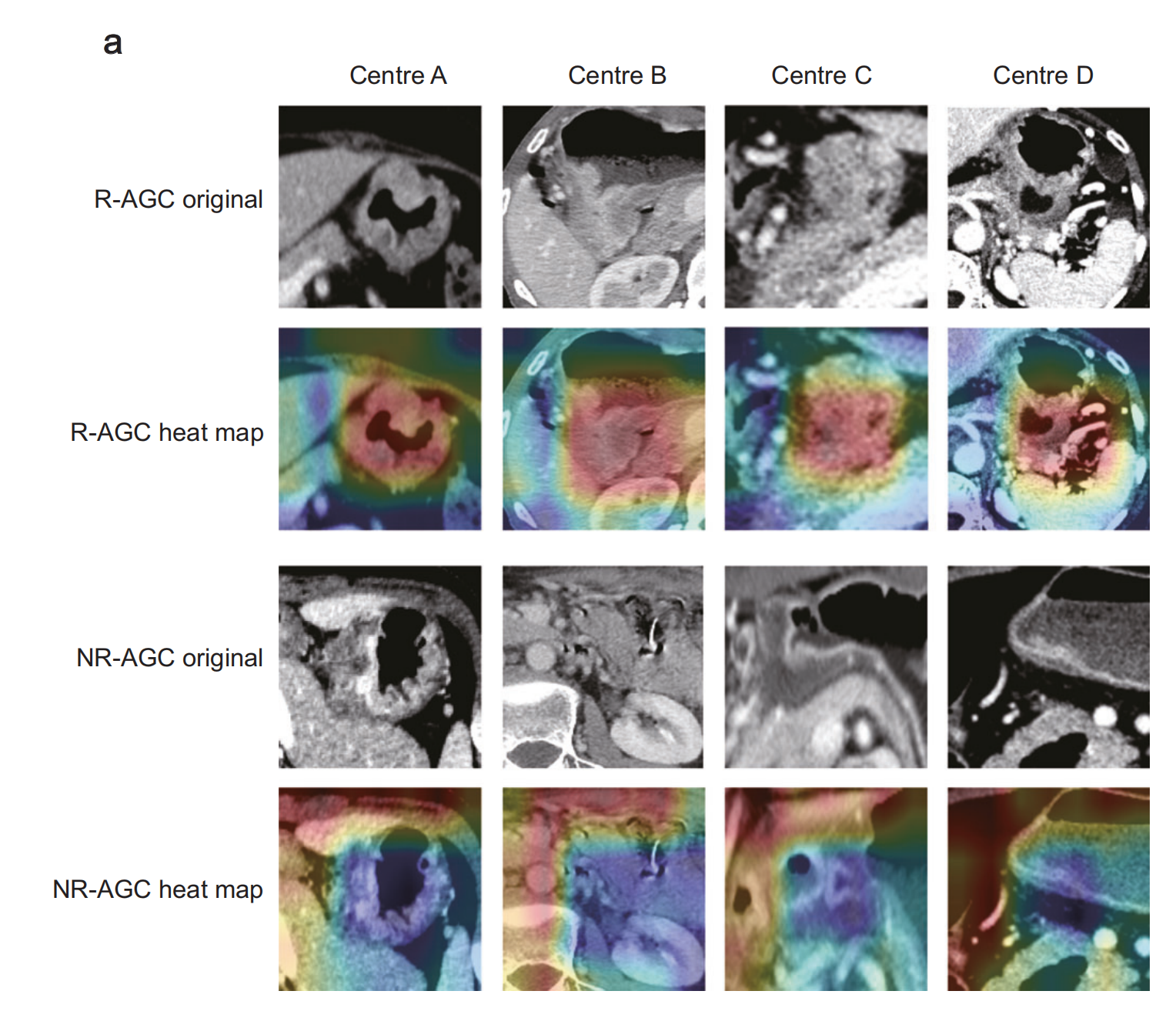
a部分:模型注意力热图
- 目的:此热图展示了 RFLM 对复发和非复发(NR-AGC)胃癌图像中的不同部分的关注程度。
- 红色区域:表明模型更加关注的区域,暗示这些区域在区分复发和非复发病例中更为关键。
- 蓝色区域:显示模型关注程度较低的部分,意味着这些部分在分类过程中的重要性较低。
- 含义:这种视觉表示有助于理解哪些特征或医学图像的哪些部分对于预测胃癌复发更为重要。
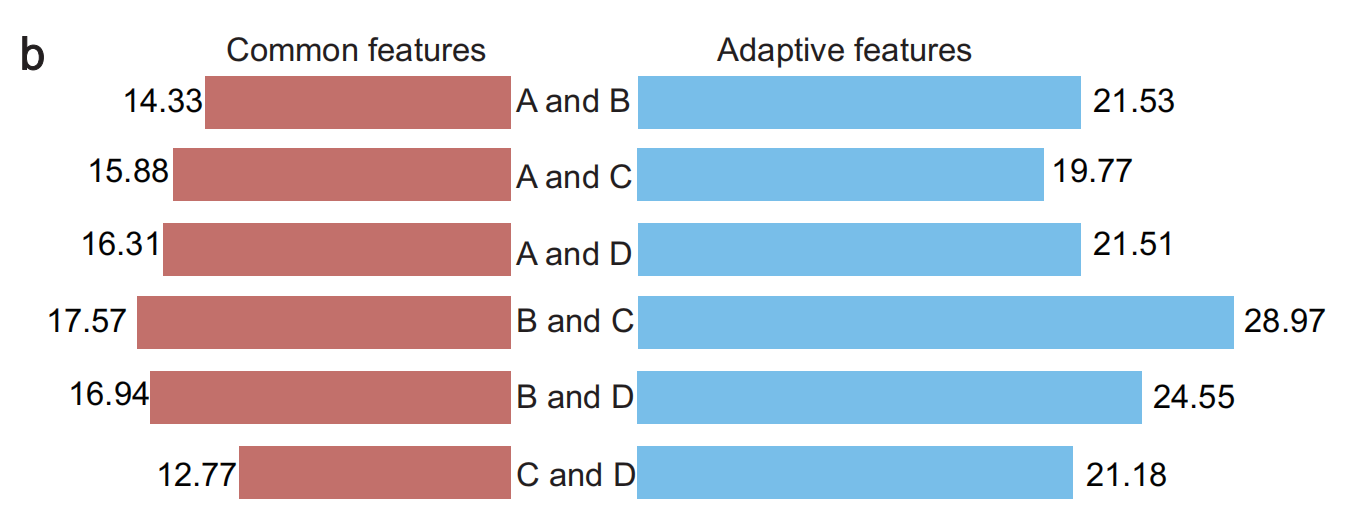
b部分:欧几里得距离图
- 目的:此图比较了四个中心数据点的共同特征和自适应特征之间的欧几里得距离。
- 共同特征(左侧):代表在不同数据中心一致的特征,表明它们在模型预测中的普遍重要性。
- 自适应特征(右侧):反映了特定于单个数据中心的特征,显示模型如何适应本地数据特征。
- 含义:这种比较有助于理解模型捕获全局(共同)和本地(自适应)模式的能力,这对于跨不同数据源的鲁棒联合学习至关重要。

c部分:得分图表
- 目的:这些图表展示了 RFLM 为四个数据中心的阳性(R-AGC)和阴性(NR-AGC)图像分配的得分。
- 阳性和阴性图像:得分有助于直观地展示模型如何区分复发和非复发胃癌图像。
- 统计检验:使用独立 t 检验(双尾)来统计验证两个类别得分之间的差异。
- 含义:这部分图表为模型的预测性能提供了定量评估,有助于确认 RFLM 在分类胃癌复发方面的有效性。
总体意义
图 4 对于展示 RFLM 在处理复杂、多源医学数据方面的有效性至关重要。它显示了模型不仅从数据中集体学习(共同特征),还适应本地数据的细微差别(自适应特征)。热图和得分图表以及统计验证为模型的性能提供了全面的视图,有助于确认其在预测胃癌复发方面的潜力。对于对 AI 驱动的诊断工具感兴趣的研究人员和临床医生来说,这个图尤其重要。


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








