第陆章 Overview of the next generation sequencing technology
6.1 What is Next Generation Sequencing Technology?
测序:把DNA上的核苷酸顺序测出来

第一代:
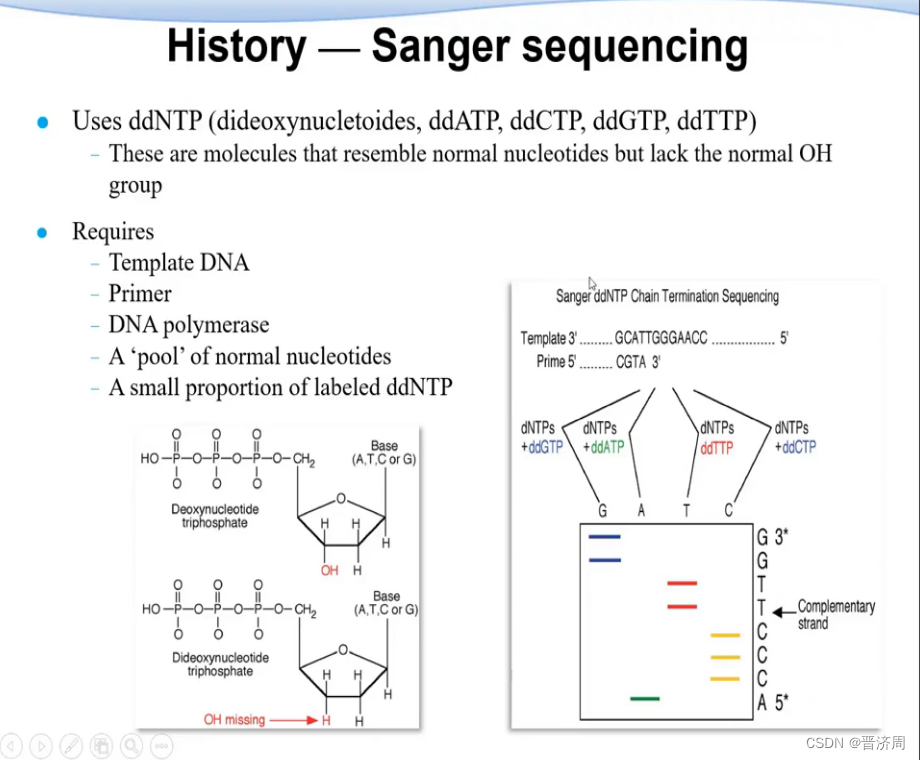

1. 二代测序技术简介

高通量 ; 序列短

6.2 Platform overview
6.2.1 Illumina Genome Analyzer
Illumina测序平台技术和测序原理

扩增技术: 桥式扩增
方法: 边合成边测序
扩增过程:
1:在样本里面获得DNA序列 ,序列两端接上接头(Adapters);
2: 固定在装置上,处理成单链DNA;
3: 把单链DNA变成桥式的结构,加入游离的四种碱基,把序列进行无限扩增;
4: 一次扩增后,将桥型竖直,再进行上述步骤进行扩增。
测序过程:边合成边测序
1:扩增过程结束以后,加上标记上颜色的四种碱基(A,C,G,T)
2: 边合成时,边用激光扫描,测其颜色和序列
3: 读出互补链的顺序,再通过碱基互补配对的原则,得出模板链的核苷酸顺序。
6.2.2 SOLiD: Sequence-by-ligation
SOLiD测序平台技术和测序原理
最大的区别
颜色的编码,SOLiD是双碱基编码,而Illumina是单碱基编码

过程:

人工合成
一个如下探针

一种颜色标识256个探针
前五个是正常的脱氧核苷酸,后面三个是通用碱基
前两个核苷酸探针决定标记的颜色是什么。
测序过程



一轮反应只知道颜色,而不知道碱基
设计引物,与接头进行碱基互补配对,加入1024种颜色探针,从待测序列的第一个到第八个bp,只有唯一一种颜色探针与其互补配对。
二次重复时,把引物往前错一位,把第一个位置留了下来,重复过程

优势
可以识别测序错误,可以分别是测序错误(只改变一个颜色)还是基因的突变(连续改变两个颜色),简单易操作
不足
工序繁琐
6.3 Biological applications
二代测序技术的生物学应用

测DNA
- 从头测序
- 基于参考基因组的重测序
- 宏基因组(微生物)
测RNA
- 基因的表达
- miRNA和一些新的非编码RNA
研究蛋白质和DNA/RNA相互作用
- ChIP-seq 转录因子和DNA结合位点的位置
- CLIP-seq RNA和蛋白质相互作用
表观遗传学
- DNA 碱基化
- 组蛋白修饰
- 染色质结构
- 核小体定位
DNA测序
- 全基因组的测序(人和动物)
- 癌症基因组研究
- 靶向基因组测序(只测基因组的一部分)
- 混合基因组测序
- 拷贝数变异
- 结构变异
RNA测序
- 测基因的表达量

6.4 Data processing workflow
1. 二代测序数据分析基本流程
类别

大小

流程

2. 二代测序数据的质量分数
测序的质量
- 比对的质量
- 碱基的质量
- 识别的质量
质量分数
 p是测错的概率,acc是成功的概率
p是测错的概率,acc是成功的概率
6.5 Sequence Alignments
1. 二代测序数据比对算法介绍
按照目的区分: 1:全局比对(从头比到尾); 2:局部比对(中间找到一个最佳匹配)
最佳匹配措施

比对上+1;没比对上+0;罚分:空位-3,扩展+0.1
算法设计


从矩阵的最后一个元素,往回回溯

如果有负分和0进行比对
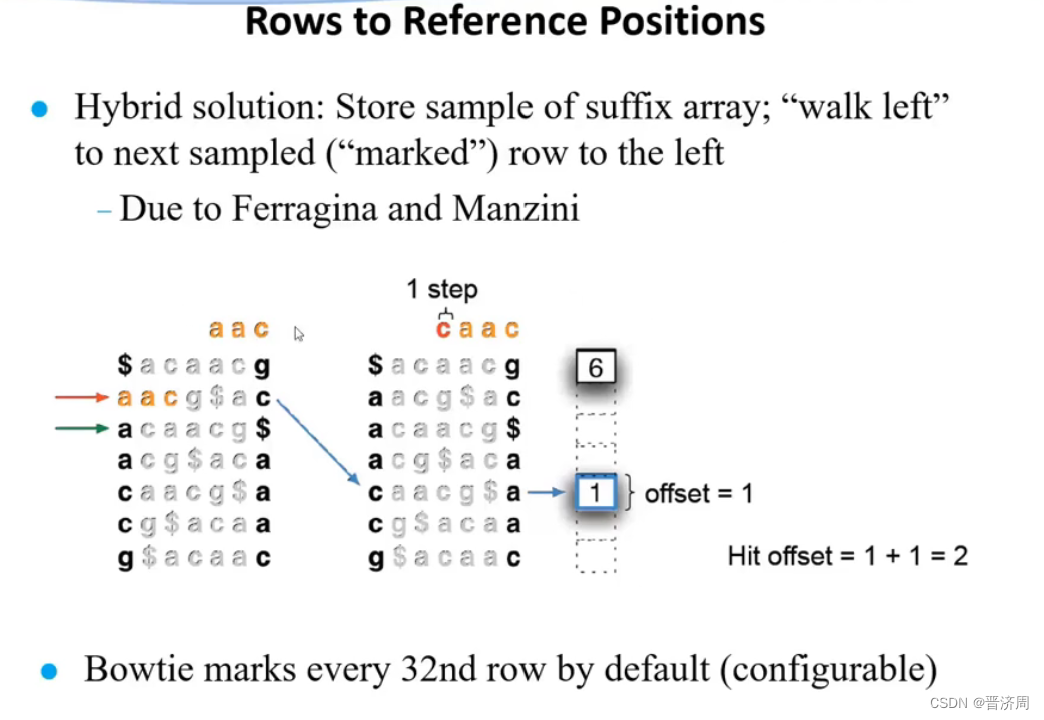
2. 基于前缀树/后缀树的BWT短序列快速比对算法



是可以还原回去的




6.6 Genome assembly
6.6.1 K mer counting
K-mer频次计算方法和意义
基因组组装的第一步:计算长度为k的基因组出现的次数或频率

挑战
- 计算量巨大
- 测序可能导致出现错误
- 将所有存储在内存困难
- 存储在硬盘中困难
- 开发并行技术去提高速度
 一次两次高,是测错导致的,去掉不影响结果
一次两次高,是测错导致的,去掉不影响结果
过程
- 对四个碱基进行编码 A(00),C(01),G(10),T(11)。

- 比对时,取编码小的
算法
- 基于哈希表的算法

常用算法

分而治之

不存在相同的 k-mers 在minmizer当中
升级

6.6.2 Genome Assembly
二代测序数据组装算法
从头组装

算法

贪心算法

出错

耗时

基于欧拉图的算法

使用K-mers 可以处理测序错误:出现错误就会出现气泡,产生分支

测错了的频次是很低的,通过频次可以区分杂合子和测试错误
过程
- 测序
- 构建布鲁因图
- 简化
- 错误移除
K的选择


第柒章 DNA-seq, RNA-seq and ChIP-seq
7.1 DNA-seq Introduction and Application
1. 基因芯片简介DNA-seq的基本原理与数据分析流程

方法:

2. 遗传变异与表型的概念
遗传变异

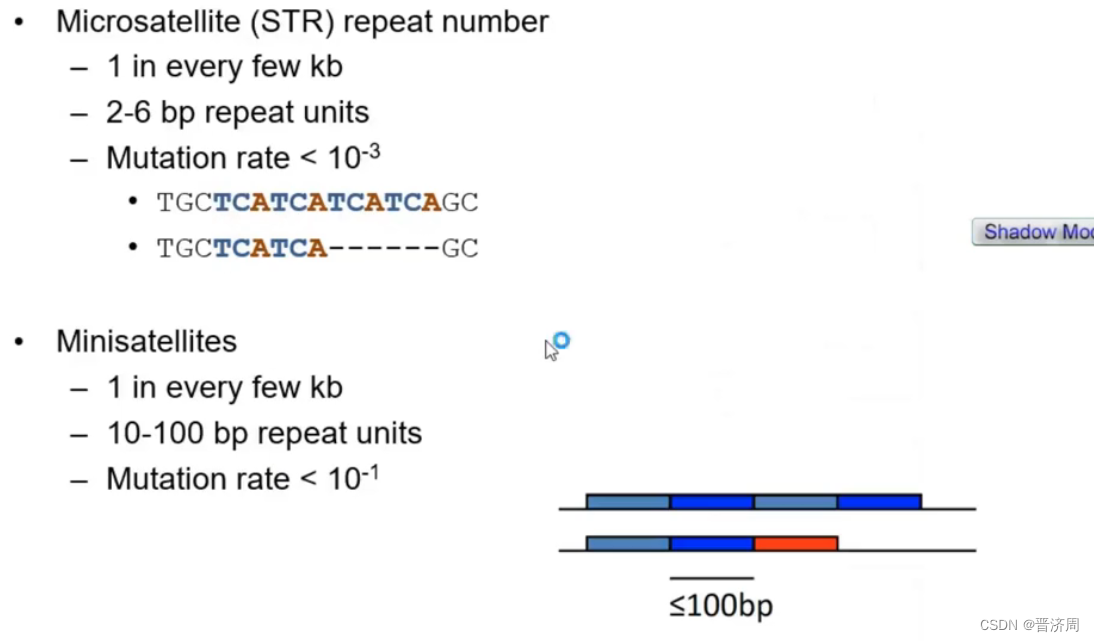
SNP


结构变异

研究原因

大部分变异不会引起疾病
并不是所有遗传变异都会引起疾病
7.2 DNA-seq Refined alignment
基因芯片简介DNA-seq精确比对的前提条件和方法

拿样本序列和参考基因组进行比对,比对错的地方进行局部重新比对即可
重比对的步骤

7.3 Variant identification
序列变异的识别算法
只使用于二倍体生物基因组
变异
纯和变异(变的一样) 杂合变异(部分一样,部分不一样)
措施



结构变异

通过显示器去测量重复度进行比对
7.4 Overview of RNA-seq experiments
1. 基因表达、RNA剪切的概念
功能

可以测基因表达的范围(等级) ; 可变(选择性)剪切; 等位特异的表达
2. RNA-seq的基本原理

- 先逆转录成cDNA
- 打成片段
- 接上接头
- 测顺序
- 与参考基因组比对片段
得出的结果

1: 基因表达水平-----> 计算差异表达; 2:选择性剪切; 3:转录层面的变异; 4:非编码RNA
3. RNA-seq与基因微阵列的区别
好处
- 不依赖于已知的基因结构 可以测(蛋白编码基因,非编码RNA,功能元件,基因融合)
差别

基因表达水平的范围
7.5 RNA-seq data analysis
1. RNA-seq数据分析流程
挑战
- 序列比对
- 转录组的重构
- 表达水平的变量
序列比对


利用的工具

基因表达水平
影响条件
- 基因长度
- 测量深度
- 基因表达的程度


2. RNA-seq代表性比对算法的基本原理

表达:
1: 取并集表达
2:取交集表达
差异表达分析

选择性剪切的问题
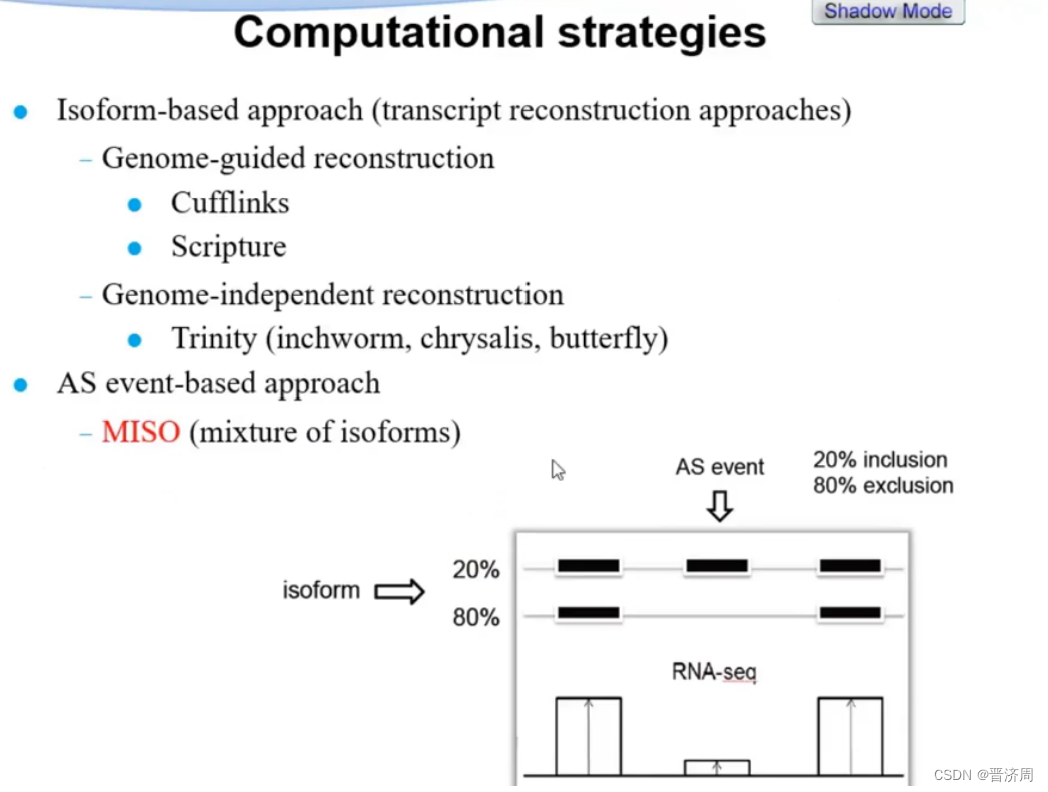
判断基因融合

基因融合问题判断

7.6 ChIP-seq
1. 蛋白质结合、染色质状态等概念


2. ChIP-seq的基本原理
例子:
生物学应用: 转录因子结合位点


组蛋白修饰

步骤
1:峰值检测
峰值的类型
转录因子 RNA聚合酶
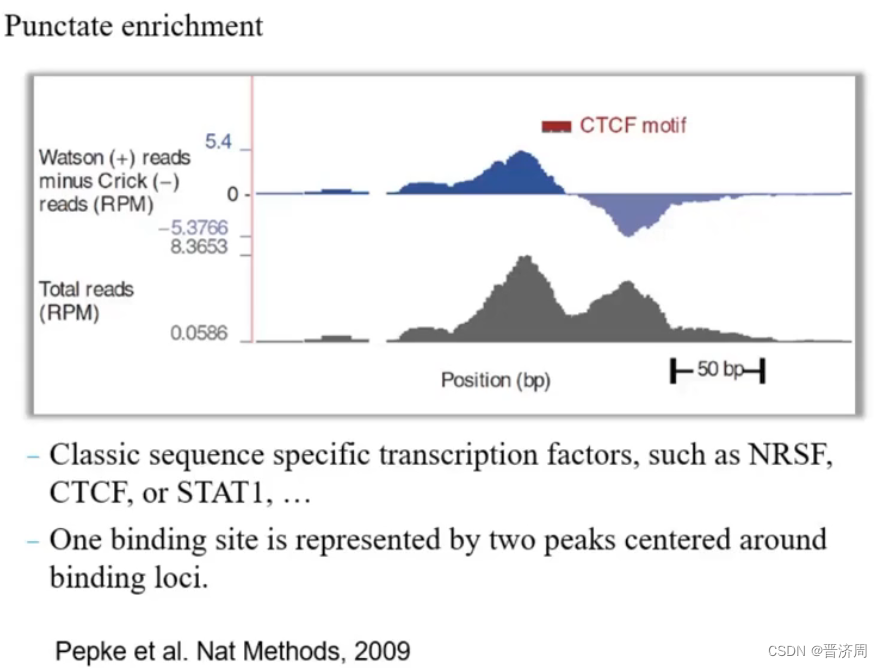

组蛋白修饰











 本文详细介绍了下一代测序技术,包括Illumina和SOLiD平台的工作原理、生物应用,以及DNA-seq、RNA-seq和ChIP-seq的实验原理、数据处理流程和分析方法。重点关注了测序过程、数据质量、比对算法和基因组组装技术。
本文详细介绍了下一代测序技术,包括Illumina和SOLiD平台的工作原理、生物应用,以及DNA-seq、RNA-seq和ChIP-seq的实验原理、数据处理流程和分析方法。重点关注了测序过程、数据质量、比对算法和基因组组装技术。



























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








