本篇文章讲解遗传算法,主要从三个方面入手:
- 遗传算法的思想:物竞天择
- 遗传算法的实例:函数优化,寻找句子,最短路径(TSP)问题
- 遗传算法的优化:精英主义思想
(第一部分刚开始有点迷糊不要紧,建议把过一下第一部分,看完第二部分再回过来品味第一部分)
一、遗传算法的思想:
1,遗传算法:
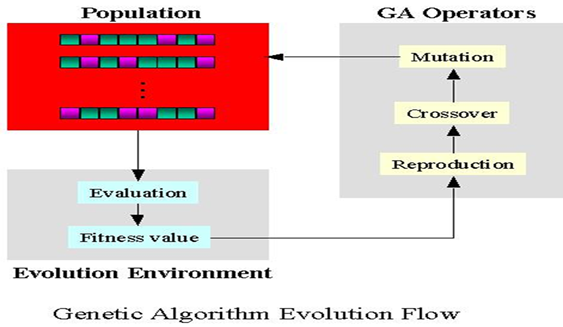
(1),遗传算法认识:遗传算法是模仿自然界生物进化的一种随机全局搜索和优化方法,它是一种自适应的优化算法。模拟物竞天择的生物进化过程,通过维护一个潜在解的群体执行了多方向的搜索,并支持这些方向上的信息构成和交换。是以面为单位的搜索,比以点为单位的搜索,更能发现全局最优解。
(2)遗传算法的过程: 遗传算法其实过程很简单,就是一直迭代到收敛。
基本遗传算法伪代码
/*
* Pc:交叉发生的概率
* Pm:变异发生的概率
* M:种群规模
* G:终止进化的代数
* Tf:进化产生的任何一个个体的适应度函数超过Tf,则可以终止进化过程
*/
初始化Pm,Pc,M,G,Tf等参数。随机产生第一代种群Pop
do
{
计算种群Pop中每一个体的适应度F(i)。
初始化空种群newPop
do
{
根据适应度以比例选择算法从种群Pop中选出2个个体
if ( random ( 0 , 1 ) < Pc )
{
对2个个体按交叉概率Pc执行交叉操作
}
if ( random ( 0 , 1 ) < Pm )
{
对2个个体按变异概率Pm执行变异操作
}
将2个新个体加入种群newPop中
} until ( M个子代被创建 )
用newPop取代Pop
}until ( 任何染色体得分超过Tf, 或繁殖代数超过G )2,遗传算法细节: 遗传算法流程性问题很简单,只有部分有差别(选择、交叉、变异);算法重点应该关注问题解空间的编码、解码,以及适应度函数设计。
(1)基因编码方式:基于编码是对问题潜在解进行“数字化”编码的方案,建立表现型和基因型的映射关系。常用的编码方式有三种:
- 二进制编码:二进制编码是指采用二进制位串对问题解进行编码;对应的解码则是先把二进制转换为十进制(可以利用矩阵的乘积进行计算),然后在把这个十进制归一化到问题解空间中;本文以函数优化为例介绍。
- 符号编码: 符号编码一般是指采用ascii码对字符问题解进行编码;对应的解码则是把ascii码转化为字符;本文以配对句子为例介绍。
- 序列编码: 序列编码是指采用数字序列对问题解进行编码;解码没有固定说法,即是把这个序列翻译成对应的实际问题;本文以TSP问题为例介绍。
(2)适应度函数: 适应度函数是你对问题的认识,因为我们是根据适应度进行选择的。设计的原则是:对于好的个体,适应度要大;反之,适应度小。
- 函数优化:可以用函数值作为适应度;如果设计轮盘选择(概率选择),则要把适应度修正,转换为正数。
- 配对句子:用匹配的字符数。
- TSP:用路径总长的倒数作为适应度。
(3)选择、交叉、变异 遗传算子: 遗传算子其实对于大部分的情况都是固定写法,只是有细微的差别,这里简单的介绍一下相同的固定思路,具体差异看TSP问题实例。(这里所说的固定是指当你选定的选择、交叉、变异的策略方法确定后,写法大部分一样,并非指方法策略是唯一的。)
- 选择:常用轮盘法:就是根据每个个体的适应度大小给每个个体赋予一个选择概率,然后根据这个概率随机的选择。
- 交叉:选择两个父亲,交换一部分基因编码,产生新的个体
- 变异:改变某个基因的一部分。
三者的作用:
选择的作用:选择好的个体群;优胜劣汰,适者生存;
交叉的作用:产生新的个体;保证种群的稳定性,朝着最优解的方向进化;
变异的作用:产生新的个体;保证种群的多样性,避免交叉可能产生的局部收敛。
二、遗传算法实例:(本文所有代码都是用python实现)
1,函数优化问题: 求函数y(x)=sin(10*x)*x + np.cos(2*x)*x的最值,x取值范围:[0,5]。
实例的完整代码,包括可视化展示:函数优化
(1)编码: 我们采用二进制编码,可以根据你的精确度选定位数,这里采用10位。
解码: 先将二进制转换为十进制,再将十进制归一化的问题空间内。
DNA_SIZE = 10 # 编码长度
POP_SIZE = 100 # 种群大小
CROSS_RATE = 0.8 # 交叉概率
MUTATION_RATE = 0.003 # 遗传概率
N_GENERATIONS = 200 # 迭代次数
X_BOUND = [0, 5] # x的取值范围def translateDNA(pop): # 转换DNA到十进制,并归一化到x得区间内
return pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2 ** DNA_SIZE - 1) * X_BOUND[1]
'''
这里要注意两个地方:
1,权重weight=np.arange(DNA_SIZE)[::-1]),这里的数组切片处理容易让人迷惑,[开始:结束:步长],这的负号代表的是从后向前
2,这里的十进制计算使用pop与weight的内积计算得到,巧妙的运用的矩阵的内积
'''(2)适应度: 这里用函数值代表适应度,但是用轮盘选择,所以要进行非负转换。
def get_fitness(pred): # 计算适应度值
return pred + 1e-3 - np.min(pred) # 转化值,使得概率为正
# return pred(3)选择: 轮盘法
def select(pop, fitness):
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True, p=fitness / fitness.sum())
return pop[idx]
'''
# 这里的选择是调用choice方法进行,p代表的选择的概率大小(轮盘选择法),size是选择的个数,我们选择保持种群大小不变
'''(4)交叉: 采用多点交叉:
def crossover(parent, pop): # 交叉过程
if np.random.rand() < CROSS_RATE: # 产生0-1的随机值
i_ = np.random.randint(0,POP_SIZE,size=1) # 选择另外一个交叉个体
cross_points = np.random.randint(0,2,size=DNA_SIZE).astype(np.bool) # 选择交叉点,这里假设每个基因都可以交叉
parent[cross_points] = pop[i_,cross_points] # 完成交叉
return parent(5)变异:每一个位都有几会变异
def mutate(child):
for point in range(DNA_SIZE):
if np.random.rand() < MUTATION_RATE:
child[point] = 1 if child[point] == 0 else 0 # python中的三元运算符,等价于:child[popint] = np.where(child[point]==0],1,0)
return child2,配对句子: 就是在所有字符里面构成出你自己设定的句子:TARGET_PHRASE = ‘I love LuoSiHan!。
实例的完整代码,包括可视化展示:配对句子
(1)编码:用ascii码来表示字符串。解码:把ascii码翻译成字符串。ascii的取值范围【32,126】。
TARGET_PHRASE = 'I love LuoSiHan!' # 目标句子,目标DNA
POP_SIZE = 300 # 种群大小
CROSS_RATE = 0.4 # 交叉概率
MUTATION_RATE = 0.01 # 变异概率
N_GENERATIONS = 1000 # 迭代次数
DNA_SIZE = len(TARGET_PHRASE) # DNA长度
TARGET_ASCII = np.fromstring(TARGET_PHRASE,dtype=np.uint8) # 转化为数字
ASCII_BOUND = [32,126] # 编码的取值范围def translateDNA(self,DNA): # 解码:转化为字符串就行了
return DNA.tostring().decode('ascii')(2)适应度: 计算与目标配对字符的个数
def get_fitness(self): # 适应度大小:匹配了多少位
match_count = (self.pop == TARGET_ASCII).sum(axis=1)
return match_count(3)选择、交叉、变异: 选择、交叉和基本的一样;变异也只是随机赋值,[32,126]中的一个值,而不是0,1改变。具体看源码。
3,TSP最短路径问题: 问题就是要遍历每一个城市,使得路径长度最小。
实例的完整代码,包括可视化展示:最短路径
(1)编码: 采用路径的城市序列编码。
解码:将这个城市序列翻译成,每个城市横、纵坐标,用来计算长度。
def translateDNA(self,DNA,city_position): # 解码:返回DNA序列代表的城市顺序的x,y坐标
line_x = np.empty_like(DNA,dtype=np.float64)
line_y = np.empty_like(DNA,dtype=np.float64)
for i,d in enumerate(DNA):
city_coord = city_position[d]
line_x[i,:] = city_coord[:,0]
line_y[i,:] = city_coord[:,1]
return line_x,line_y(2)适应度: 采用路径长度的倒数。
def get_fitness(self,lx,ly): # 计算适应度:距离越大,适应度越小
total_distance = np.empty((lx.shape[0]),dtype=np.float64)
for i,(xs,ys) in enumerate(zip(lx,ly)):
total_distance[i] = np.sum(np.sqrt(np.square(np.diff(xs)) + np.square(np.diff(ys)))) # 计算距离
fitness = np.exp(self.DNA_size * 2 / total_distance)
return fitness, total_distance(3)选择:选择和基本的一样。
(4)交叉:不同的是要保证每一个城市都要访问,不能单纯交叉。
def crossover(self,parent,pop): # 交叉:不能简单的把两个互换,必须保持所有点都要访问
if np.random.rand() < self.cross_rate:
i_ = np.random.randint(0,self.pop_size,size=1)
cross_points = np.random.randint(0,2,size=self.DNA_size).astype(np.bool)
keep_city = parent[~cross_points]
swap_city = pop[i_,np.isin(pop[i_].ravel(),keep_city,invert=True)]
parent[:] = np.concatenate((keep_city, swap_city))
return parent(5)变异:不同的是要保证每一个城市都要访问,不能单纯变异。这里采用交换基因中的两个位置。
def mutate(self,child): # 变异:不能简单改变某一个值,必须保持所有点都要访问
for point in range(self.DNA_size):
if np.random.rand() < self.mutate_rate:
swap_point = np.random.randint(0, self.DNA_size)
swapA, swapB = child[point], child[swap_point]
child[point], child[swap_point] = swapB, swapA
return child三、遗传算法的优化:精英主义思想。也就是每次把好的给保留下来。它要优化的进化思路,并没有对交叉、变异做改变。进化过程:每次进化没有统一的选择了,它每次选择两个个体,对这两个进行比较,把好的保留下来,并用好的去改变不好的(交叉)。
实例的完整代码,包括可视化展示:精英主义
(1)进化过程:
def evolve(self, n): # 进化过程中,每次悬着两个,对于适应度低的那个,利用使用度高的进行交叉和变异,保留适应度高的
for _ in range(n):
sub_pop_idx = np.random.choice(np.arange(0, self.pop_size), size=2, replace=False)
sub_pop = self.pop[sub_pop_idx]
product = F(self.translateDNA(sub_pop))
fitness = self.get_fitness(product)
loser_winner_idx = np.argsort(fitness)
loser_winner = sub_pop[loser_winner_idx] # 排序:第一个是适应度小的,第二个是适应度大的
loser_winner = self.crossover(loser_winner) # 交叉
loser_winner = self.mutate(loser_winner) # 变异
self.pop[sub_pop_idx] = loser_winner
DNA_prod = self.translateDNA(self.pop)
pred = F(DNA_prod)
return DNA_prod, pred(2)交叉:
def crossover(self, loser_winner): # 交叉:把适应度高的个体,交叉点对应的的值给适应度低的个体
cross_idx = np.empty((self.DNA_size,)).astype(np.bool)
for i in range(self.DNA_size):
cross_idx[i] = True if np.random.rand() < self.cross_rate else False
loser_winner[0, cross_idx] = loser_winner[1, cross_idx]
return loser_winner





















 16万+
16万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








