想做生物信息分析,在linux系统中分析,很多时候我们要分析一个内容会用到一个分析软件,那么开展这个分析的第一步就是要把这个软件安装到我们的linux系统中才可以分析,对于linux初学者而言,安装软件也不是那么容易的,所以初学者在第一步就放弃了,因为不懂linux的工作原理一旦安装报错就很难解决。那么在linux系统中有么有像360软件管家一样的工具,只要输入软件名然后自动安装呢?答案是有的那就是conda,有很多的生信软件都可以通过conda安装,省去了安装、调试、设置环境的烦恼。经常有安装到崩溃的软件,conda一行命令就搞定了。
conda简介
Conda 是一个开源的软件包管理系统和环境管理系统,用于安装多个版本的软件包及其依赖关系,并在它们之间轻松切换。Conda 是为 Python 程序创建的,适用于 Linux,OS X 和Windows,也可以打包和分发其他软件。通俗的讲:conda相当于我们windows里面的360软件管家,想要那个软件点击安装就好了;安装conda
conda分为anaconda和miniconda。anaconda里面会包含很多常用的和软件的版本(但是这里的常用不代表你常用),miniconda则是精简版,需要啥装啥,所以推荐使用miniconda。下载网址
conda官网:https://docs.conda.io/en/latest/miniconda.htmlwget -c https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh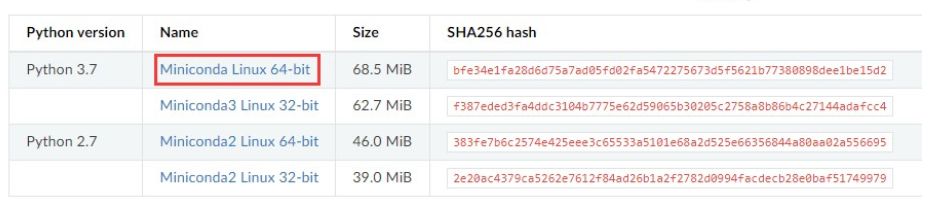
安装命令:
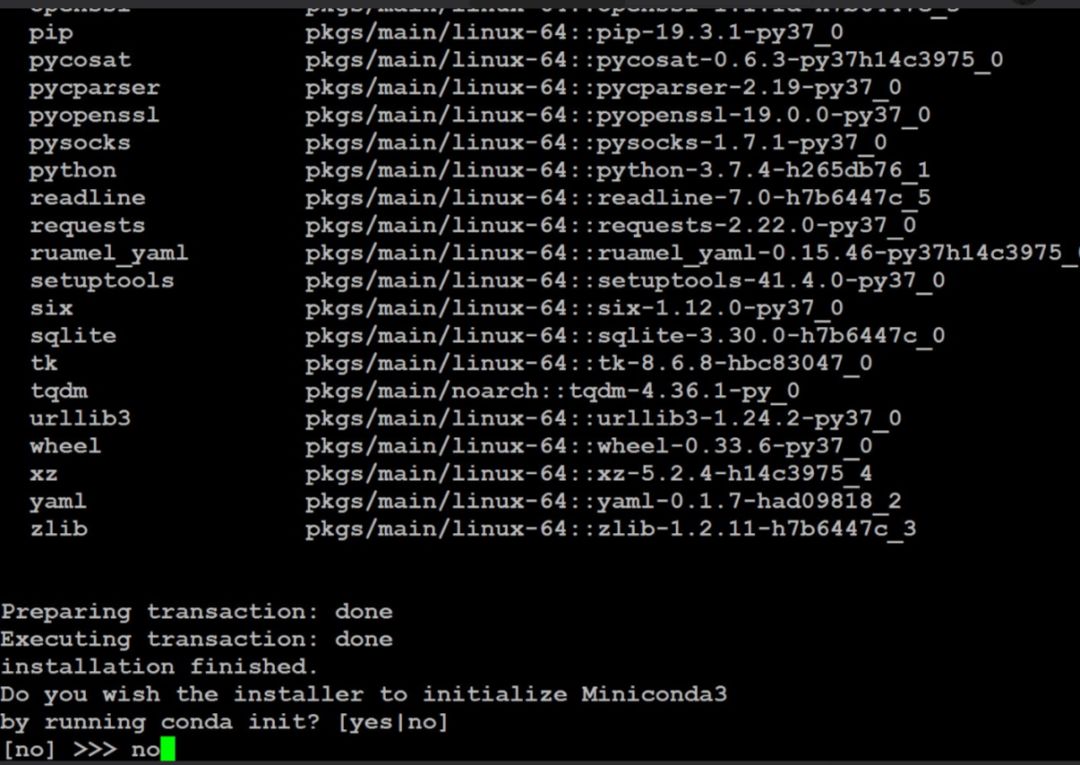
sh Miniconda3-latest-Linux-x86_64.sh #运行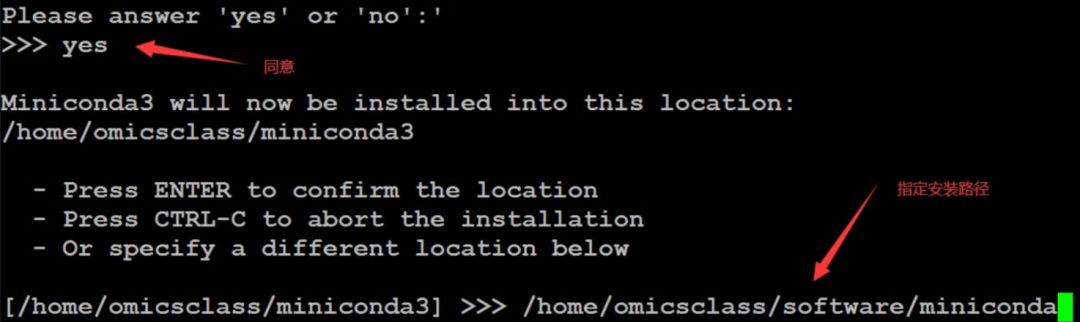 安装的过程会输出一些日志,显示在屏幕上,也就是安装了一些常用的包:
安装的过程会输出一些日志,显示在屏幕上,也就是安装了一些常用的包:
注意:最后询问你是否设置miniconda为默认环境,一般初学者建议输入no 然后enter,如上图最后所示。到这里 miniconda 已经安装完成了。
启动conda
在上一步选择no之后,输入conda是会报找不到此命令的。那么可以找到你刚才安装的miniconda路径(我这里是/home/omicsclass/software/miniconda),cd到miniconda3的bin目录下面,能看到有一个activate 和deactivate ,分别是启动和退出环境。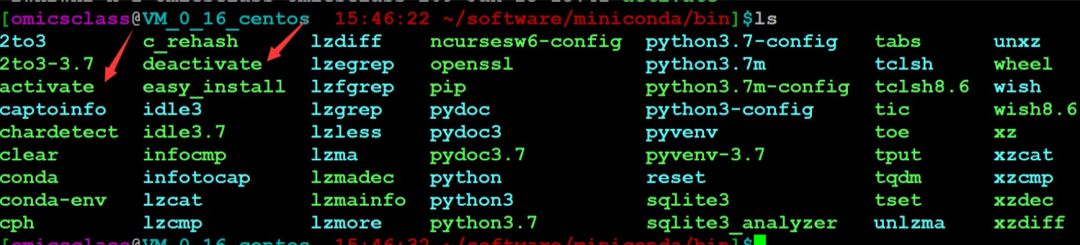 接下来在安装路径的bin目录下:启动conda进入基础(base)conda环境:
接下来在安装路径的bin目录下:启动conda进入基础(base)conda环境:
conda activate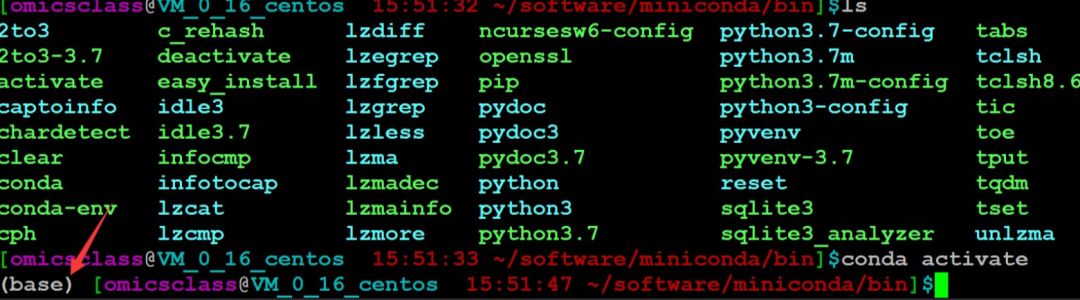 当命令行前面出现(base)的时候说明现在已经在conda的环境中了。这个时候可以使用:conda list 查看环境中安装的软件以及版本:
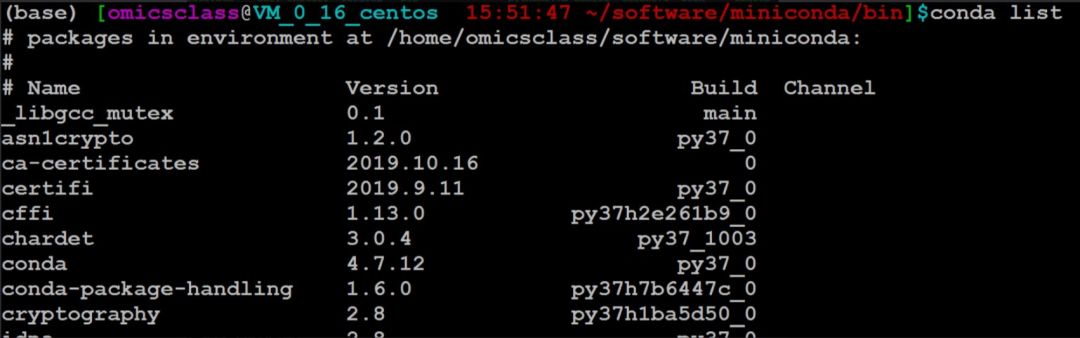
当命令行前面出现(base)的时候说明现在已经在conda的环境中了。这个时候可以使用:conda list 查看环境中安装的软件以及版本:
添加镜像地址:
miniconda相当于我们windows里面的360软件管家,在安装软件的时候,会自动的从一些网络库中下载软件源码并安装,默认的库服务器大多在国外,因此,为加快软件下载安装的速度,我们可以添加国内的镜像给conda,这样可以加快我们的安装速度,可在命令行运行以下命令添加,另外还有一个生物领域非常重要的库(channel)地址,bioconda,里面有我们常用的生物信息数据分析的软件,例如 bwa,samtools等等,大家可以运行以下命令添加:#官方默认的channel
conda config --add channels bioconda
conda config --add channels conda-forge
conda config --add channels genomedk
#国内清华的channel
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/ conda config --set show_channel_urls yes #设置显示安装的频道
conda config --get channels #查看已经添加的channels
vim ~/.condarc #已添加的channel在哪里,也可以修改这个文件,手动添加和删除利用conda安装生物信息软件
以下命令大家进入conda环境之后就可以运行尝试安装软件了:#安装软件命令
conda install isoseq3#如果不知道软件名字,以及版本可以搜索
conda search isoseq#安装指定版本的软件#conda install 软件名=版本号
conda install isoseq3=3.2.1#查看已经安装的软件
conda list#卸载已经安装的软件
conda remove isoseq3conda deactivate
注意:退出conda环境之后,我们的软件命令不能直接运行了,其实我们安装的软件都在miniconda的bin目录下:/home/omicsclass/software/miniconda/bin 我们可以绝对路径运行这些命令就可以。大家可以去试试安装软件吧,如果对linux感兴趣可以点击阅读原文学习linux。
延伸阅读: 1. 拨乱反正-请下载新的2019年中科院期刊分区及影响因子汇总表 2. 如何检测外源基因的插入位置?! 3. 如何下载基因组及查找基因 4. NCBI-SRA数据提交流程 5. qRT-PCR相对定量计算详解 6. 收藏贴-绘图工具教程合集-科研必需技能! 7. 文献检索下载+期刊分析+投稿指南一条龙服务-此等神器,值得收藏! 8.100+篇2019年基因家族分析SCI(1-3月发表)免费领(文末领取) 9. 充电课-(适合小白自学生信)《Linux生信分析环境搭建Bio-linux》 10. 充电课-限时免费领取《癌症TCGA-文章套路解析》课程 11. 充电课-免费领取《illumina测序原理及Fastq文件解读》视频课程 12. 充电课-转录组扫盲篇 13. 充电课-限时免费领取《基因家族分析详解课程》 14. 充电课-升华你的转录组文章-转录因子分析 15. 充电课-《如何在NCBI数据库中检索、下载感兴趣的测序数据》视频课程限时领取





















 322
322











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








