作者按
我希望我的投稿能给所有从事脑功能研究的同学、同行、老师带来帮助,能让他们在山重水复疑无路时有所借鉴。本文章是原创文章,如需转载,请联系原作者。
脑功能MRI数据处理手册
作者:范嘉晨 指导老师:刘加成单位:东南大学(江苏省分子影像与功能影像重点实验室)
目录:
本手册介绍
相关软件
图像格式
Bold预处理
Resting-Bold建模、统计分析、结果可视化
Task-Bold建模、统计分析、结果可视化
DTI预处理
TBSS及DTI结果可视化、FDT
结构像预处理
VBM及结果可视化
附录一:task-Bold处理相关参数
附录二:VBM处理相关参数
附录三:resting-Bold处理相关参数
附录四:DTI处理相关参数
附录五:行为学范式与磁共振扫描对应关系
第七章:DTI预处理
本手册处理静息态磁共振数据及弥散张量成像数据使用FSL6.0.0。
扩散张量成像(DTI)是一种新型的磁共振成像技术。利用DTI技术,可以进行基于活体的大脑解剖连接和神经环路的研究,而且近年来被应用于大脑解剖网络的构建和分析,可以从全局角度来进一步认识大脑的组织结构和功能。该技术自问世以来,在认知神经科学和临床神经科学方面得到了广泛的应用并取得了巨大的成就。该技术能够提供脑组织内水分子的扩散信息,从而能间接反映脑白质纤维束的物理和功能特性,因此它广泛用于神经白质、骨骼肌、心肌等各向异性介质的病理特异性研究。这对于相关疾病的临床诊断具有潜在的价值,有可能对那些危险性大的疾病如脑梗阻和心肌梗阻作到早期发现和及时治疗。近年来,国外已经用DTI 技术对一些疾病如多发性硬化症(multiplesclerosis,MS)、慢性中风(stoke)、阿尔茨海默病(Alzheimer disease, AD)精神分裂症(schizophrenia)等疾病进行了病理的特异性研究。由DTI 数据导出的神经纤维束成像(fiber tractography)可以对人的中枢神经纤维精细成像。这突破了传统的尸体解剖学脑白质纤维束追踪方法,使得脑白质纤维束的活体研究成为可能,已成为脑科学研究领域的热点课题之一。根据所显示的活体神经纤维束的构造,人们已经发现了人脑和心脏的一些其它方法未能揭示的组织结构,这对认识大脑结构以及探索神经疾病、精神疾病的生理机制是极其有利的补充。 DTI 主要是利用大脑内各种组织的水分子在外加磁场下表现出不同的扩散行为而进行成像。在均匀介质中,水分子的运动在各个方向上的扩散速度是相等的,其扩散表现为各向同性;在纤维性介质中,水分子的运动在沿着纤维束的方向上的扩散速度最快,而在垂直于纤维束的方向上扩散速度最慢,其扩散表现为各向异性。大脑组织中白质、灰质和脑脊液中纤维性成分的含量存在显著的差异,所以水分子在不同类型脑组织中的扩散呈现出不同的扩散行为,从而为大脑的扩散张量成像提供了物理基础。 DTI 图像不同于传统的 MR结构图像, 它不是用单一的灰度值来描述大脑内各个体素的特性,而是用一个二阶张量(3×3的对称正定矩阵)来刻画大脑内每个体素的水分子扩散特性。该二阶张量的物理意义可描述为三维空间的一个椭球;椭球的三个轴的方向对应于该矩阵的三个特征向量,轴长等于三个特征向量所对应的特征值。通过对多个方向应用扩散敏感梯度来采集信号,可以获得椭球表面的一系列采样点,从而利用数学的方法计算出张量矩阵的各个独立元素。以上是DTI的一些概念性的东西,现在开始说说具体怎么处理DTI数据 首先我们使用的Oxford University 的Analysis Group ,FMRIB开发的fsl这款软件。这款软件可以处理FMRI,MRI,DTI的数据。

平时我们遇到问题或者想查看其手册,可以通过下图的方式。有些细节问题可以去它的email forum询问。
1.首先进行图像格式转换,因为我们从飞利浦机子上拷贝下来的图像格式是dicom格式,需要将其转换成Nifti格式,最好是4D Nifti。我们可以在终端输入dcm2nii,查看其用法,直接在终端利用dcm2nii命令转换,当然也可以通过dcm2nii图形界面添加。将后缀名为dcm的原始数据转换为nii.gz格式的数据,nii.gz格式的数据不损失原始数据信息,并且能节省大量空间。FSL能够处理的是nii.gz格式的数据。
需处理的文件包括:T13D数据和DTI数据。3D结构像生成原文件、o开头、co开头的文件。
其中o开头的文件主要是进行了reorient的,而co是经过切割了neck的。
一般用于空间normalize都选用co开头的文件;
对于DTI数据,Philips的数据选用已x开头的.nii.gz文件。

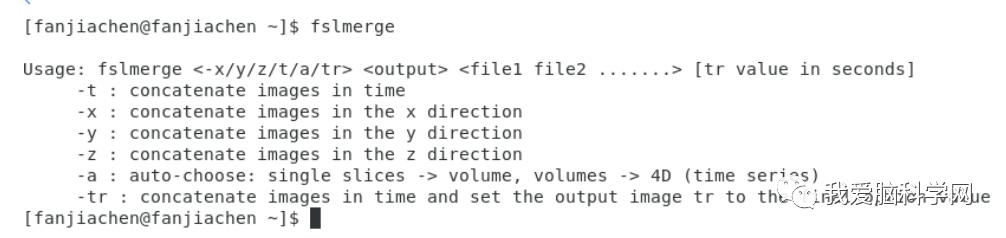
一般正确转换后,我们的图像会包含33个volumes,第1个volume是b0,接下来32个volumes是b1000,如果b0和b1000是分离的,可以使用fslmerge进行图像融合。除了生成x开头的nii.gz文件,还会有.bvec文件(显示b的方向)和.bval文件(显示b值)。(bval文件中存放的b在每次扫描的值,bvec存放的是每次扫描的方向值。)

2.提取b0图像
需处理的文件为:.nii.gz格式的DTI数据
命令:fslroi 开头的DTI的nii.gz格式的文件> b0 0 -1 0 -1 0 -1 0 1 ##fslroi
输出文件:b0.nii.gz
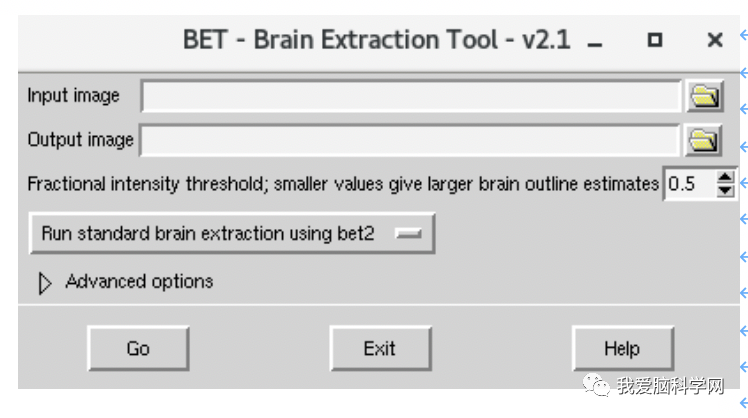
3.剥脑(也可以在终端输入Bet,打开图形界面,点点点)
命令:bet2 b0.nii.gz nodif_brain -m -f 0.3
bet2 开头的3D T1数据> <输出名字> -m -f 0.3 ##bet [options]## -m 生成二进制大脑mask;-f 部分强度阈值(0-1),预设是0.5,值越小,估计大脑的框架更大。
需处理的文件包括:.nii.gz格式的3DT1数据和DTI数据
输出文件:nodif_brain.nii.gz和nodif_brain_mask.nii.gz
需要检查剥脑效果,后面如果想做TBSS的话,
个别被试的剥脑结果不好的话会影响整个骨架的mask。
4.涡流校正
命令:eddy_correct 开头的DTI的nii.gz格式的文件> data 0 ##eddy_correct<4d input> <4d output> 参考层面>
输入文件:.nii.gz格式的DTI数据
输出文件:data.nii.gz
此处生成的文件data.nii.gz就是包含后面一系列处理需要使用的文件了。
5.计算张量,FA,MD值等
命令:dtifit --data=data.nii.gz --out=data--mask=nodif_brain_mask.nii.gz--bvecs=<以.bvec为后缀的文件> --bvals=<以.bval后缀的文件>
输入文件:eddy_currect生成的文件data.nii.gz,nodif_brain_mask.nii.gz 以.bvec和.bval后缀的文件
输出文件:data_FA.nii.gz data_L1.nii.gz data_L2.nii.gz data_L3.nii.gz
data_MD.nii.gz data_MO.nii.gz data_SO.nii.gz data_V1.nii.gz
data_V2.nii.gz data_V3.nii.gz
## --data dti数据文件; --out输出文件名; --mask 剥脑的二进制mask文件;--bvecs b vectors 文件;--bvals b values 文件。
预处理会产生如下文件:data_FA.nii.gzdata_L1.nii.gz data_L2.nii.gz data_L3.nii.gz data_MD.nii.gz data_MO.nii.gzdata_SO.nii.gz data_V1.nii.gz data_V2.nii.gz data_V3.nii.gz
此时计算出来的各项数据均是个体空间下的,没有配准到标准空间里,
不能直接做组间对比。

DTI预处理跑出FA、MD、tensor等
第八章:TBSS及DTI结果可视化、FDT
DTI处理流程——tbss(Tract-Based Spatial statistics)
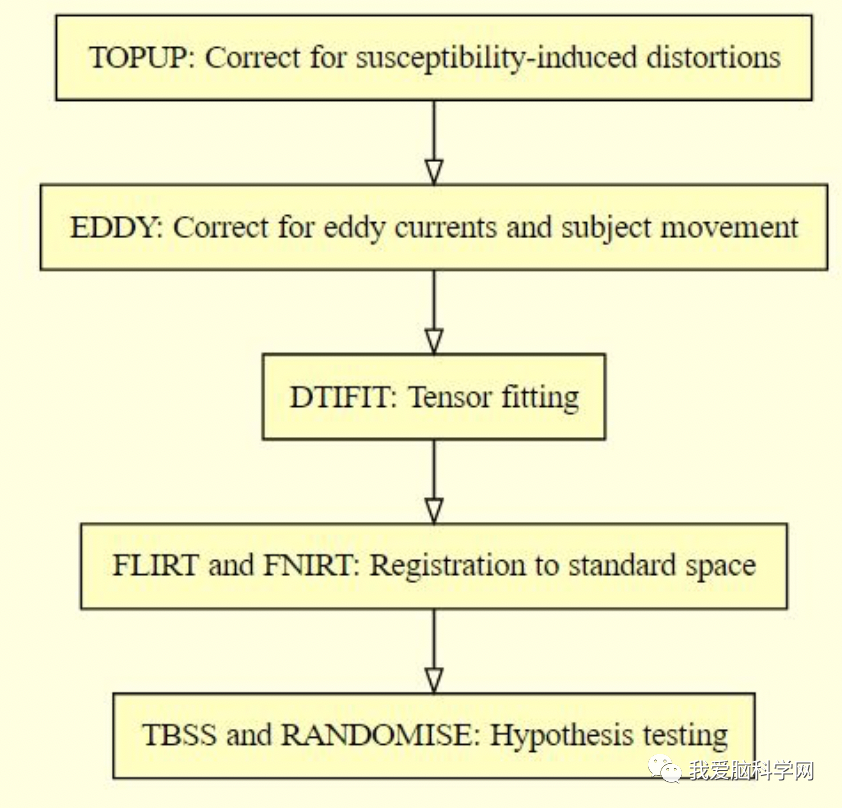
TBSS:(以下命令直接在终端输入,但是请注意目录)
1. 创建一个名为TBSS的文件,里面复制之前跑出来的所有受试的FA值(使用slicesdir *.nii.gz可以打开火狐浏览器查看受试的图片);
2. 为TBSS准备FA值(tbss_1_preproc *.nii.gz),轻微地侵蚀FA图来去除大脑边缘的伪影(erode your FA images slightly to remove brain-edge artefacts ),并且使最后一张图变为0 (zero the end slices)。这步会创建两个子文件夹(FA、origdata(存放原始FA)),使用slicesdir *.nii.gz查看,或者打开FA文件夹下面的slicesdir/index.html。
3. 配准所有受试的FA值,使用tbss_2_reg -T(推荐)来非线性配准所有受试的FA值,把FA配准到FMRIB58_FA模板上(这步是非线性配准到FMRIB58_FA_1mm模板上,注意不要在FA目录下运行此命令,否则会出错,请在TBSS目录下运行此命令)。也可以用tbss_2_reg -t 目标文件,这就可以设定非线性配准的配准对象,这一步也是在tbss目录下运行,而且目标文件必须与FA图像同级,即在同一个文件夹下。
4. 配准后处理,使用tbss_3_postreg-S(推荐使用),将所有上述第3步中配准完的图像配准到1*1*1的标准空间。然后此步所有非线性配准完的图像再被融合成一个4D图像文件(名为all_FA),这个文件被自动创建在stats文件夹下,FA图像的平均值名为mean_FA也被创建,然后FA骨架化程序创建mean_FA_skeleton。(stats文件下有all_FA.nii.gz,mean_FA.nii.gz,mean_FA_mask.nii.gz,
mean_FA_skeleton.nii.gz)
5. 把所有第四步中配准完的FA值投射到骨架上,tbss_4_prestats 0.3 ;这个命令把上一步中all_FA中的4D配准完的FA图投射到平均FA骨架上,这会创建一个包含投射后FA数据的4D图像文件all_FA_skeletonised。这个文件夹里的数据用于基于体素的统计分析。(这步产生一个all_FA_skeletonised.nii.gz文件)
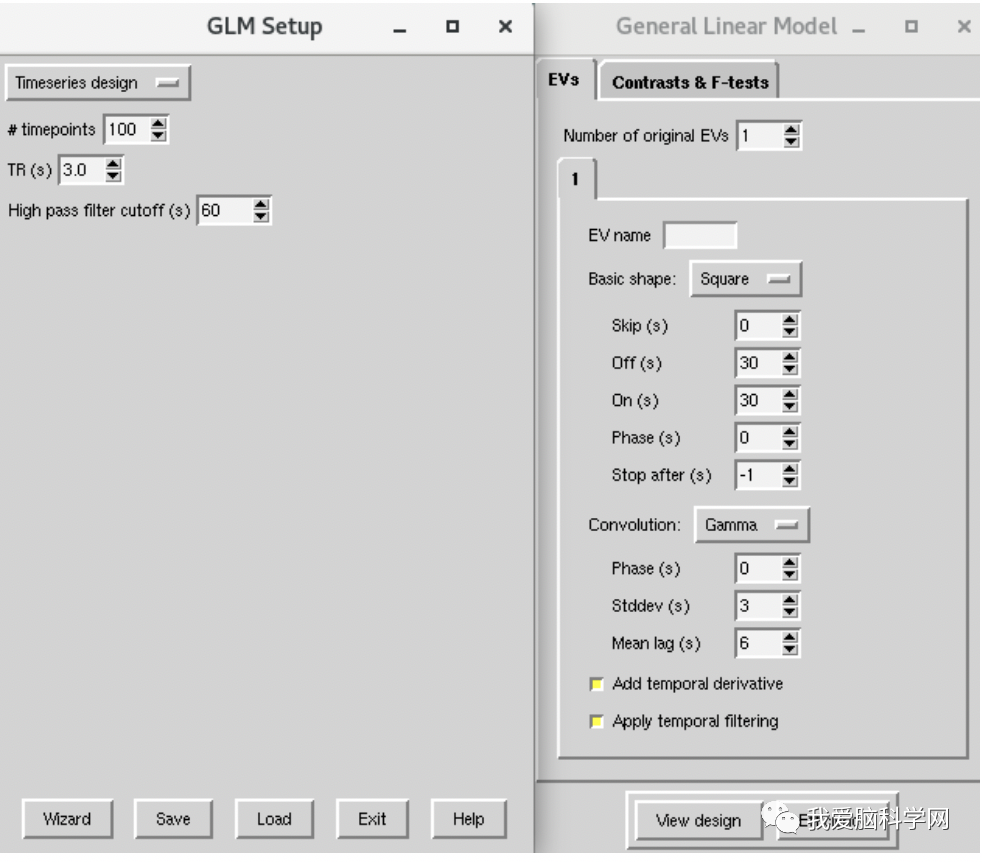
6. 在骨架化后的FA数据进行基于体素的统计分析。终端输入cd stats ,在此目录下输入Glm打开Glm的图形界面,把timeseries design改为higher-level/non timeseries design。创建自己的design,保存产生design.mat和design.con。
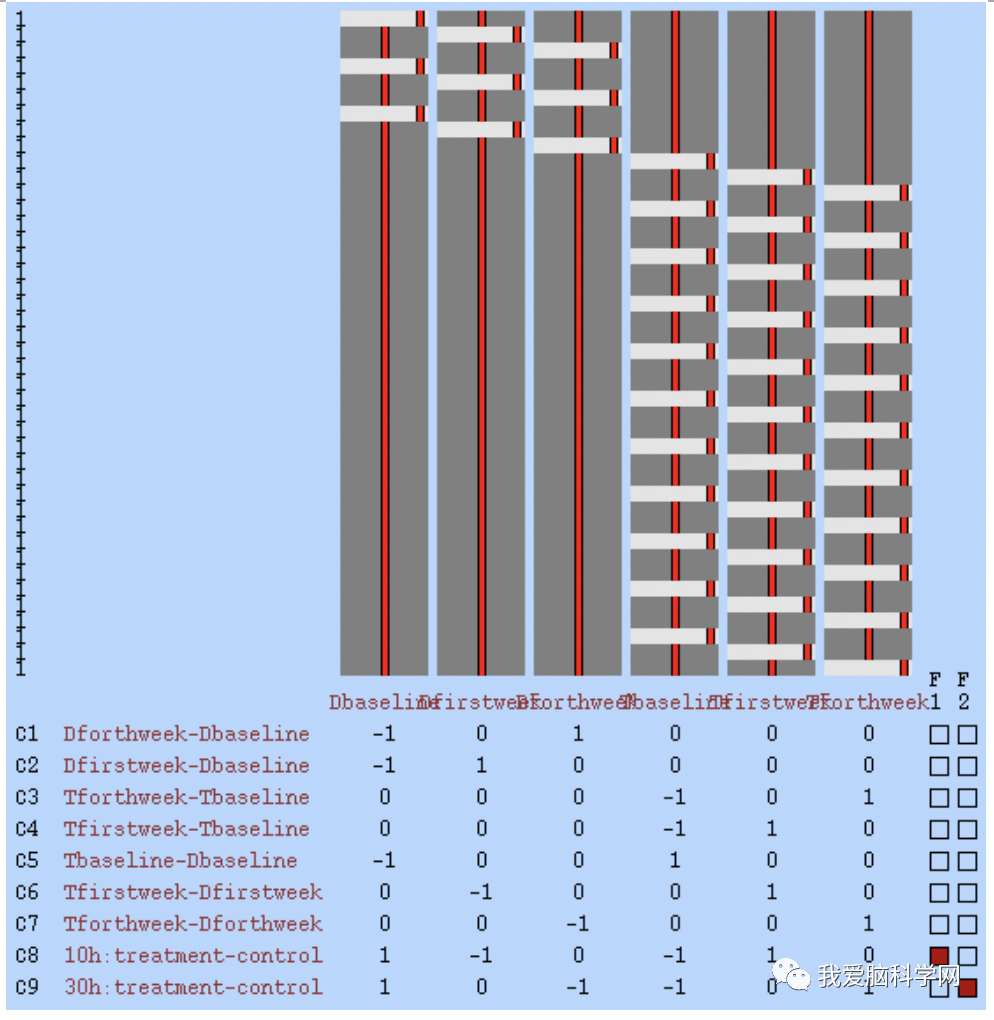
7. 注意设计矩阵里每行要一一对应原始FA的字母顺序,因为原始FA的顺序决定了4D 文件all_FA_skeletonised的顺序,可以使用cd FA ,imglob * _FA .* 命令查看确认。
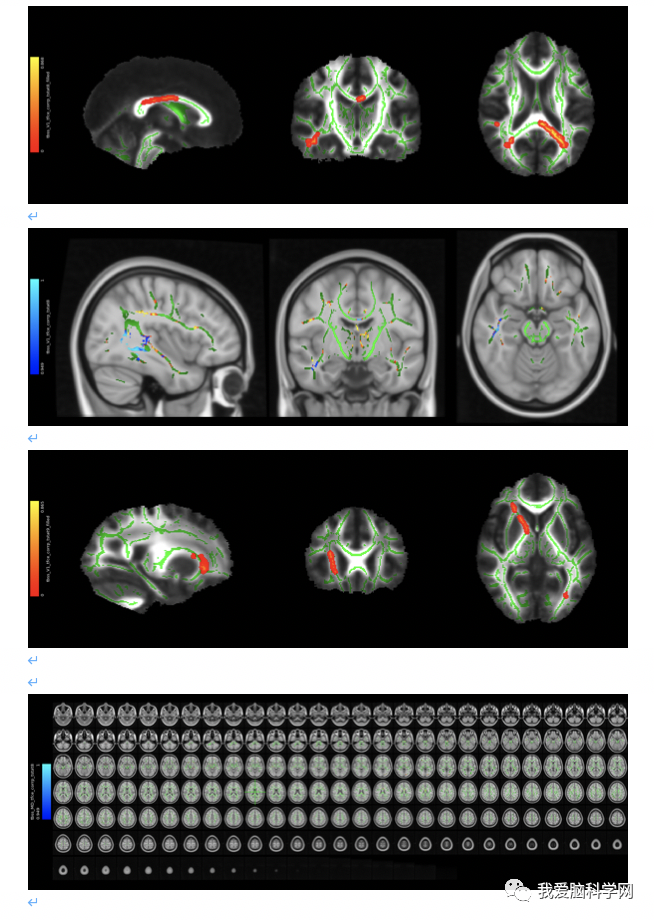
建议使用randomise。在randomise之前,准备好设计矩阵和对比文件(e.g. design.mat和design.con)。我们通过Glm图形界面来产生这两个文件(设计矩阵中的行(entries或rows)必须与原始FA图像的字母顺序匹配,因为这决定了最后4D文件all_FA_skeletonised中配准FA图像的顺序,终端输入Glm,)把Timeseries design 改为Higher-level/non-timeseries design,#input里填入要比较的受试的数目,按回车,点击wizard来选择统计方式(如two groups ,unpaired),根据要比较的对象,选择contrasts,最后以design为文件名保存设计,在终端使用less查看design.mat和design.con两个文件。使用randomise -i all_FA_skeletonised -o tbss -m mean_FA_skeleton_mask -ddesign.mat -t design.con -T2 1.5 。会有类似tbss_tstat1(存放原始unthresholded的tstat图像产生)和tbss_clustere_corrp_tstat1(多重比较校正p值后的相应cluster image(如果使用-T2,则不会以clustere命名))产生。
randomise -i -o -d -t [options],其中options一般选用—T2。
比如 randomise -i all all_FA_skeletonised -o tbss -mmean_FA_skeleton_mask -d design.mat -t design.con -c 1.5
8.如果想要展示fattened的结果,同时对平均FA图像进行掩码masking,可以使用
tbss_fill tbss_clustere_corrp_tstat1 0.95 mean_FA tbss_clustere_corrp_tstat1_filled。(或 者在fsleyes设置(display settings panel )里把插值(interpolation)选项改为——Spline interpolation,降低剪切范围的下限值,以便于平滑的区域不被修剪。)




9.关于fsleyes的截图,可以点击左上角工具条里的相机图案,截取单一层面的图;
也可以,点击view——lightbox view,浏览全脑图像,或者view——layouts——plain lightbox,然后再点相机图案,可以获得全脑图像。我们可以在图形中选择colorbar的一些属性,点击左上角工具栏内的扳手图案,打开设置,可以调整其放置位置/大小/标签位置/标签大小。比如下图设置。
DTI处理流程——FDT (FMRIB's Diffusion Toolbox)
Bedpostx output:估计每个体素的单个纤维群体
Tractography: 从单个体素生成连接性分布
Connectivity-based segmentation :使用连接性去划分体素,从种子掩模中进行概率性纤维追踪。
Crossing-Fibre Tractography:使用多纤维方向产生连接性分布。
Anatomical priors in tractography:使用排除/路点/终止掩模来进行纤维追踪
Bedpostx model spexification:选择正确的模型提高数据拟合
Using surfaces to constrain tractography:使用表面来避免虚假的连接
Tractography:
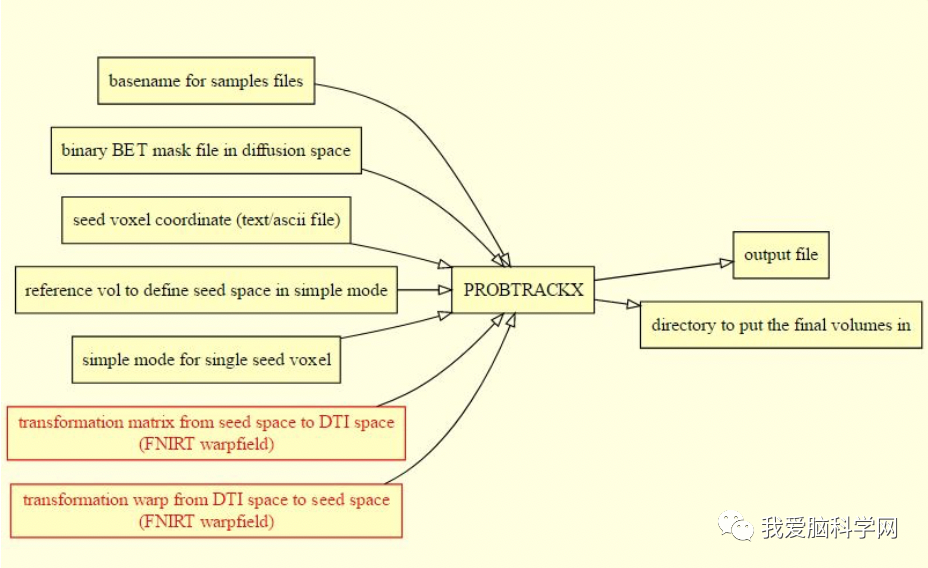
1.简单追踪——单一种子的连接性,终端输入Fdt,预设是PROBTRACKX Probabilistic tracting,选择*.bedpostX作为BEDPOSTX directory,选择体素(x,y,z)作为种子,键入输出名字,点击Go。
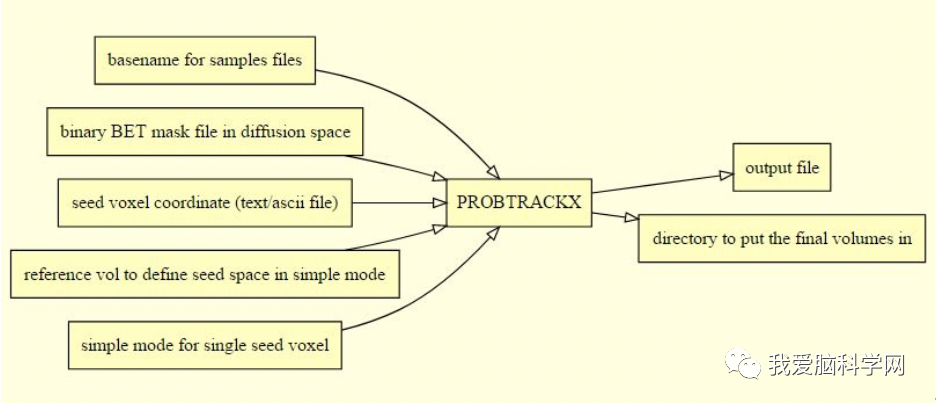
2.在非弥散张量空间进行体素追踪
终端打开Fdt,仍然进行single voxel模式,选择seed space is not diffusion(seed reference image——struct_brain, seedto diff transform 在xfms里的str2diff.mat)
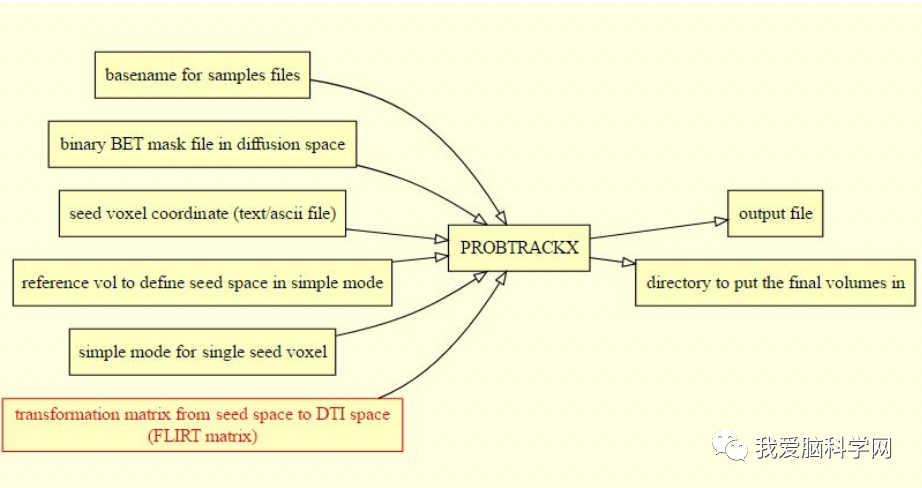
3.在MNI152空间里基于体素追踪
在标准空间里定义种子点。线性或非线性转换到标准空间都行。在线性配准里,使用standard2diff.amt作为seed to diff transfer,使用standard作为reference image;在非线性配准里,首先勾选nonlinear ,然后使用standard2diff_warp和diff2standard_warp作为transfer图像,standard作为reference 图像。
Connectivity-based segmentation :
find_the_biggest THAL2CTX_right/seeds_to_*biggest_segmentation,对产生的种子点进行丘脑的硬分割,并且将分割叠加在标准脑上。
Crossing fibre tractography:使用vecreg程序去转换dyads文件并配准到xfms/diff2standard_warp。vecreg -i dyads1 -odyads1_to_standard -r standard -w xfms/diff2standard_warp
Anatomical priors in tractography:将seed mode改为single mask,里面输入标准空间下的mask图像,在options里面number of samples改小点可以加快追踪成像的速度。使用waypoint mask和termination mask。
Bedpostx model spexification
Using surfaces to constrain tractography:
echo "97 96 125" > svox.txtprobtrackx2 \ #echo "97 96125" > svox.txt创建一个名为svox.txt包含字符串97 96 125的文本文件。
--samples=../subj1_model2_3fibres.bedpostX/merged\ --mask=../subj1_model2_3fibres.bedpostX/nodif_brain_mask.nii.gz\
-x svox.txt \
--xfm=freesurfer2fa.mat--seedref=orig.nii.gz \
--loopcheck --simple --forcedir \
--opd -V 1 --dir=Tracts --out=no_surf
echo lh.cortex.gii > stop.txt
echo rh.cortex.gii >> stop.txt
probtrackx2 \
--samples=../subj1_model2_3fibres.bedpostX/merged \
--mask=../subj1_model2_3fibres.bedpostX/nodif_brain_mask.nii.gz \
-xsvox.txt \
--xfm=freesurfer2fa.mat --seedref=orig.nii.gz \
--loopcheck --simple --forcedir --opd -V 1 \
--dir=Tracts --out=surf --meshspace=freesurfer --stop=stop.txt
fsleyes -ds world \
orig.nii.gz rh.cort.nii.gz -cm green \
lh.cort.nii.gz -cm copper \
Tracts/no_surf_97_96_125 -cm blue-lightblue -or 10 1000 \
Tracts/surf_97_96_125 -cm red-yellow -or 10 1000
第九章 ,结构像预处理,敬请期待下期推送~
以上内容来源原创投稿,文章仅限学习使用,未经原作者授权,请勿转载,不用于商业行为,若有侵权及疑问,请后台留言!
52brain,Connect Young Brains.
推荐阅读:
原创 脑功能MRI数据处理手册系列教程(一)
原创 脑功能MRI数据处理手册系列教程(二)
脑功能MRI数据处理手册系列教程(三)更新脑功能MRI数据处理手册系列教程(四)




















 2237
2237











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








